Wissenschaftliche Illustrationen
Alternativbild:
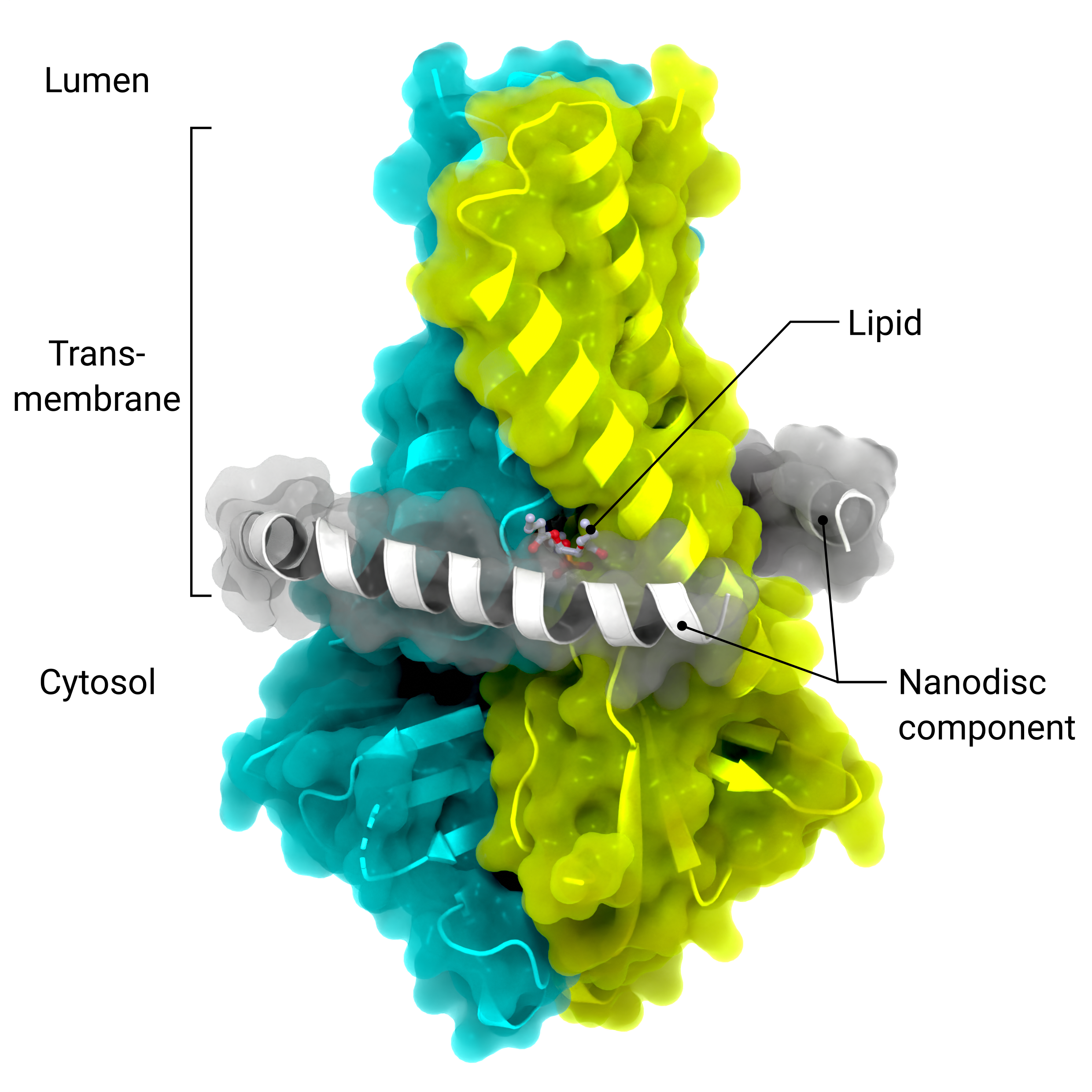
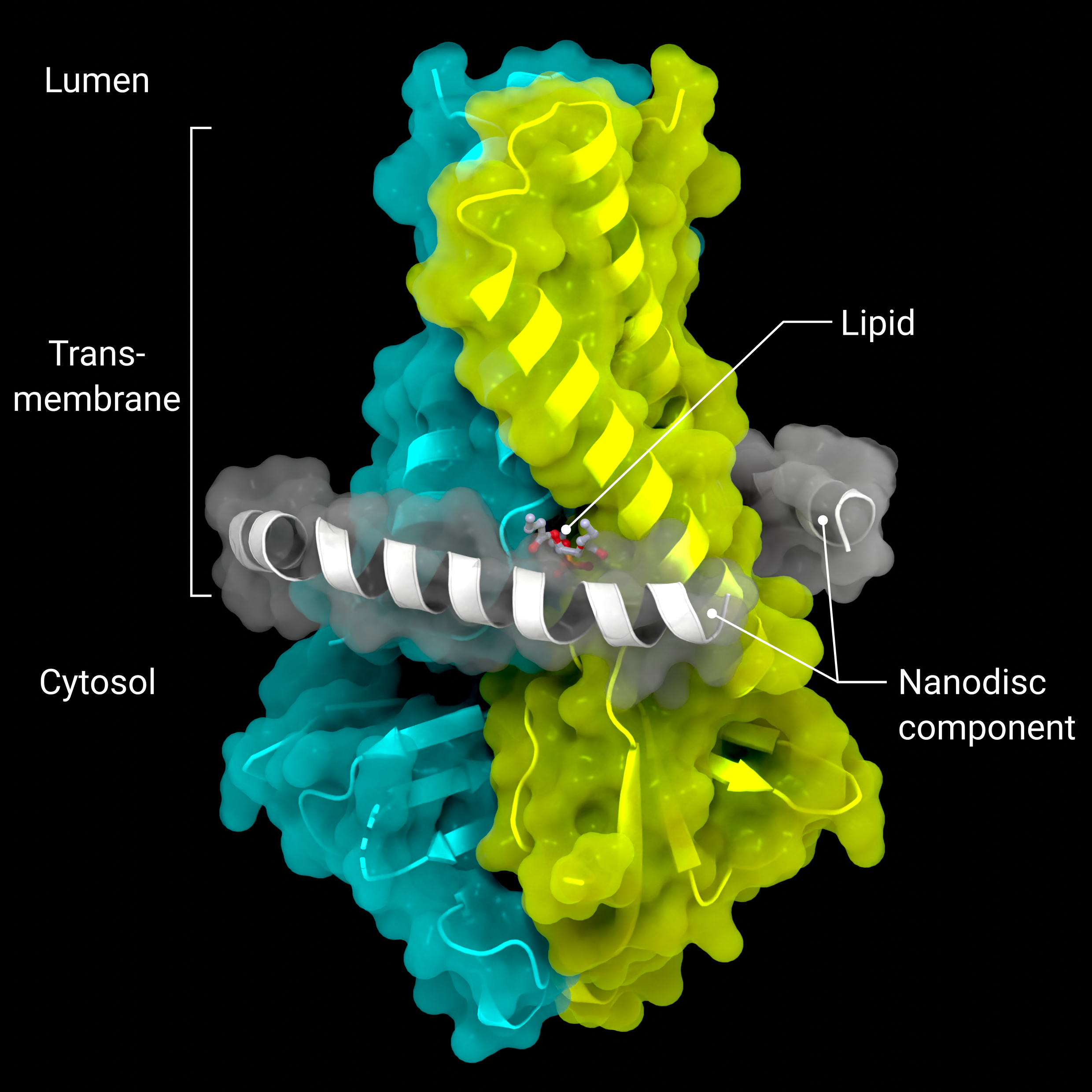
Accessory Protein ORF3a
Die Kryo-EM-Struktur des Viriporins ORF3a (PDB: 7KJR) in einer Lipid-Nanodisk (geordnete Nanodisk-Komponenten in grau)
Urheber: Coronavirus Structural Task Force - Lisa Schmidt, David C. Briggs
Lizenz: cc-by-sa
Alternativbild:
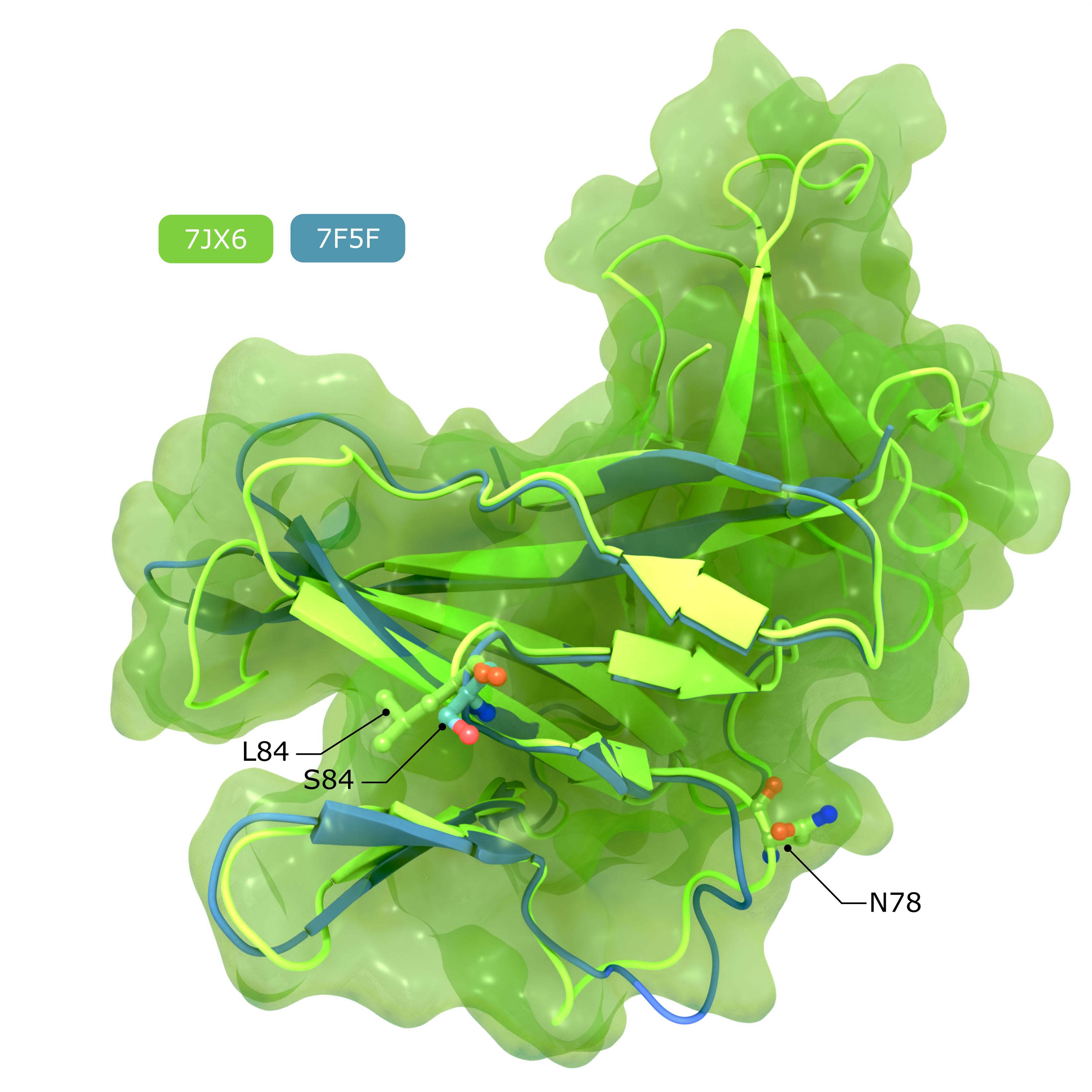
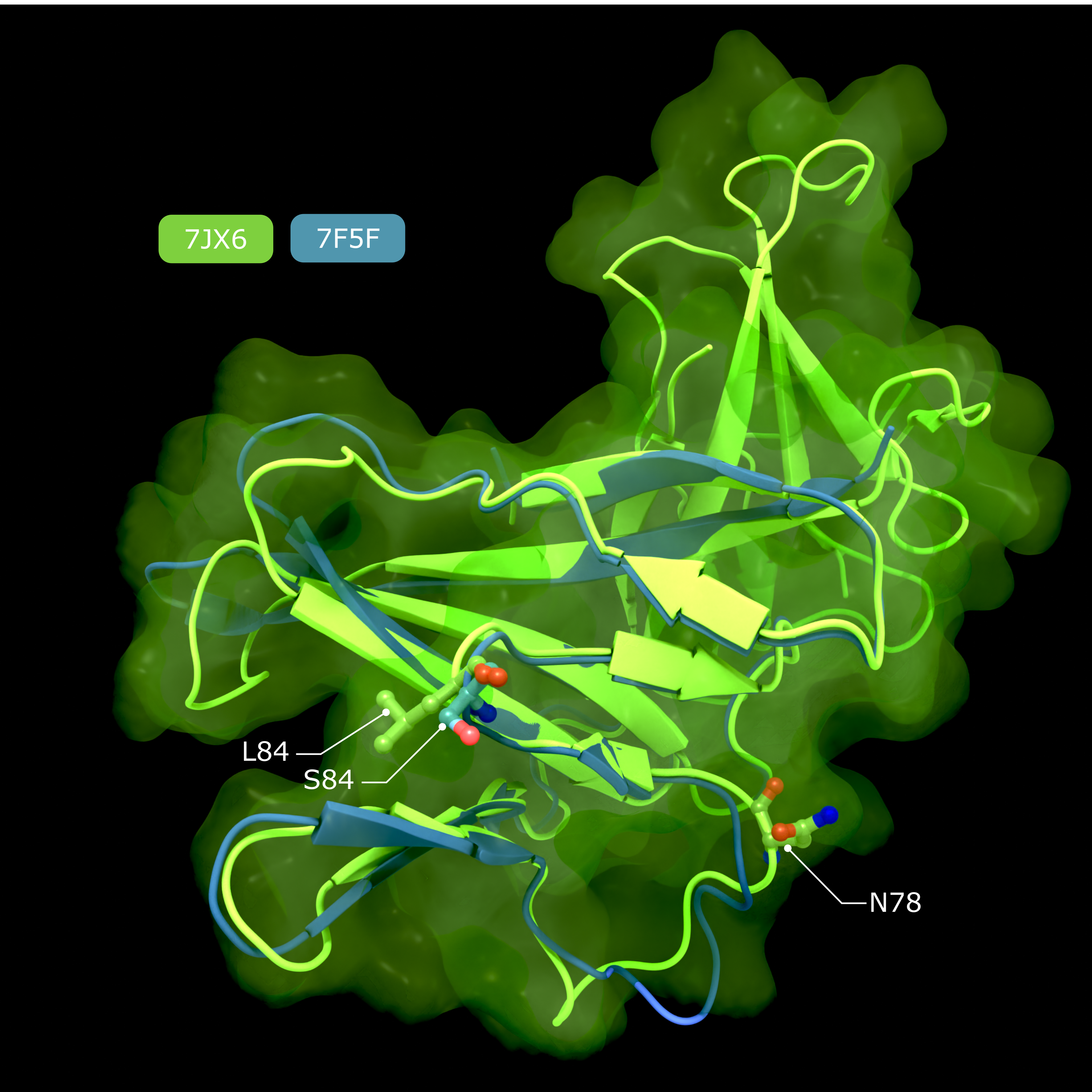
Accessory Protein ORF8
Vergleich der Strukturen von SARS-CoV-2 ORF8, die die mutmaßliche IL17RA-Bindungsfläche zeigen, die den Polymorphismus an Position 84 beherbergt, und das N-gebundene Glykan, das die IL17RA-Bindung im konventionell sezernierten ORF8 hemmt.
Urheber: Coronavirus Structural Task Force - Lisa Schmidt, David C. Briggs
Lizenz: cc-by-sa
Alternativbild:
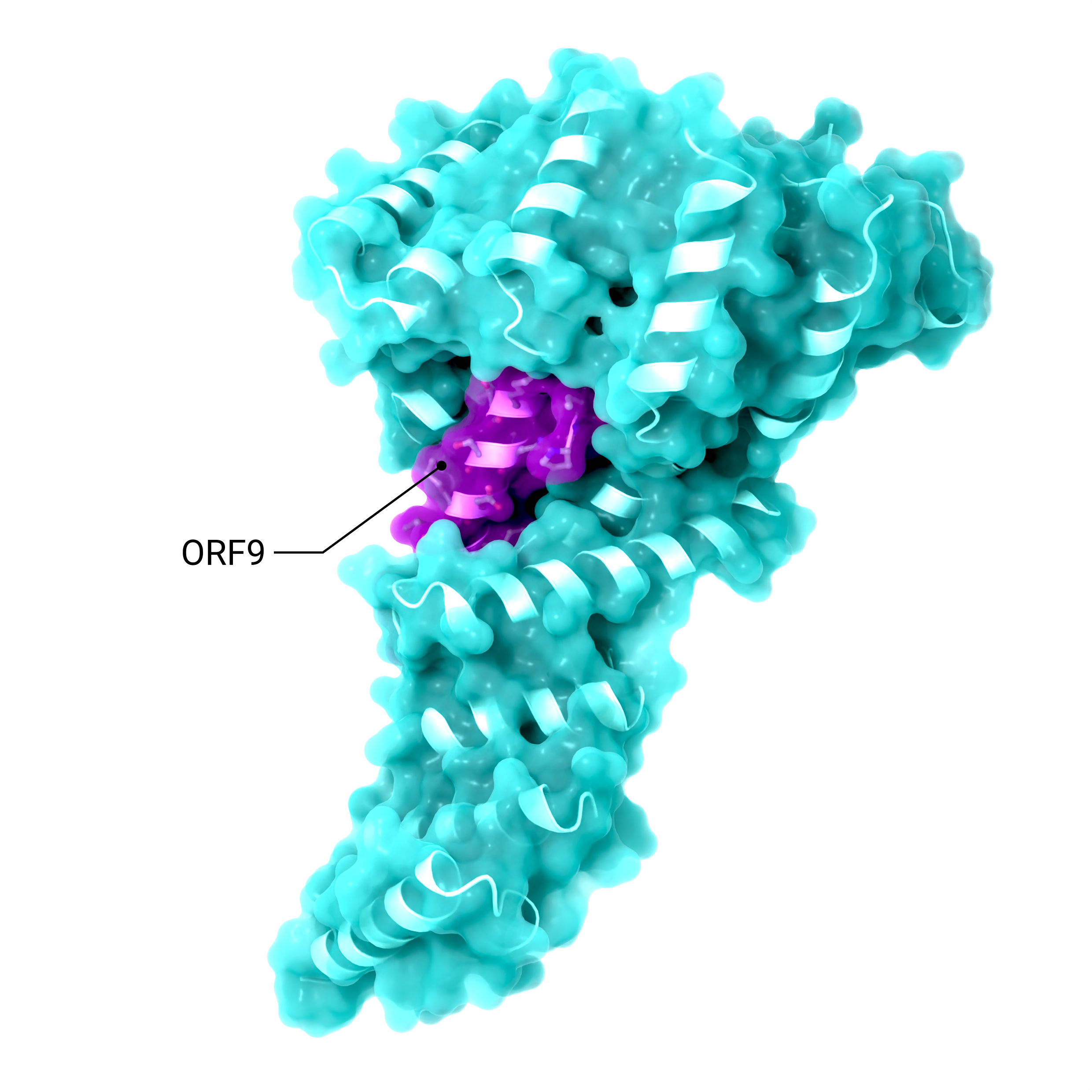
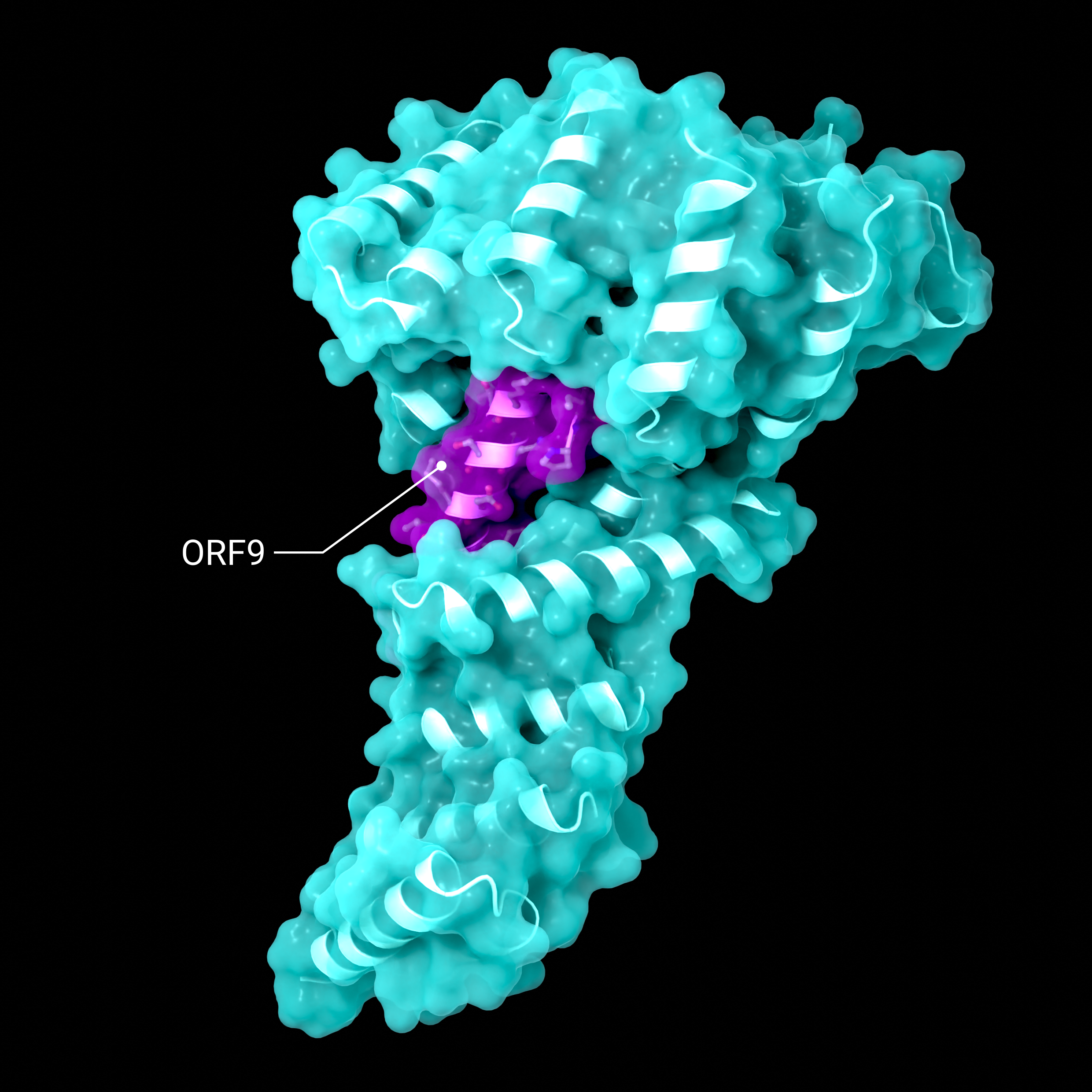
Accessory Protein ORF9
Struktur, die die Bindung von SARS-CoV-2 ORF9 an das menschliche mitochondriale Transportprotein TOM70 zeigt
Urheber: Coronavirus Structural Task Force - Lisa Schmidt, David C. Briggs
Lizenz: cc-by-sa
Alternativbild:
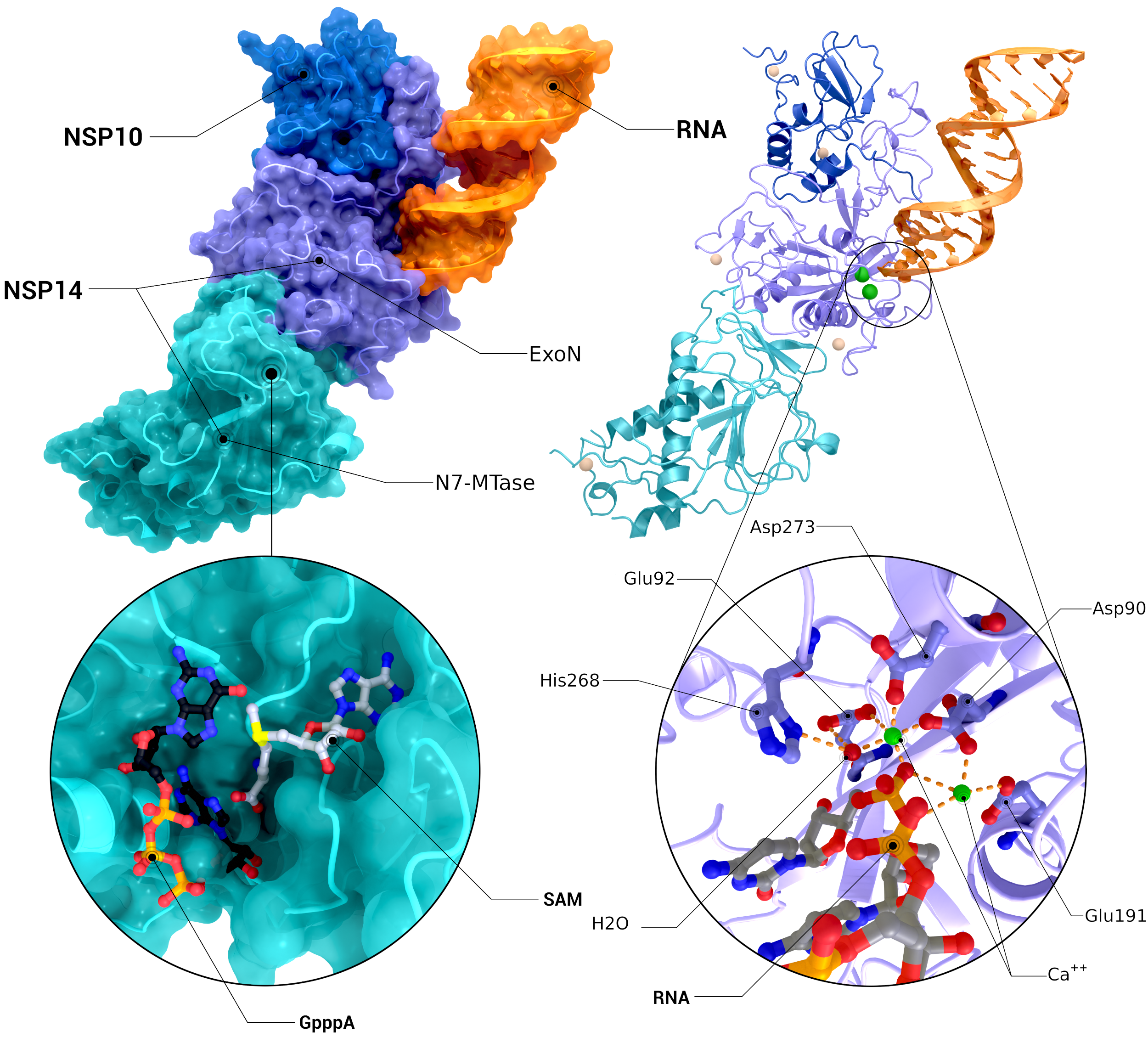
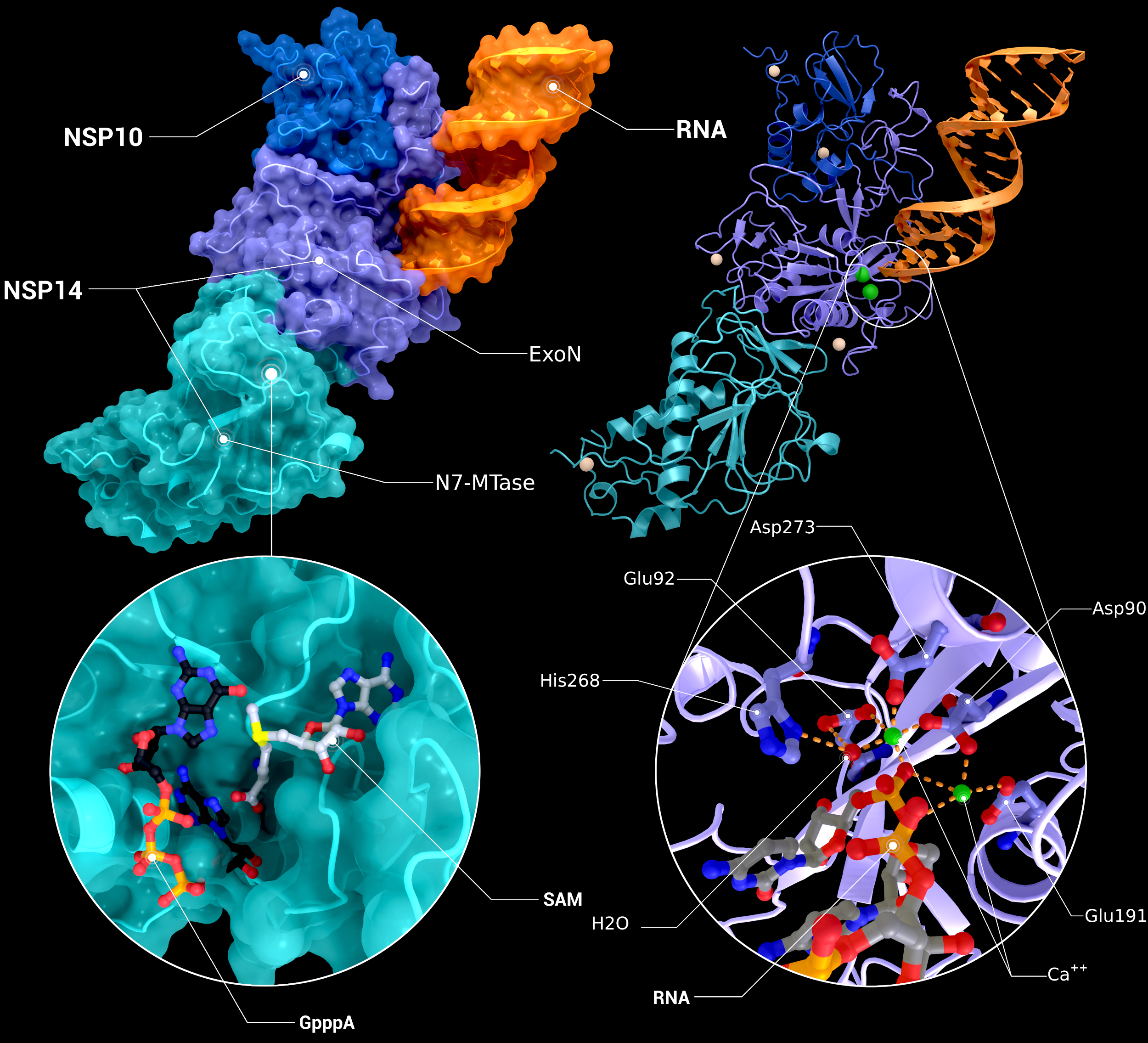
NSP10, NSP14 Heterodimer
Das Heterodimer von nsp10 mit nsp14, das an fehlangepasste RNA in seiner Exoribonuklease-Domäne gebunden ist (PDB ID: 7n0b(nsp10/14/RNA), 5c8s, 5c8t(N7-MTase introspection), 7n0b(ExoN introspection)).
Urheber: Coronavirus Structural Task Force - Protein Imager, Cameron Fyfe
Lizenz: cc-by-sa
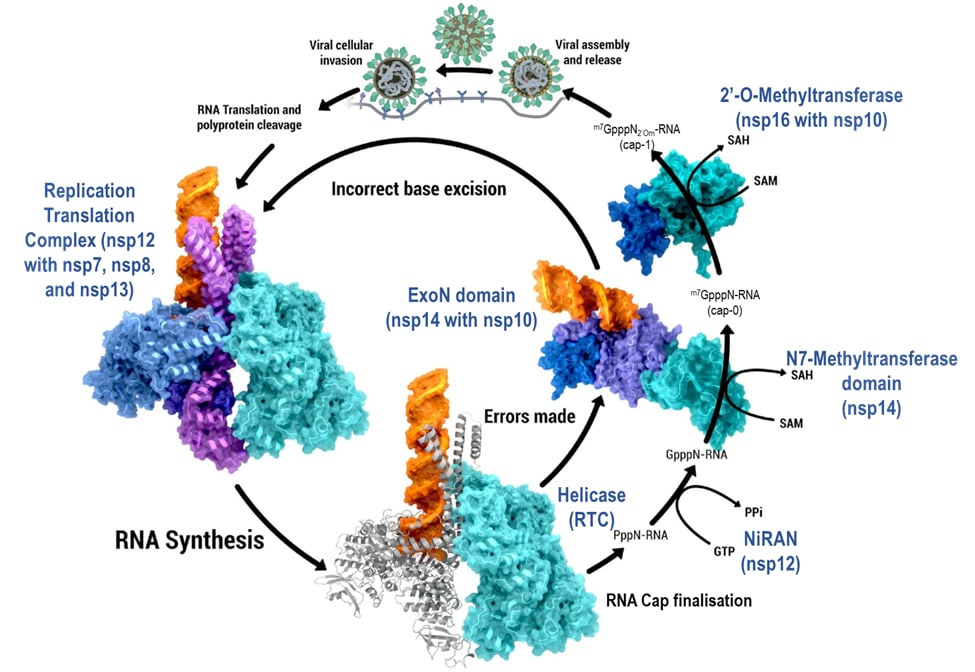
NSP10, NSP14, NSP16 - RNA Synthesis
RNA-Synthese, Fidelitätsregulierung und 5'-RNA-Cap-Finalisierung. Prozesse, Produkte und Schritte innerhalb der Virus-Reproduktion und RNA-Synthese in schwarzer Schrift; Proteine und Proteinkomplexe, die jeden Schritt ausführen, in blauer Schrift (PDB ID: 6xez (RTC), 7n0b (nsp10/14/RNA), 7l6t (nsp10/16)).
Urheber: Coronavirus Structural Task Force - Protein Imager, Cameron Fyfe
Lizenz: cc-by-sa
Alternativbild:
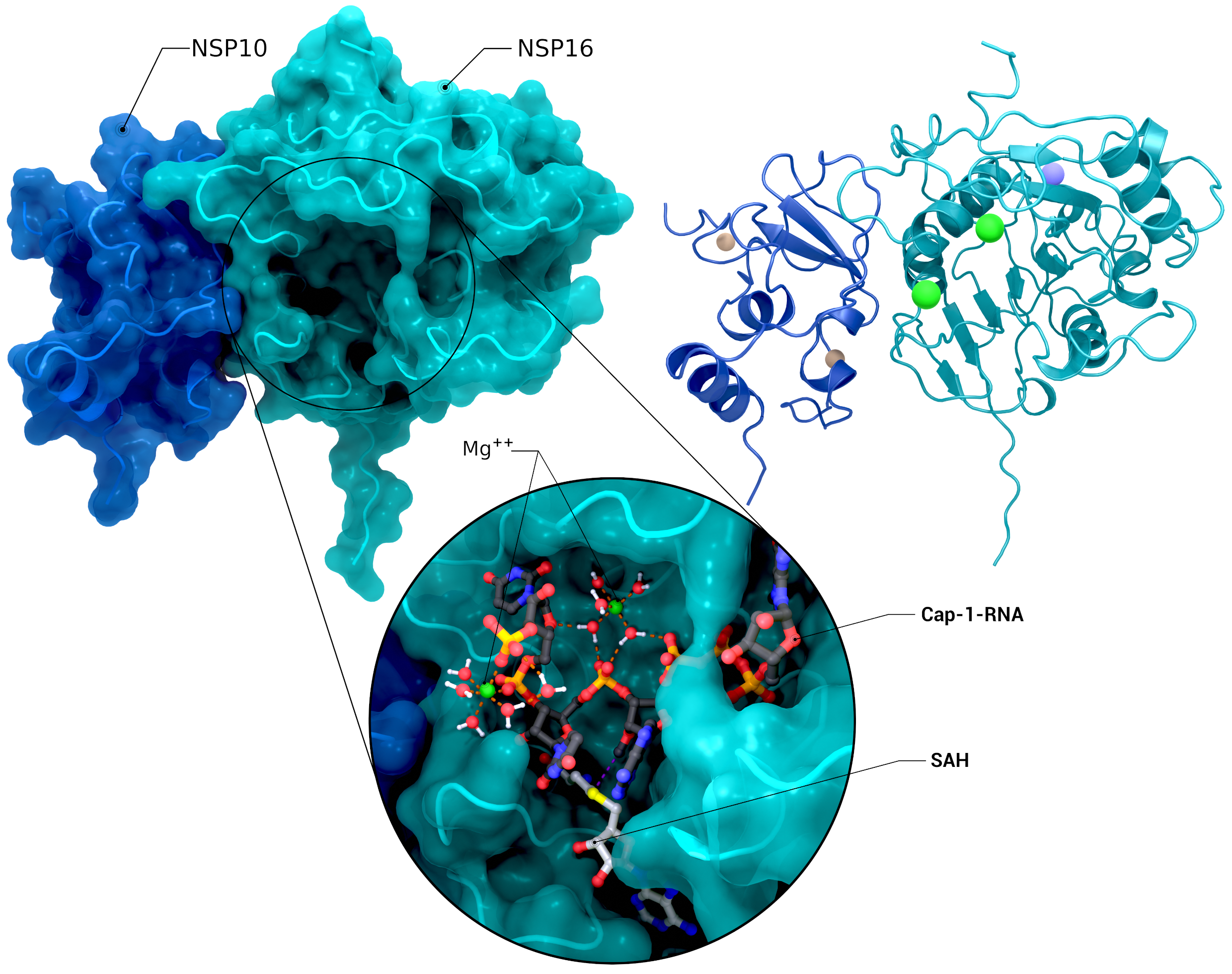
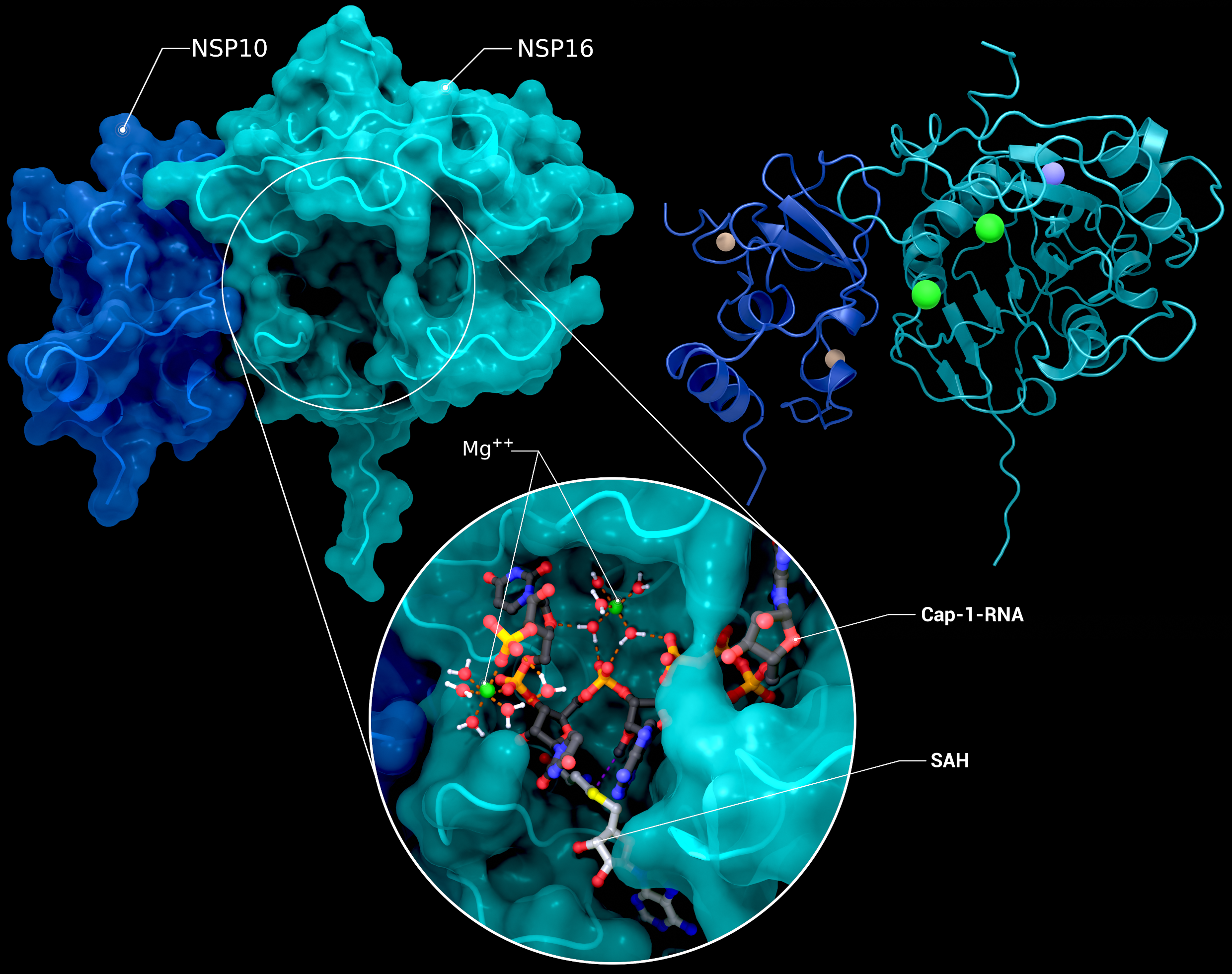
NSP10, NSP16 Heterodimer
Das Heterodimer von nsp10 mit nsp16 in einem Nachreaktionszustand mit Magnesium, seinem Cap-1-RNA-Produkt und S-Adenosyl-L-Homocystein (PDB ID: 7l6t (nsp10/16), 7jyy (nsp16 2′-O-MTase Introspektion)).
Urheber: Coronavirus Structural Task Force - Protein Imager, Cameron Fyfe
Lizenz: cc-by-sa
Alternativbild:
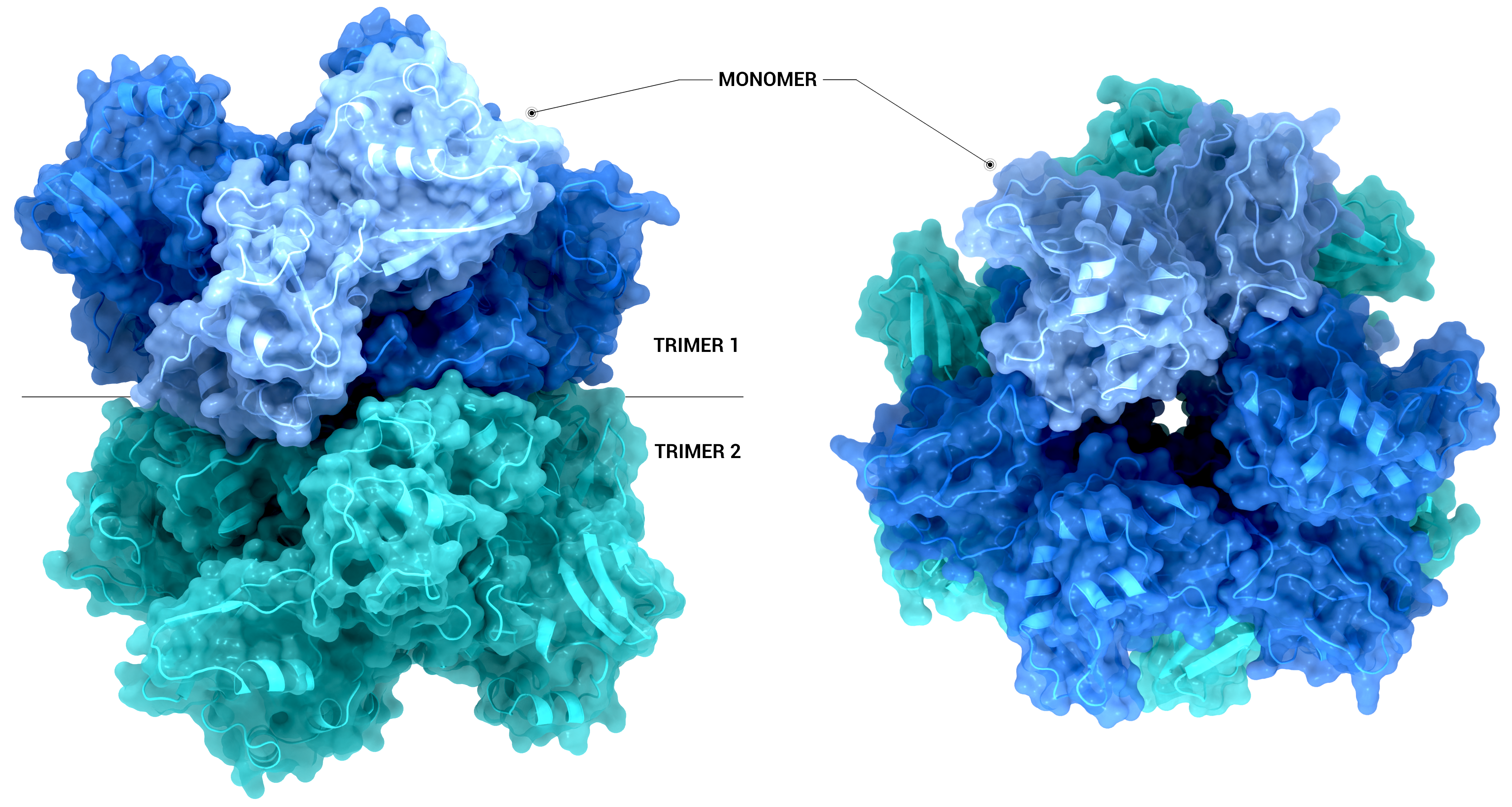
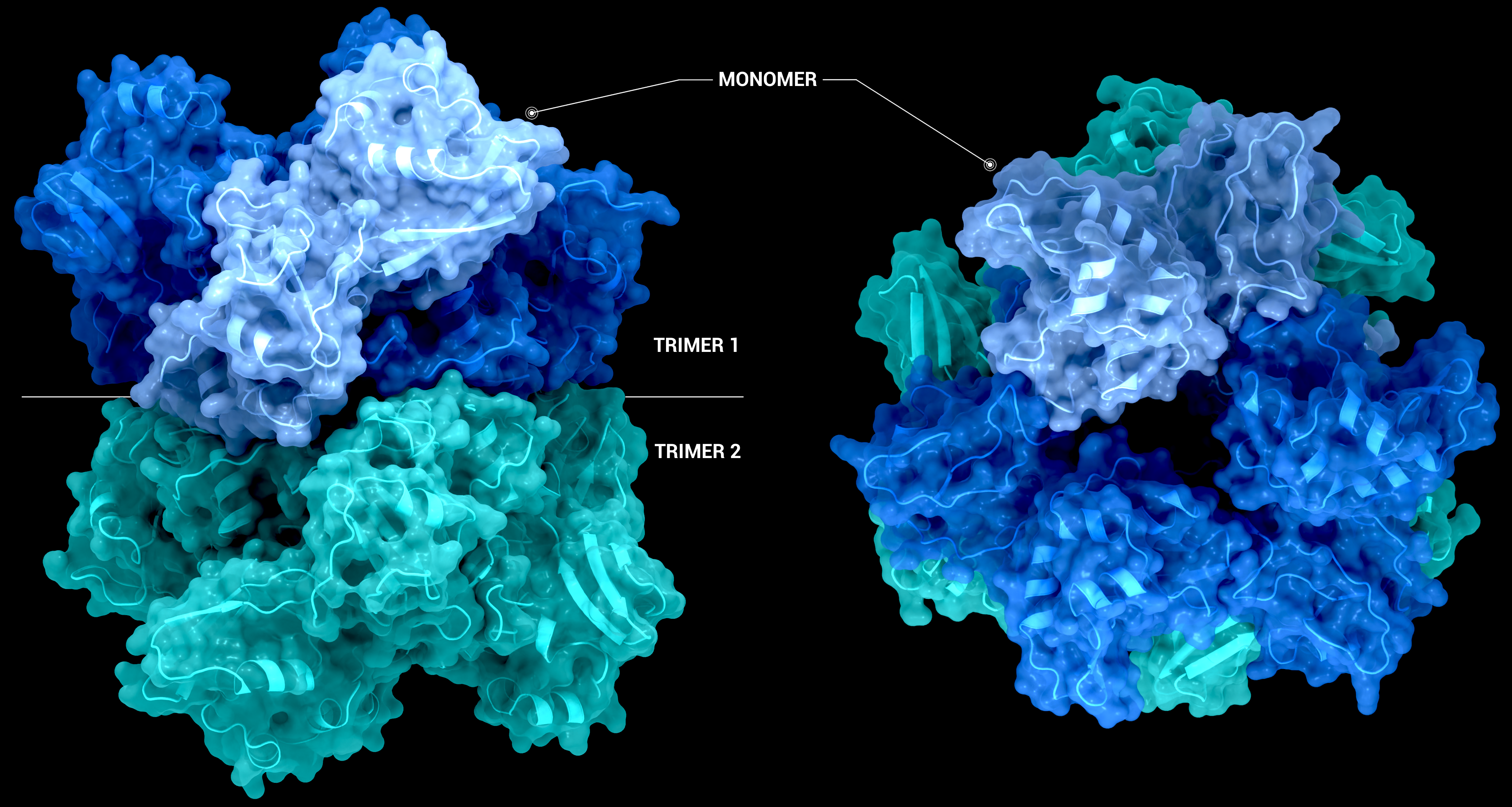
NSP15 Endoribonuclease NendoU - Hexamer
Die Struktur des nsp15-Hexamers wurde durch kristallografische Symmetrie unter Verwendung des PDB-Eintrags 6X4I erzeugt. Auf der linken Seite ist das nsp15-Hexamer als transparentes Surface und als Cartoon in der Seitenansicht dargestellt. Auf der rechte
Urheber: Coronavirus Structural Task Force - Protein Imager, Sam Horrell
Lizenz: cc-by-sa
Alternativbild:
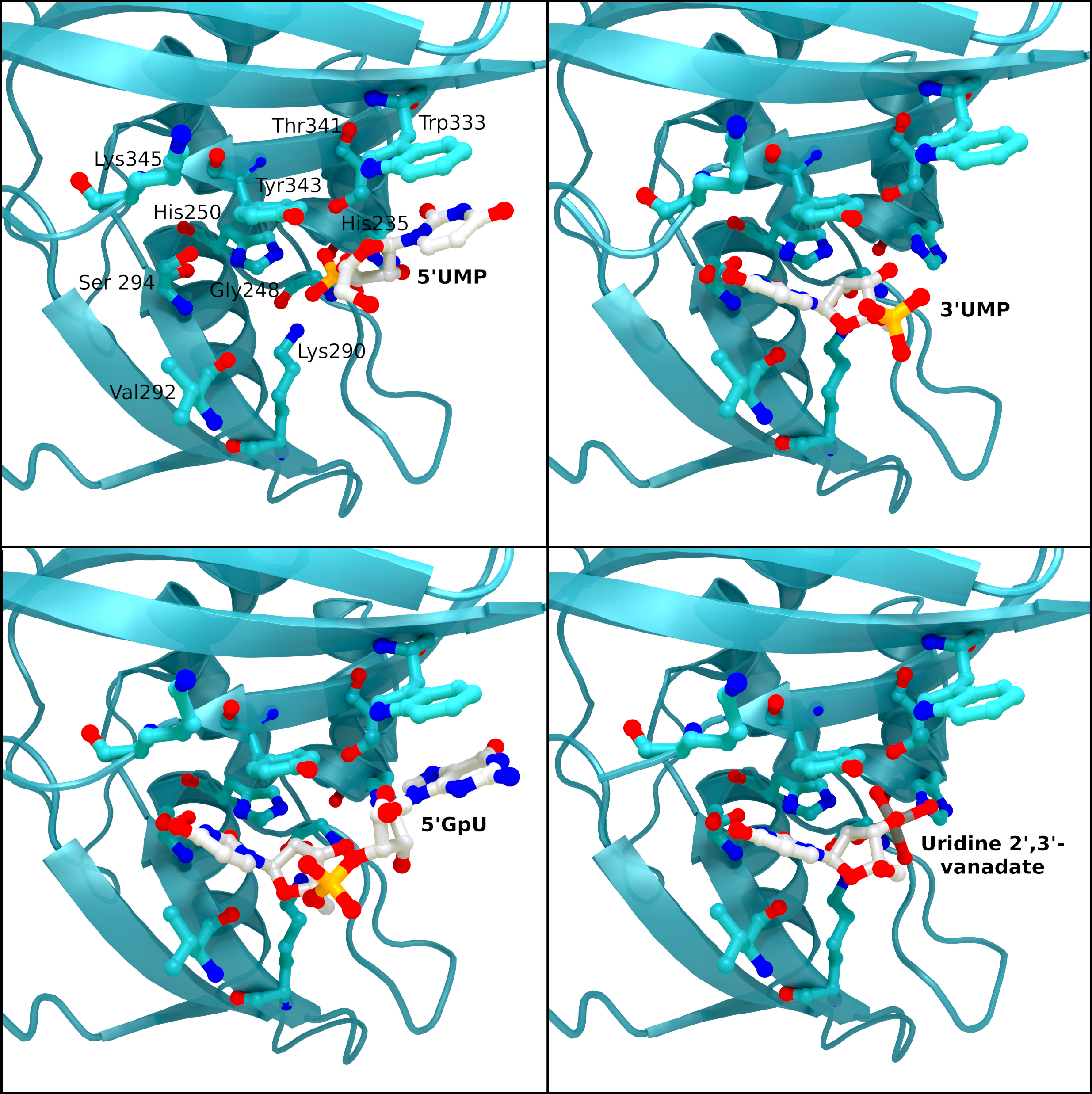
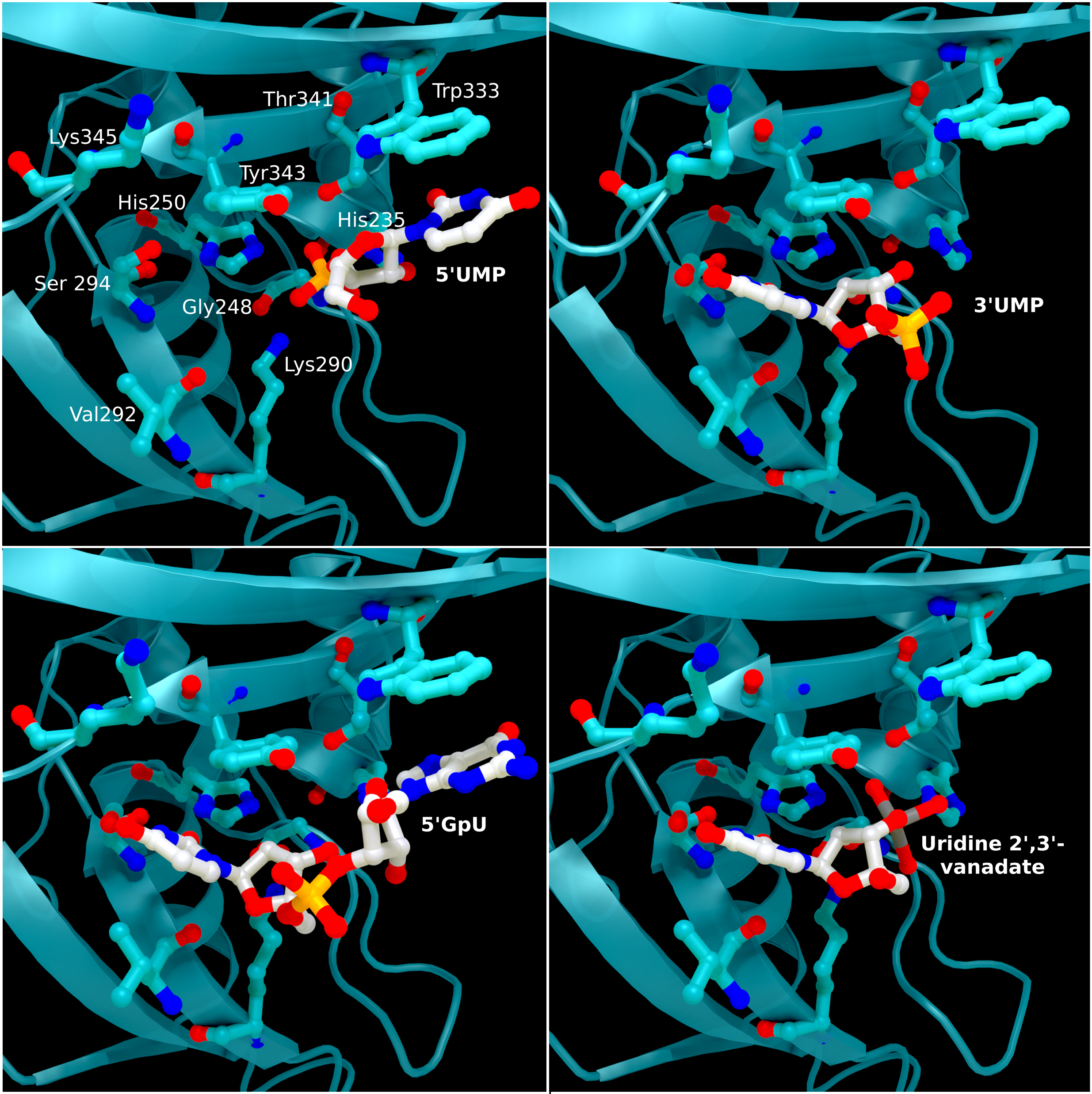
NSP15 Endoribonuclease NendoU - Ligands
SARS-CoV 2 nsp15-Kristallstrukturen des aktiven Zentrums mit gebundenen Reaktionsintermediaten. 5′UMP (PDB-Eintrag: 6WLC) oben links, 3′UMP (PDB-Eintrag: 6X4I) oben rechts, 5′GpU (PDB-Eintrag: 6X1B) unten links und das zyklische Zwischenprodukt Uridin-2′,
Urheber: Coronavirus Structural Task Force - Protein Imager, Sam Horrell
Lizenz: cc-by-sa

Alternativbild:
NSP15 Endoribonuclease NendoU - Monomer
Kristallstruktur des nsp15-Monomers, dargestellt als transparente Surface-Struktur und Cartoon (links) und als Cartoon (rechts), gefärbt nach Domäne unter Verwendung des PDB-Eintrags 6X4I.
Urheber: Coronavirus Structural Task Force - Protein Imager, Sam Horrell
Lizenz: cc-by-sa
Alternativbild:
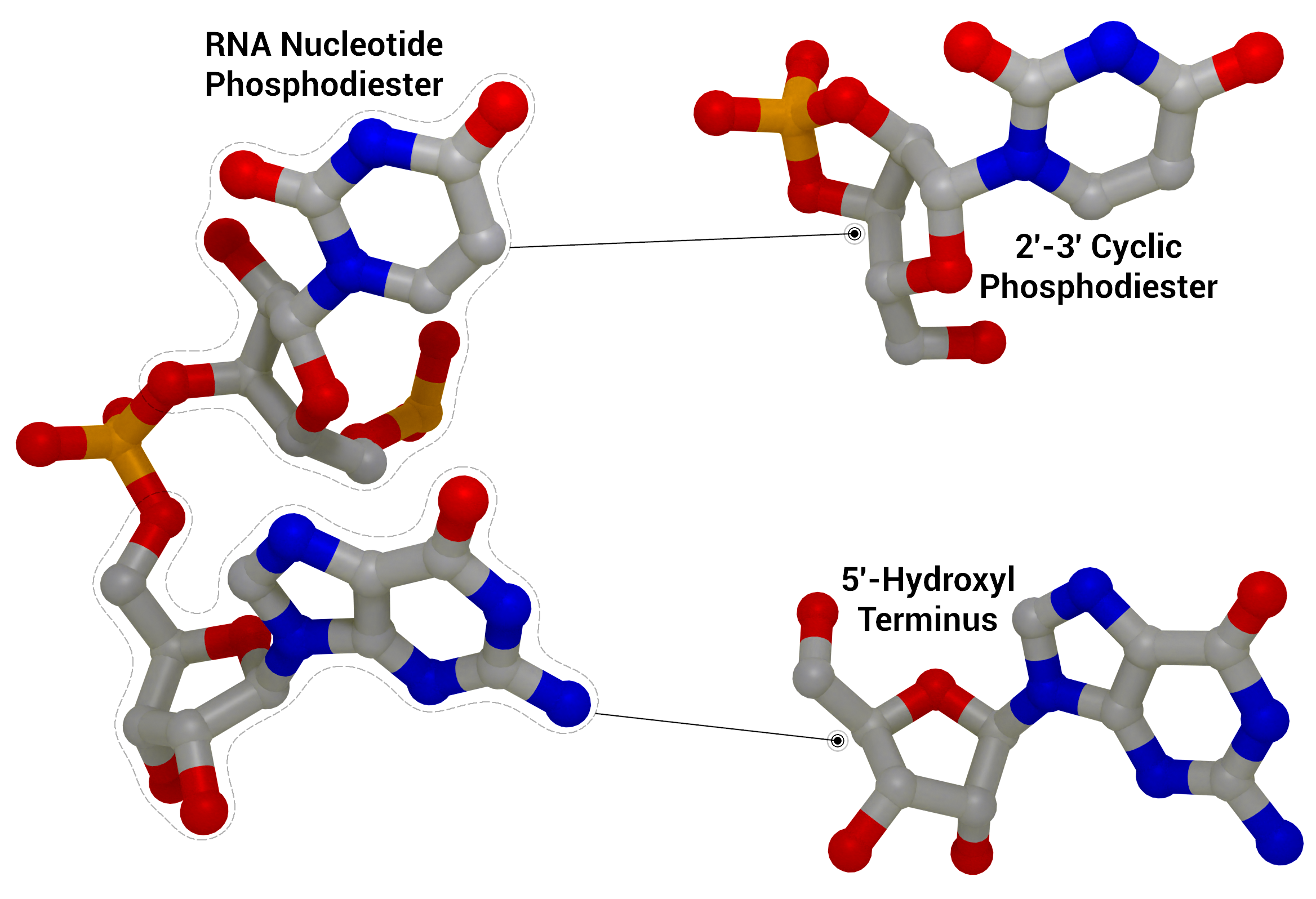
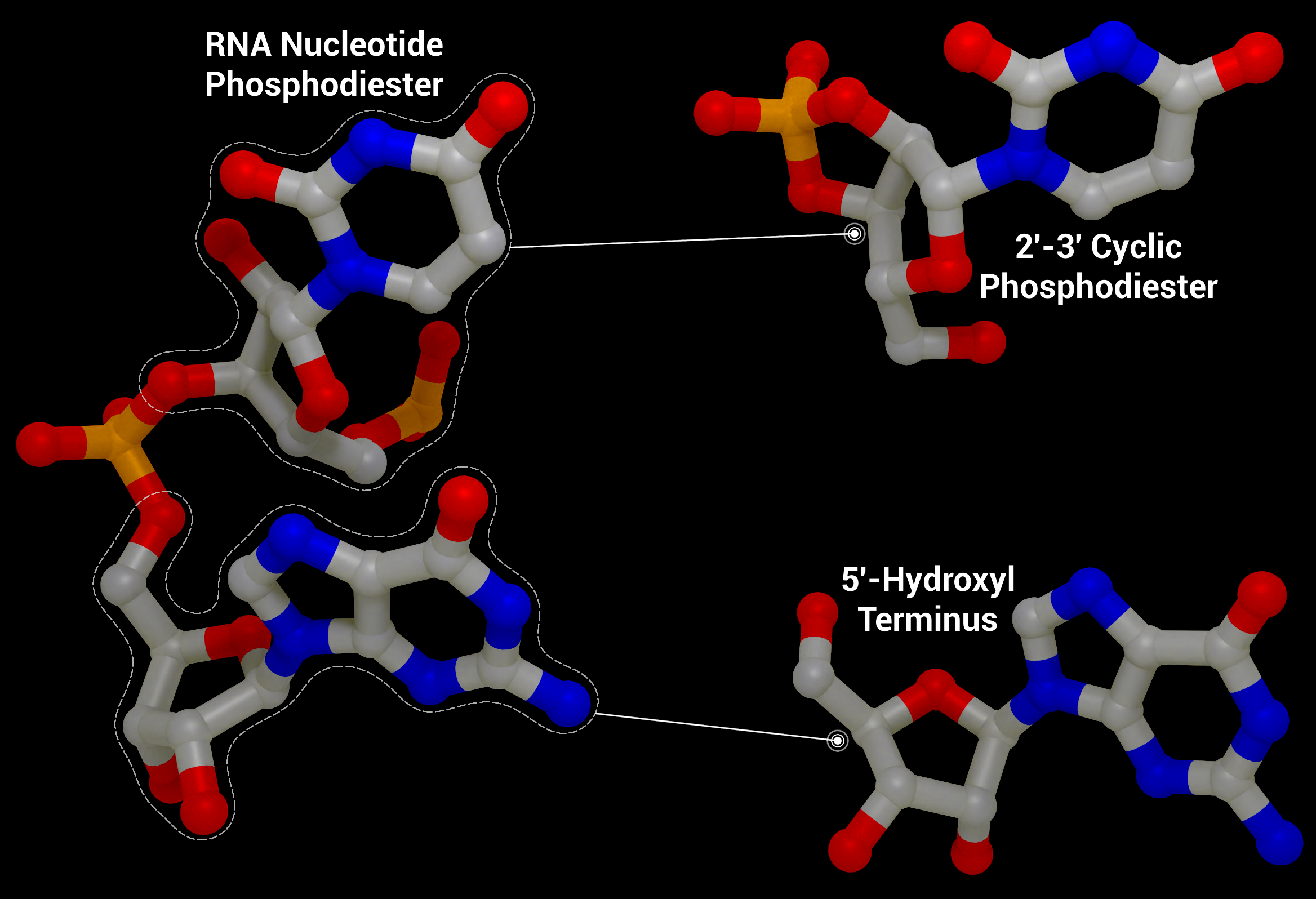
NSP15 Endoribonuclease NendoU - RNA Cleavage
RNA-Spaltung, wie sie von nsp15 durchgeführt wird, um einen zyklischen 2′-3′-Phosphodiester und einen 5′-Hydroxylterminus aus einem RNA-Nukleotid-Phosphodiester aus PDB-Eintrag 1RNA zu erhalten.
Urheber: Coronavirus Structural Task Force - Protein Imager, Sam Horrell
Lizenz: cc-by-sa
Alternativbild:
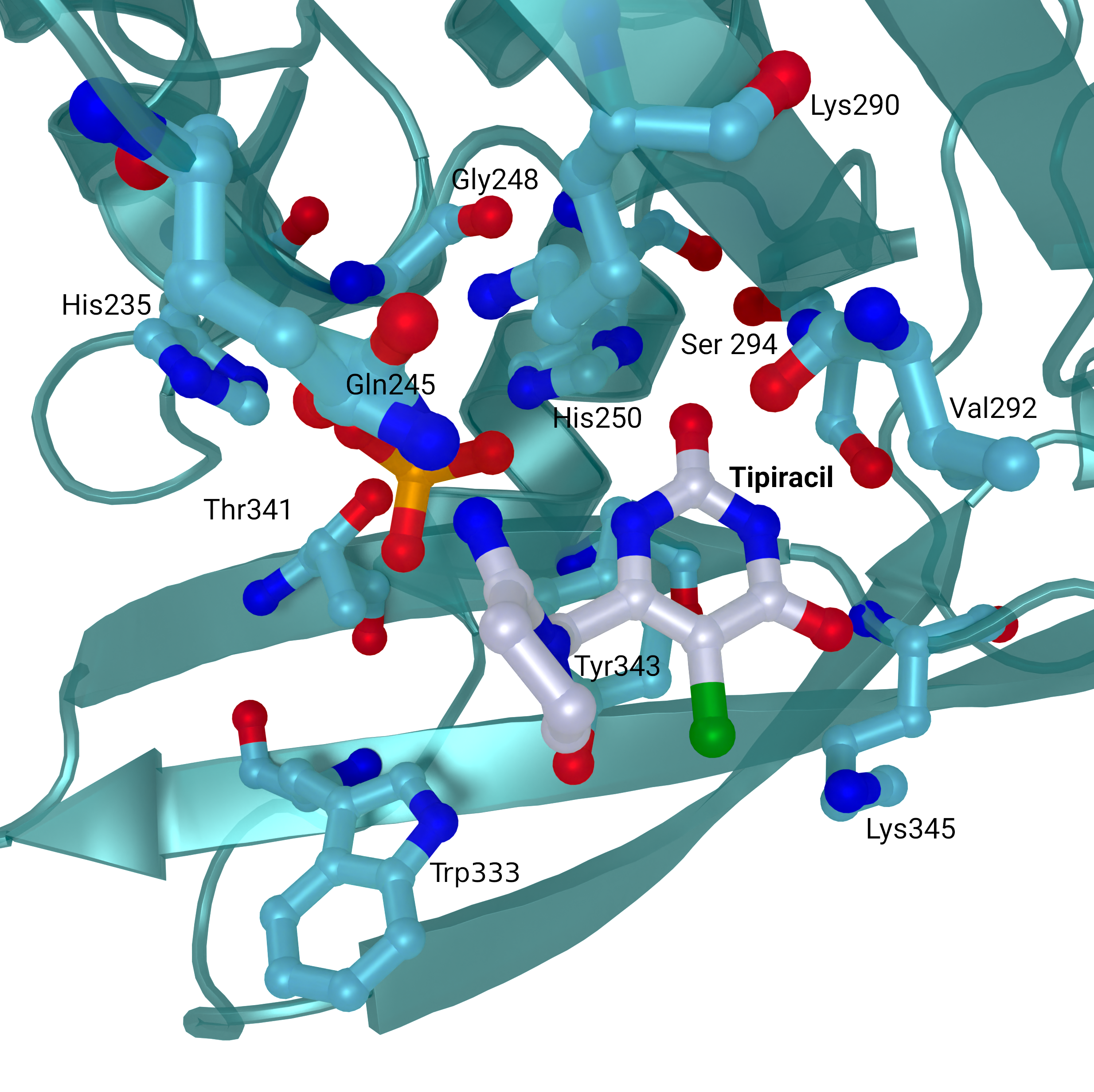
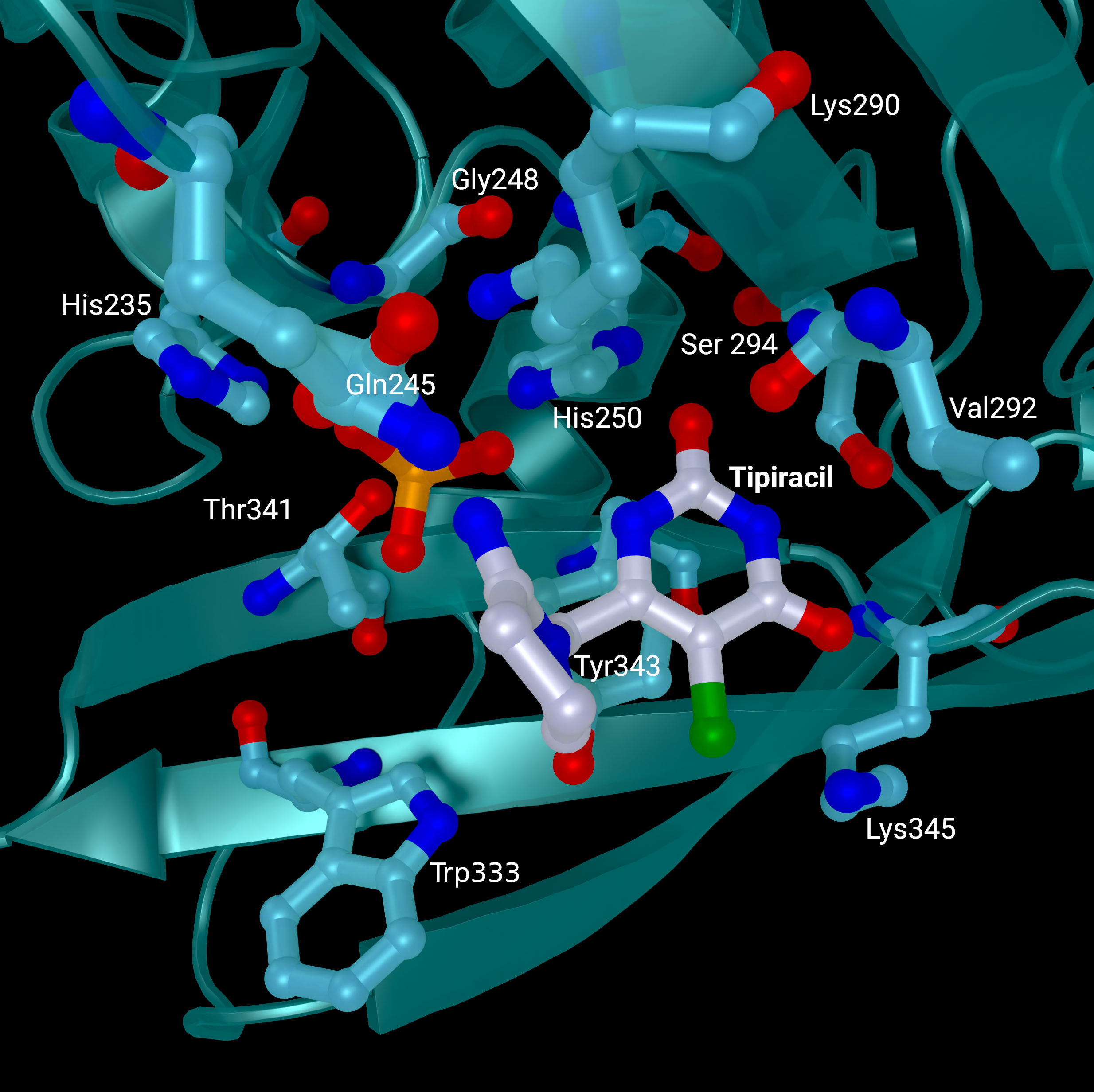
NSP15 Endoribonuclease NendoU - Tipiracil
SARS-CoV 2 nsp15-Kristallstrukturen am aktiven Zentrum mit gebundenem Tipiracil aus PDB-Eintrag 6WXC. Das Protein ist in Grüntönen eingefärbt und als Cartoon dargestellt, wobei die Reste des aktiven Zentrums und das gebundene Tipiracil als Stäbchen darges
Urheber: Coronavirus Structural Task Force - Protein Imager, Sam Horrell
Lizenz: cc-by-sa
Alternativbild:
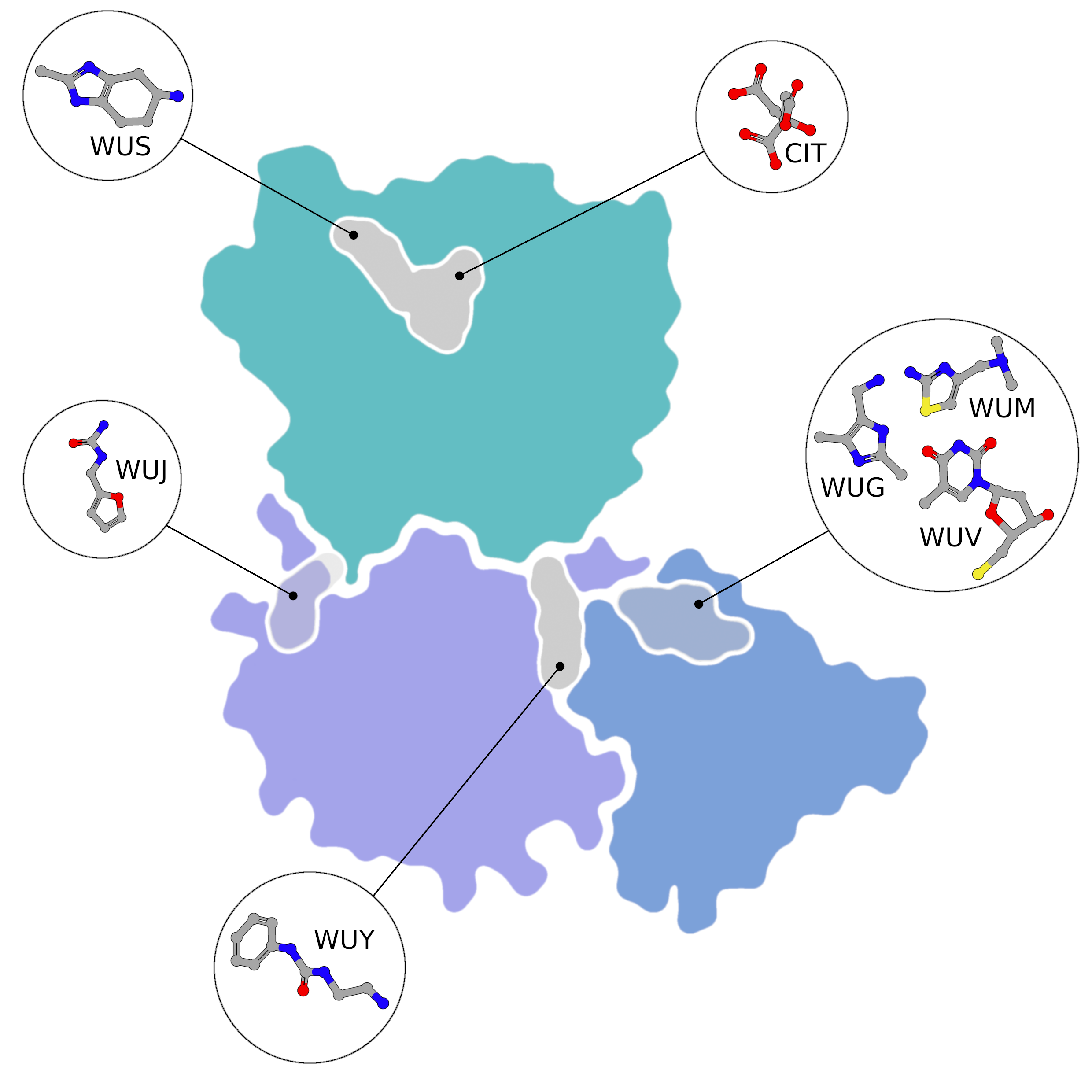
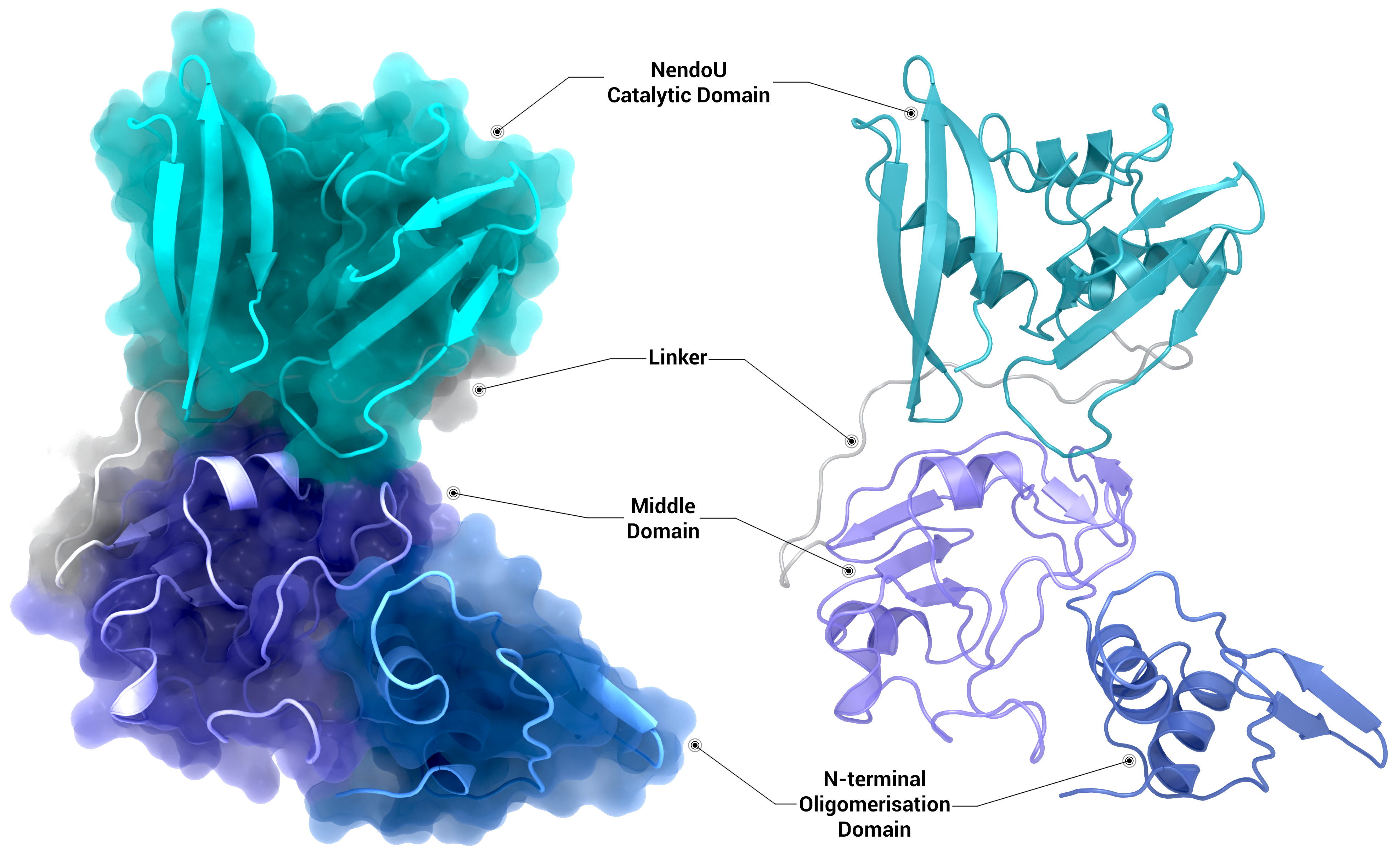
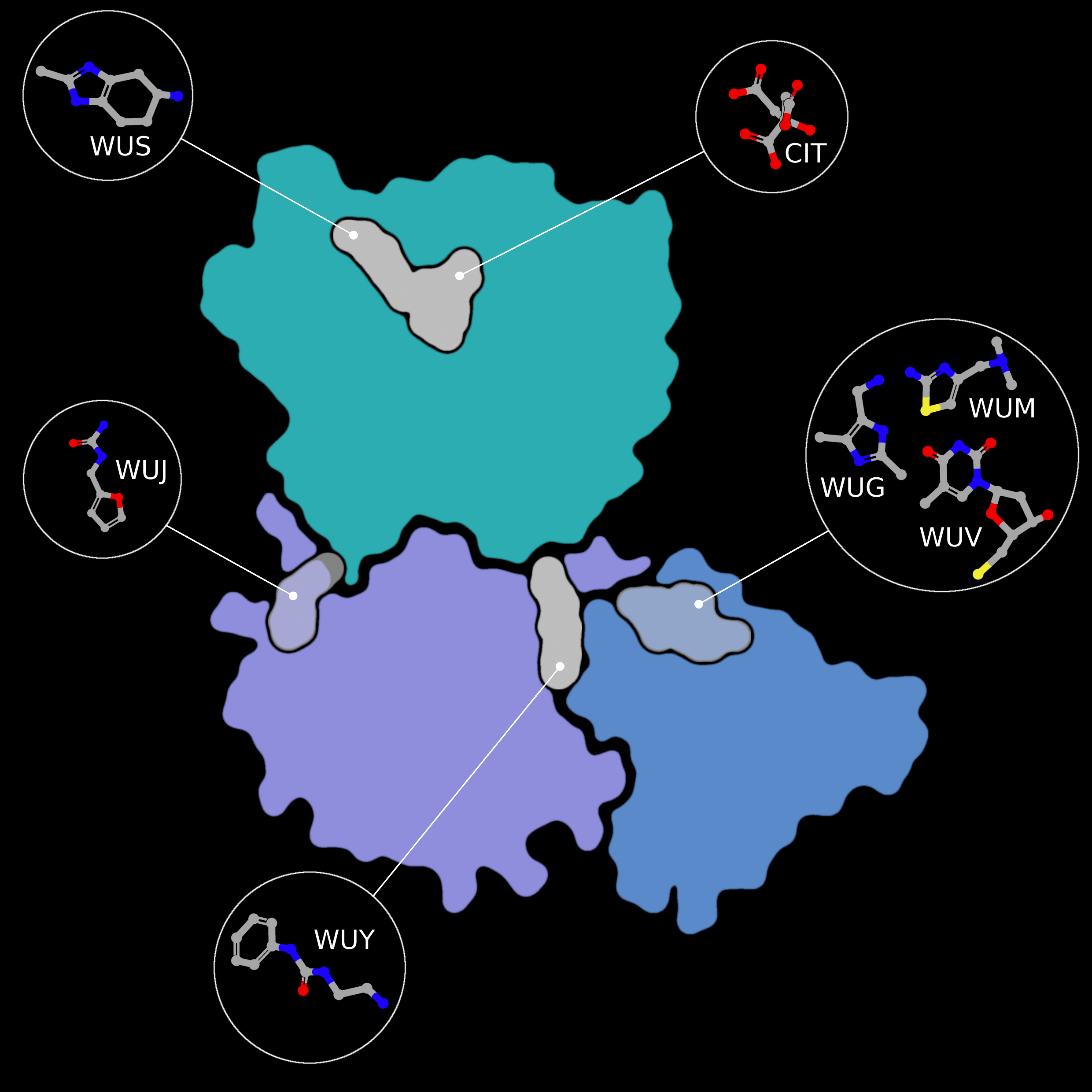
NSP15 Endoribonuclease NendoU - Topology
Screening kleiner Molekülfragmente gegen SARS-CoV-2 nsp15, wobei nsp15 als Flächen dargestellt ist, die nach Domäne gefärbt sind (NendoU in türkis, mittlere Domäne in lila und N-terminale Oligomerisierungsdomäne in blau). Die Fragmentbindung ist als flach
Urheber: Coronavirus Structural Task Force - Protein Imager, Sam Horrell
Lizenz: cc-by-sa
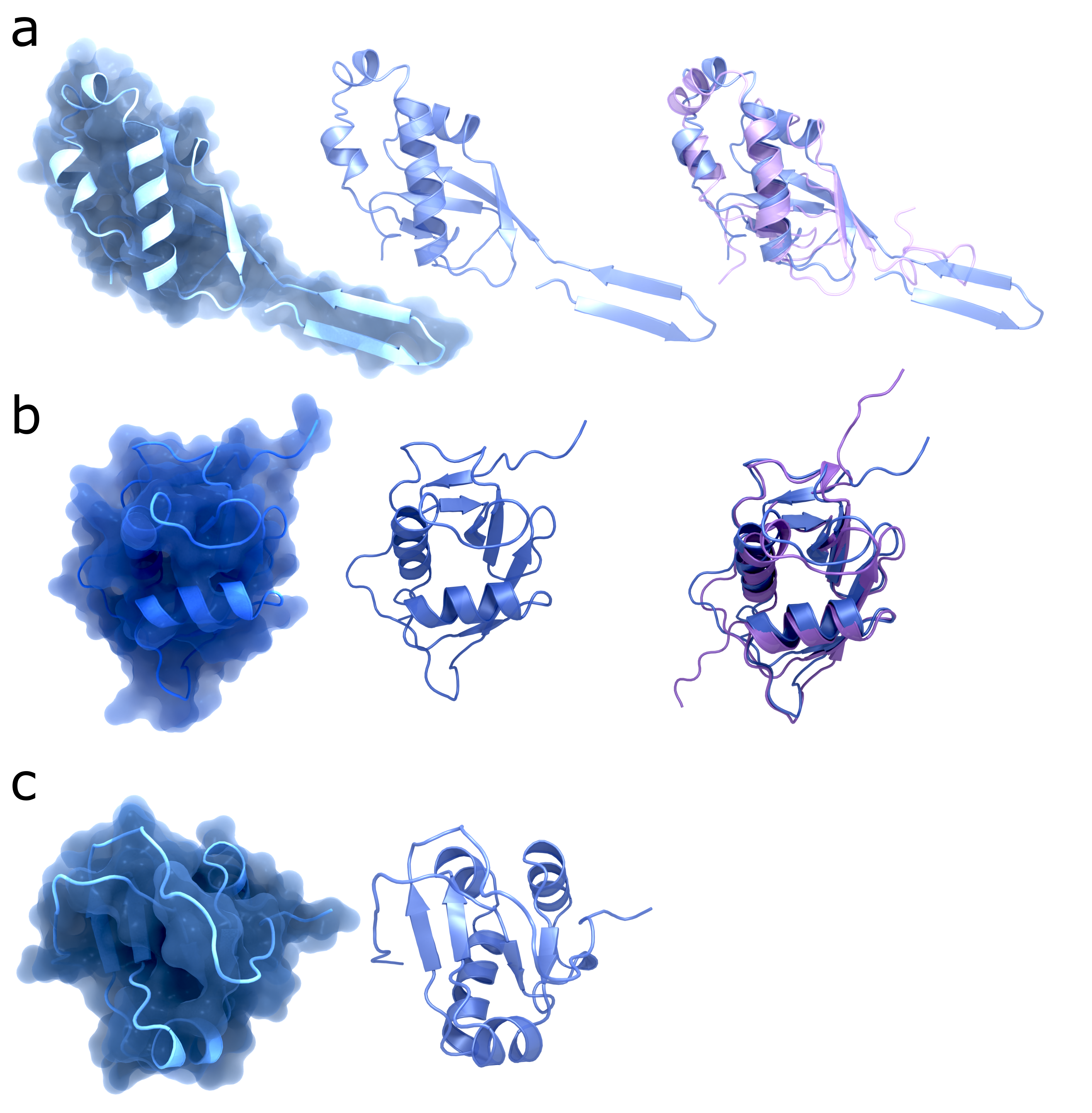
NSP3 Domain Comparison SARS-CoV-2 vs SARS-CoV-1
Strukturen von den NSP3 Domänen Ubl1 (a), NAB (b), und Y3 (c). Die linke Spalte zeigt die Strukturen von SARS-CoV-2 mit Oberfläche (PDB Codes: : 7KAG, 7LGO, 7RQG); die mittlere Spalte zeigt dieselben Strukturen ohne Oberfläche; die rechte Spalte zeigt die
Urheber: Coronavirus Structural Task Force - Protein Imager, Maximilian Edich
Lizenz: cc-by-sa
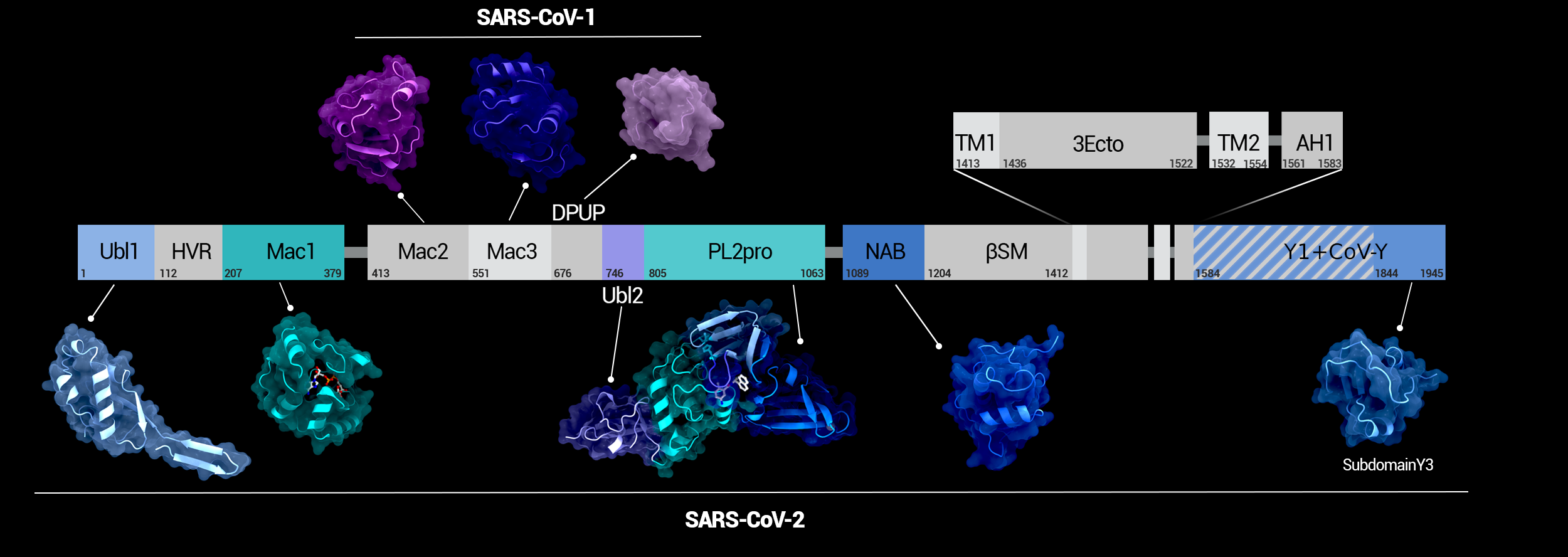
Alternativbild:
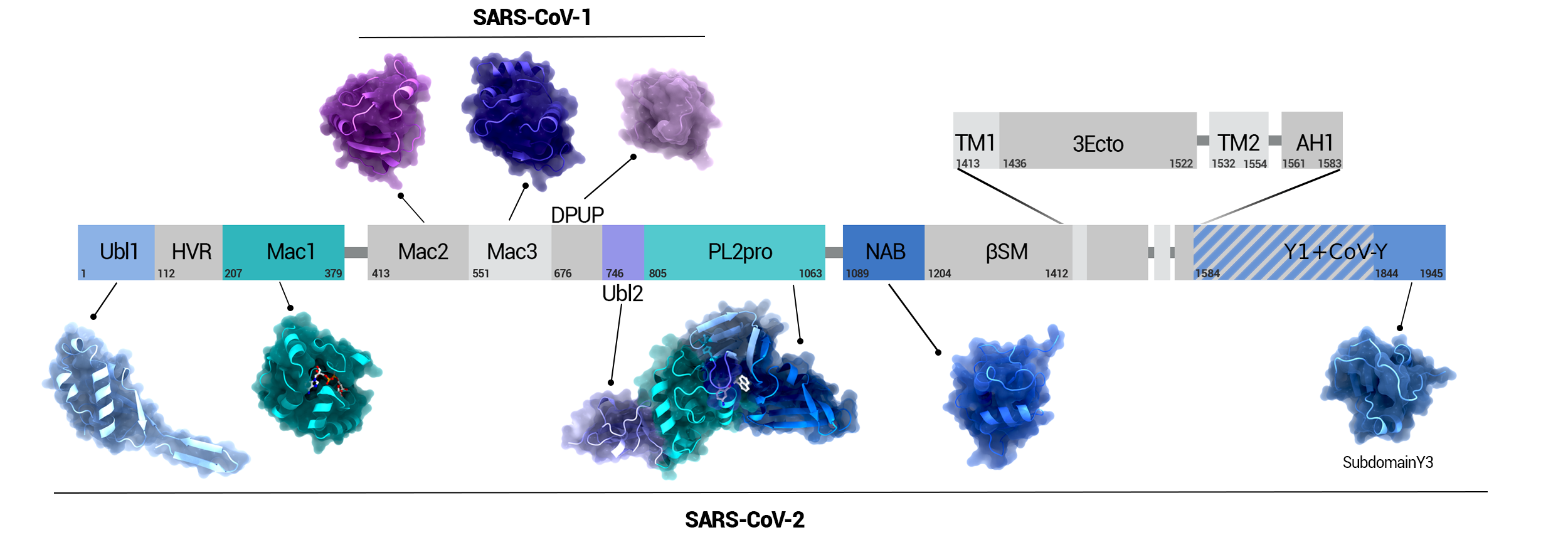
NSP3 Domain Overview
Überblick über die Domänen von NSP3 und deren Position im Protein. Farblich hervorgehoben sind jene Domänen, deren Struktur in SARS-CoV-2 gelöst wurde.
Urheber: Coronavirus Structural Task Force - Protein Imager, Maximilian Edich
Lizenz: cc-by-sa
Alternativbild:
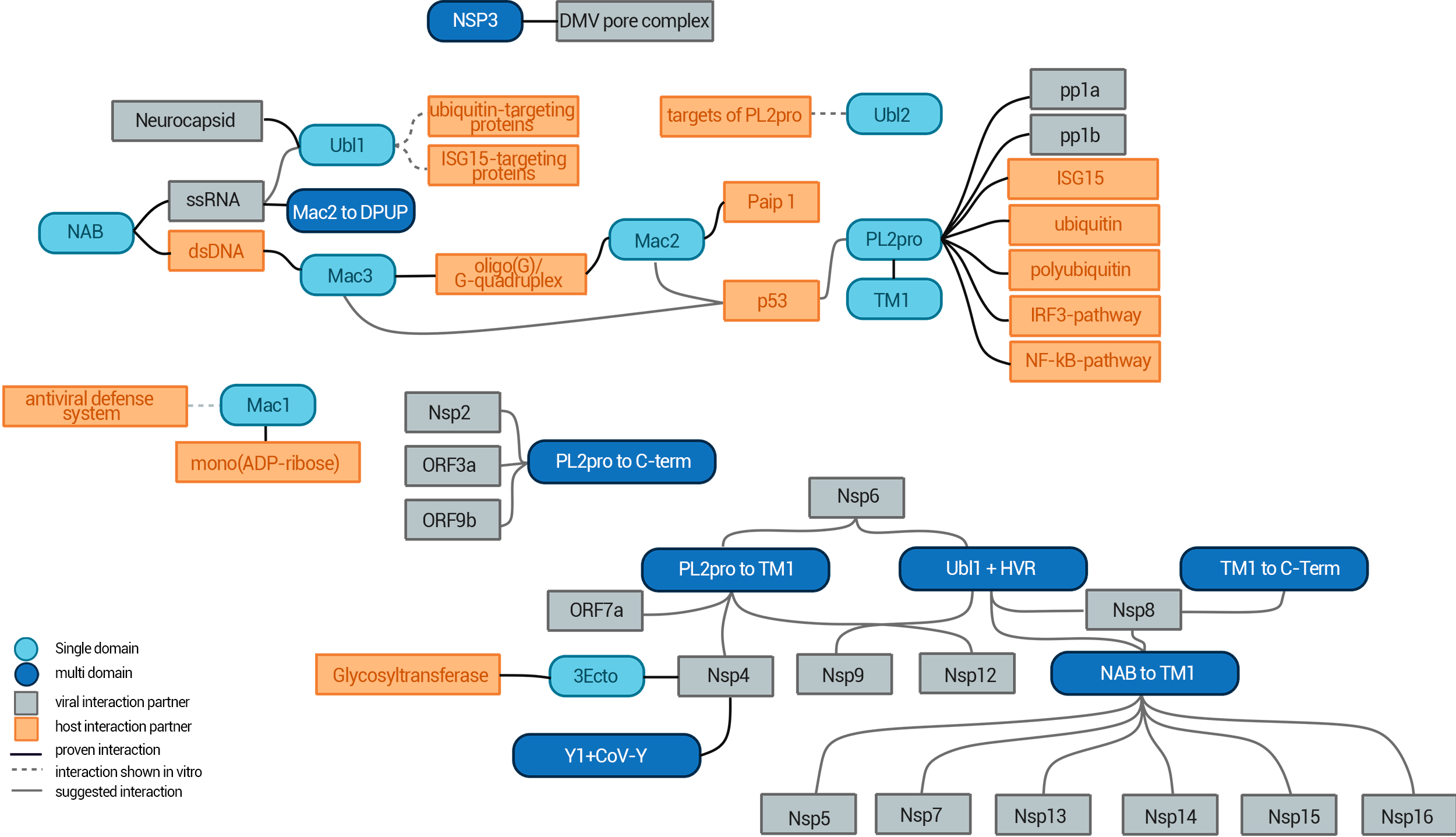
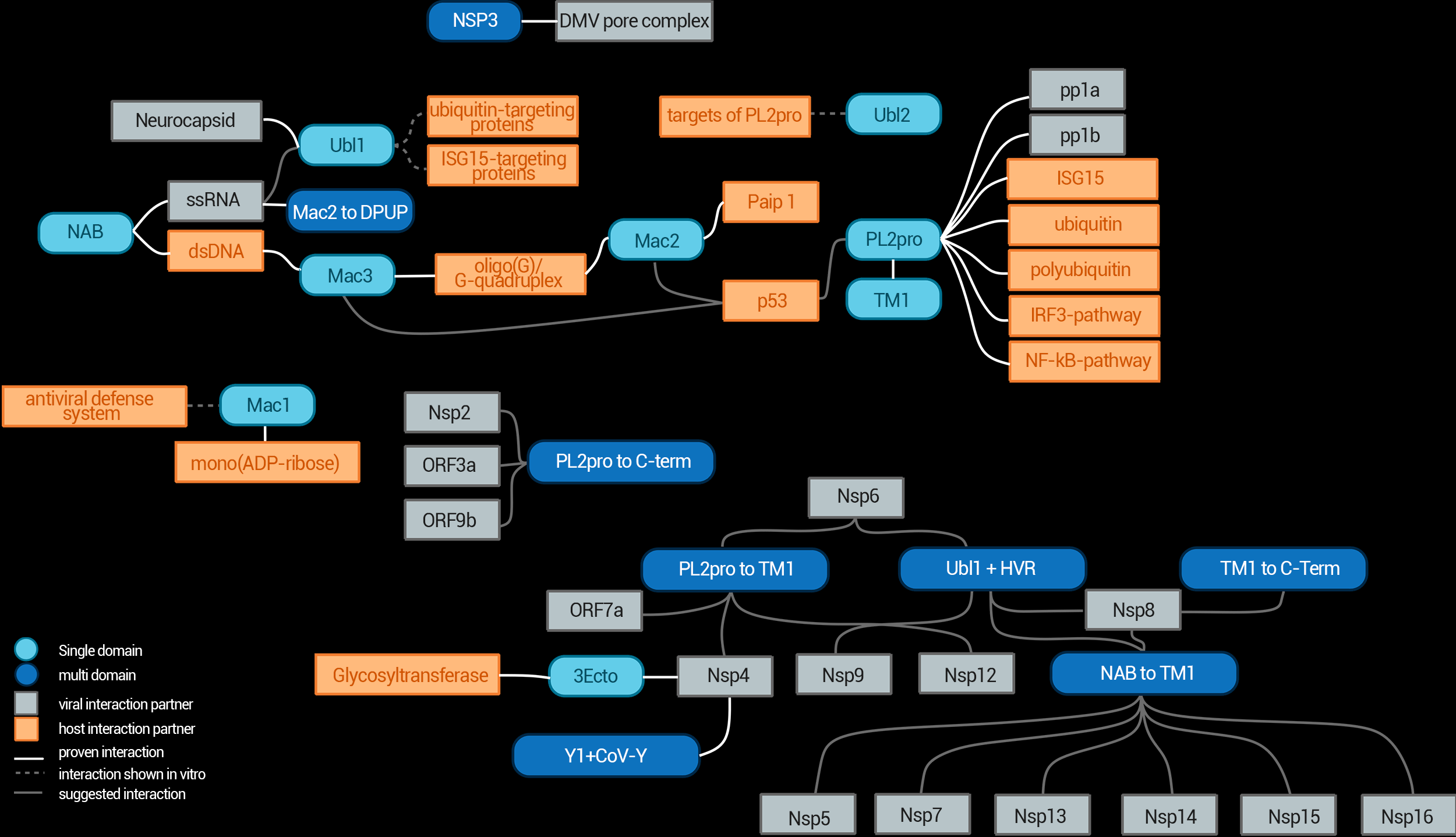
NSP3 Interaction Network
Das Interaktionsnetzwerk von NSP3 und seinen einzelnen Domänen.
Urheber: Coronavirus Structural Task Force - Maximilian Edich
Lizenz: cc-by-sa
Alternativbild:
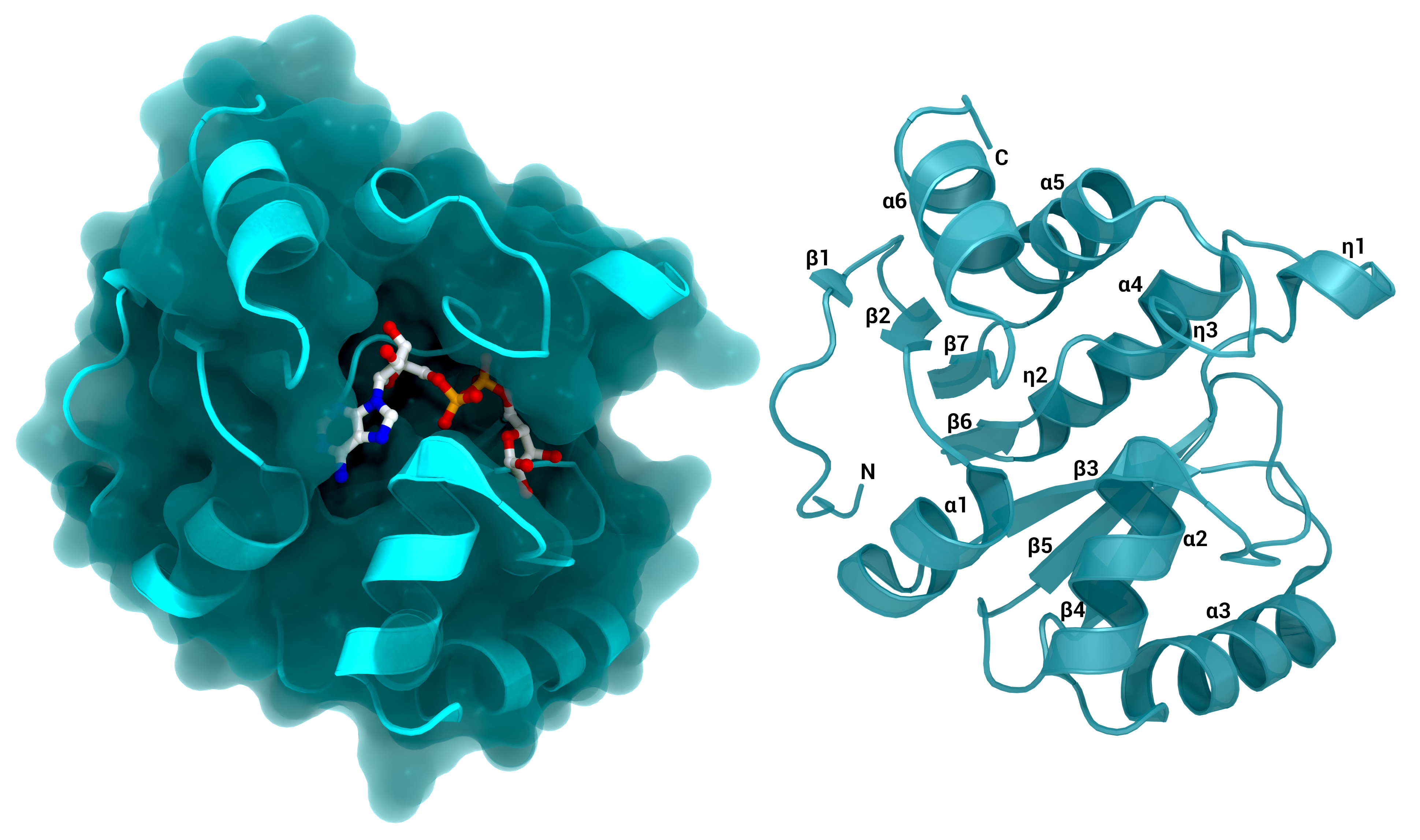
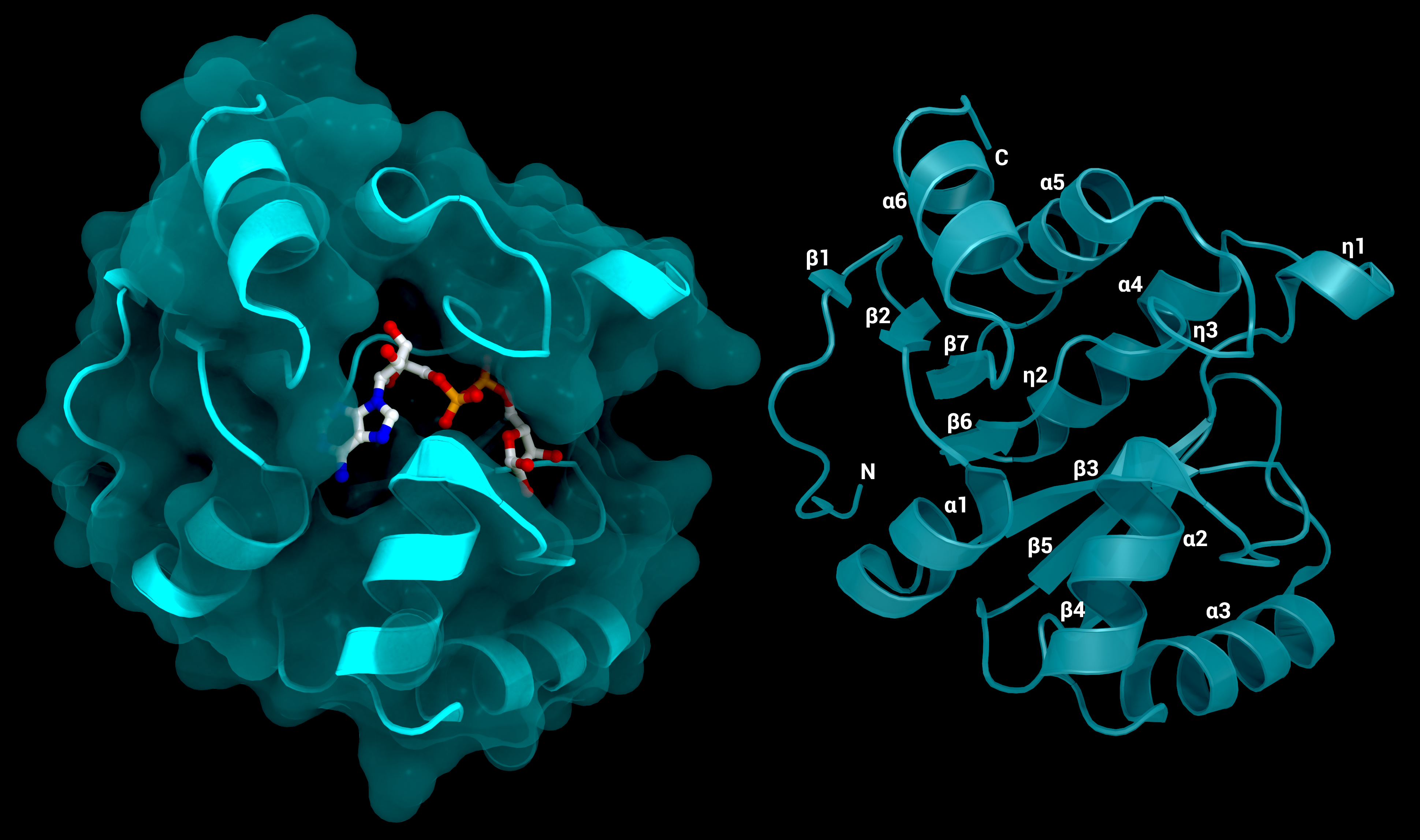
NSP3 Macrodomain 1
Die SARS-CoV-2-Makrodomäne 1 von NSP3. Links: mit Oberfläche und ihrem Substrat ADP-Ribose, PDB ID: 7KQP. Rechts: markierte Struktur ohne Substrat (PDB ID: 7KQO).
Urheber: Coronavirus Structural Task Force - Protein Imager, Lea C. von Soosten
Lizenz: cc-by-sa
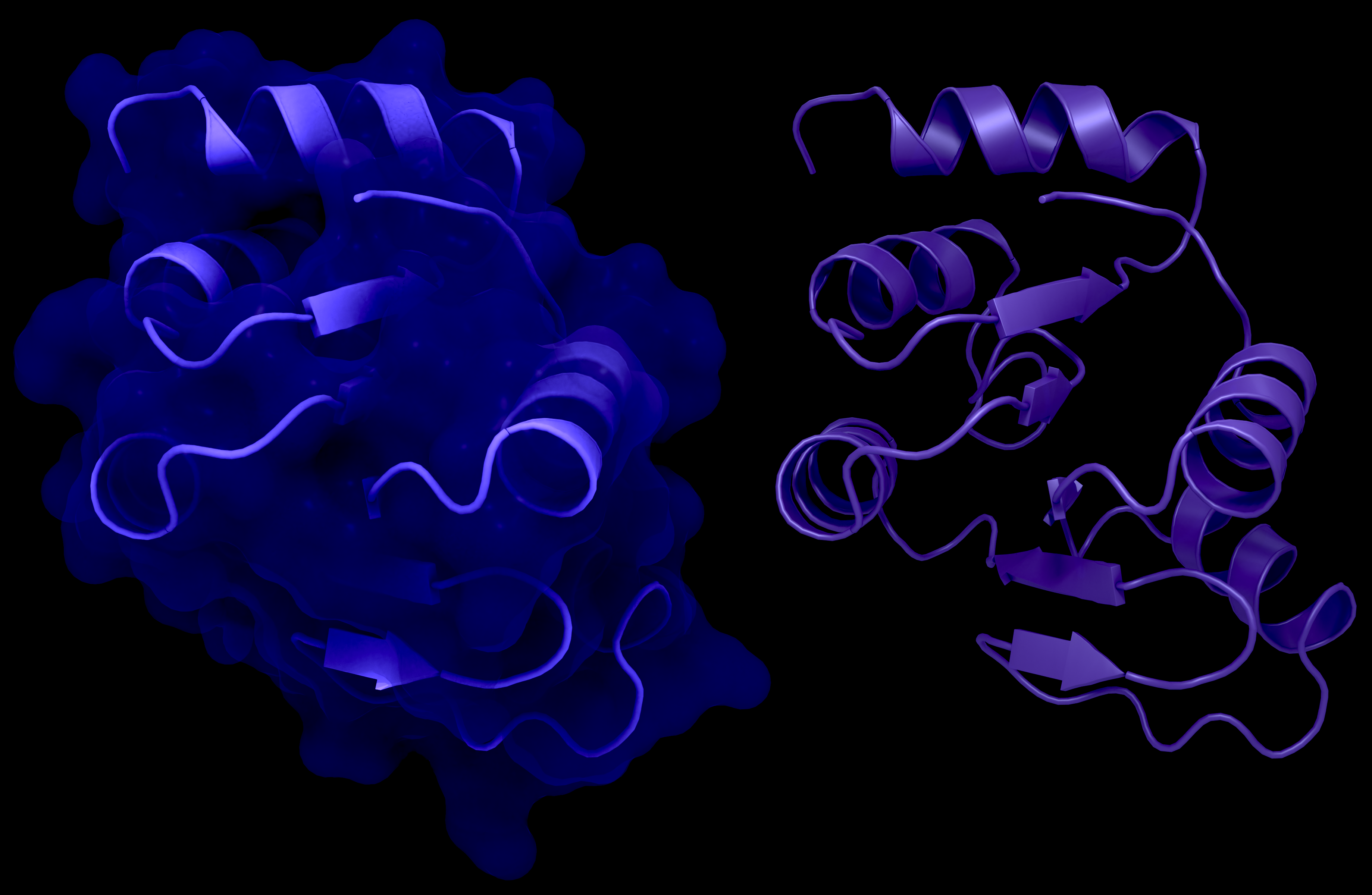
Alternativbild:
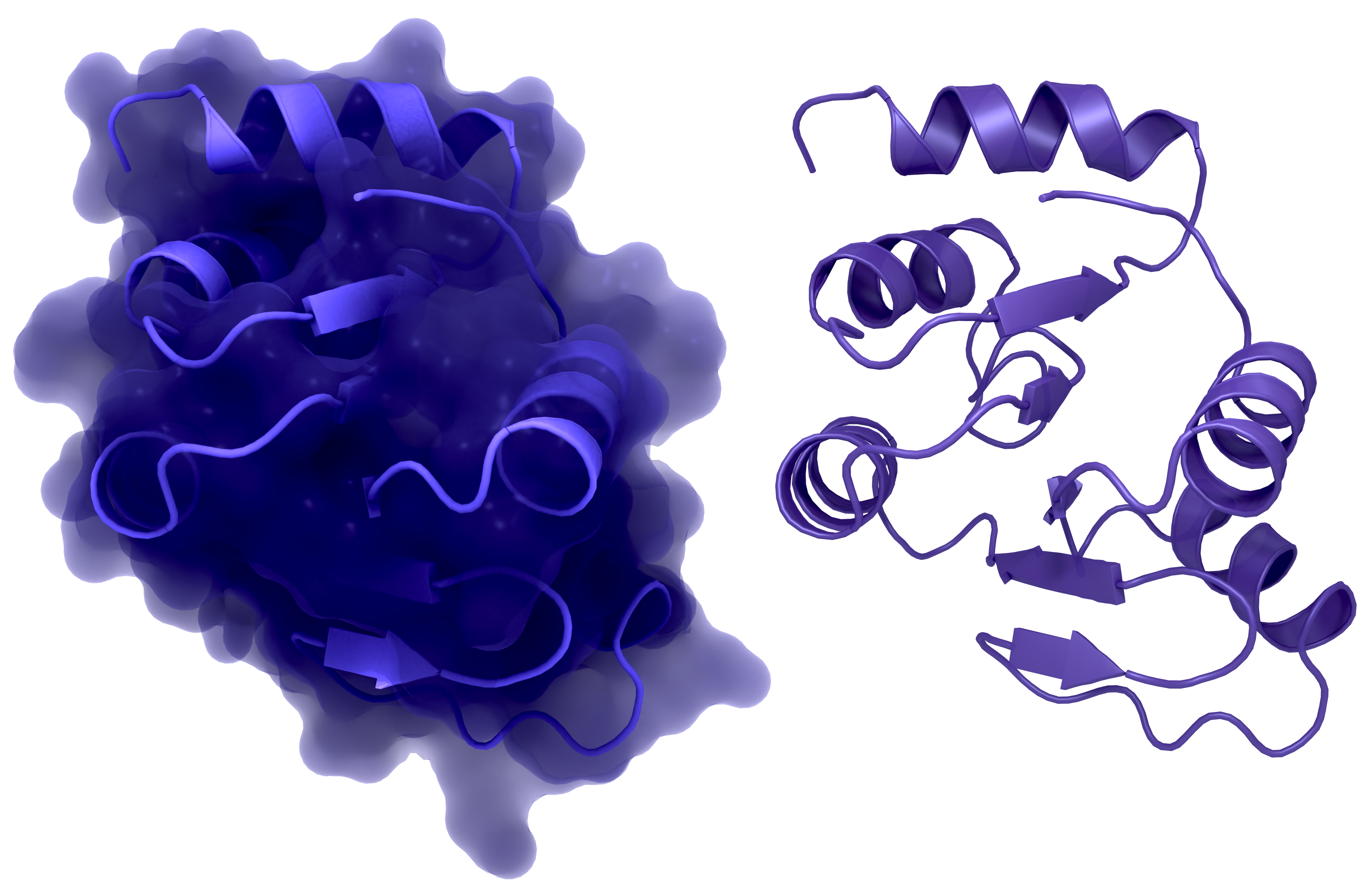
NSP3 Macrodomain 2
NSP3c oder SUD von SARS-CoV-1: a: Struktur der Makrodomäne 2 mit (links) und ohne Oberfläche (rechts), PDB ID: 2W2G, Aminosäuren 389-516. b: Struktur der Makrodomäne 3 mit (links) und ohne Oberfläche (rechts), PDB ID: 2W2G, Aminosäuren 527-652. c: Struktur des DPUP mit (links) und ohne Oberfläche (rechts), PDB ID: 2KQW.
Urheber: Coronavirus Structural Task Force - Protein Imager, Lea C. von Soosten
Lizenz: cc-by-sa
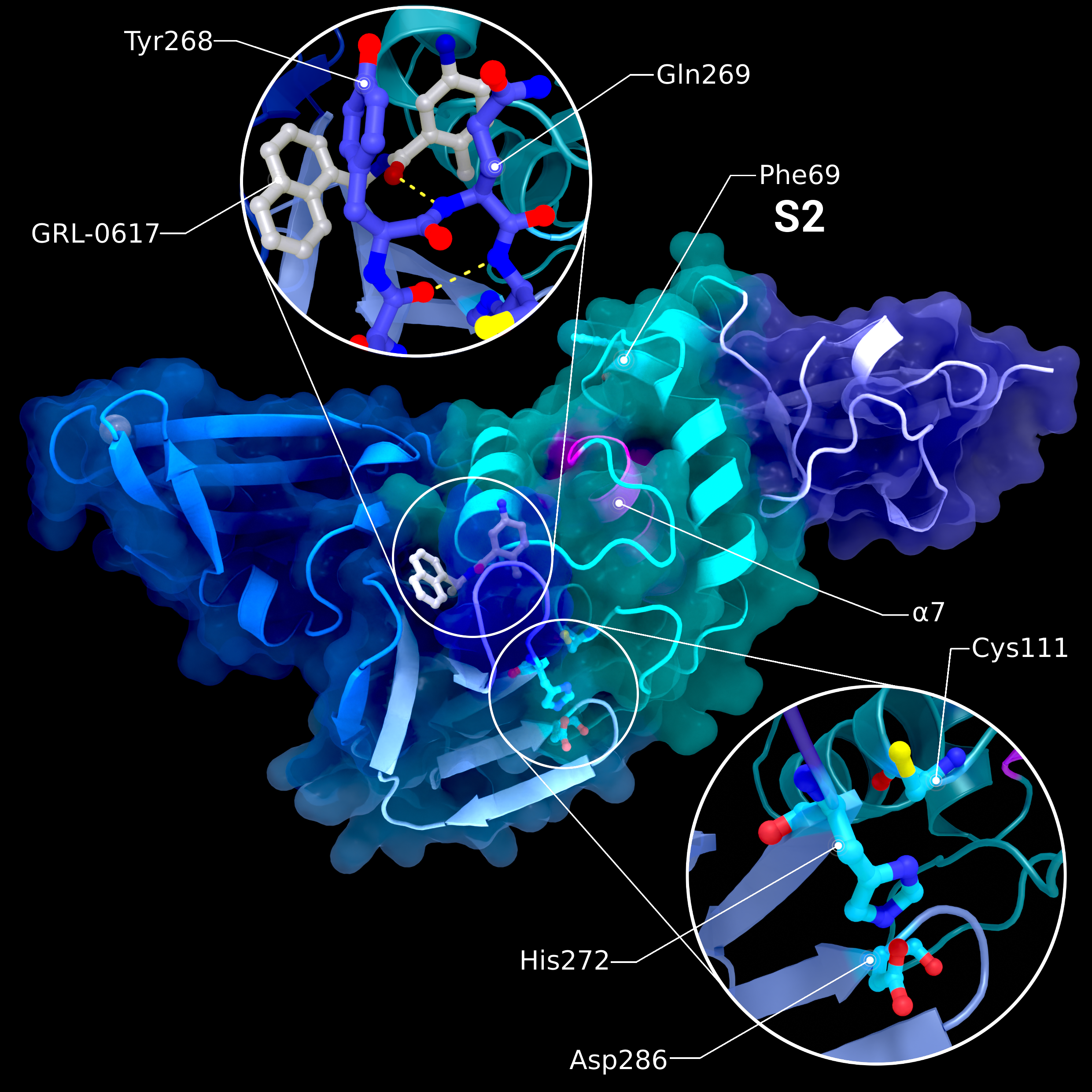
Alternativbild:
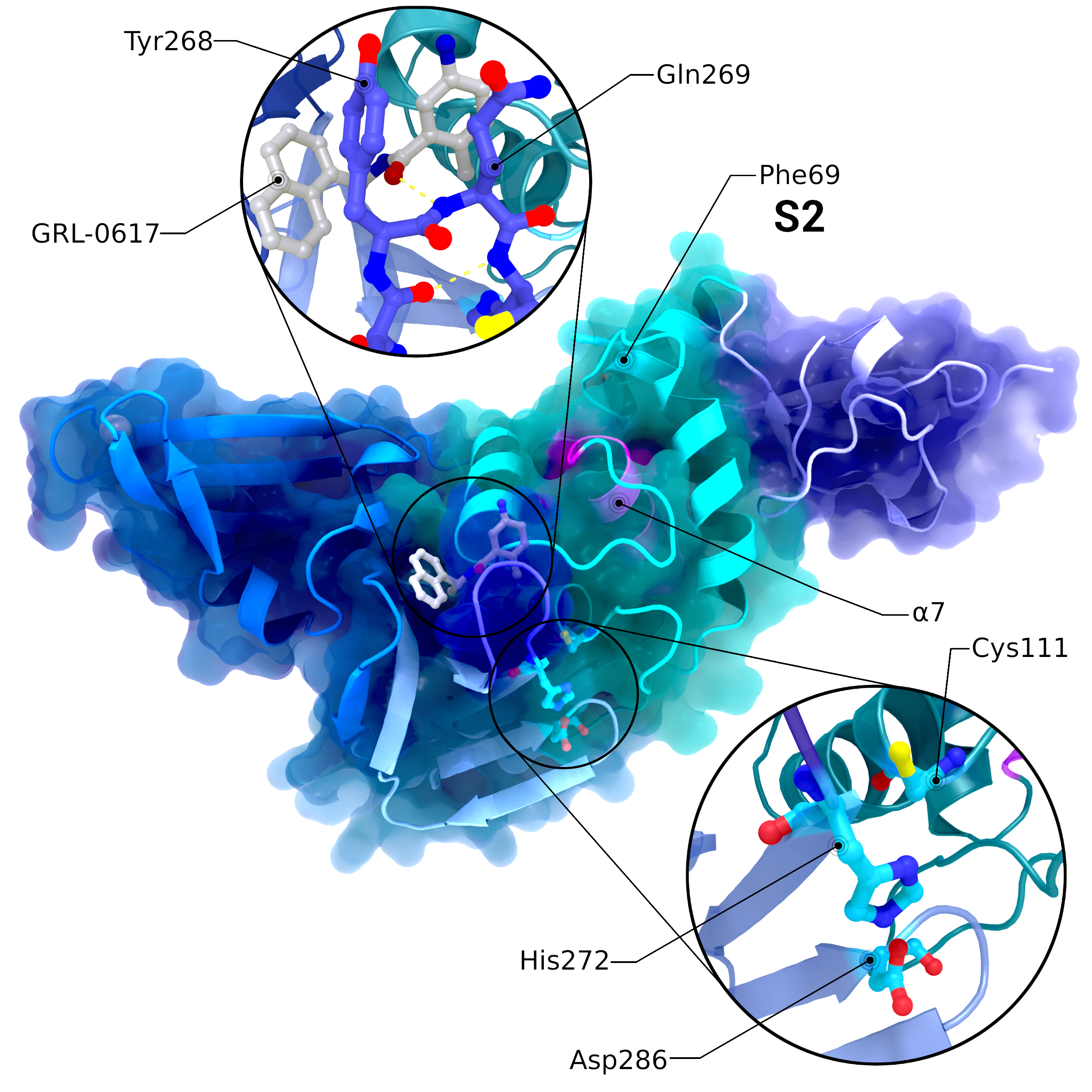
NSP3 PL2pro
Struktur PDB ID 7JRN von PL2pro mit angehängter Ubl2-Domäne (lila) und gebunden an den Inhibitor GRL-0617. Zusätzlich zu Ubl2 besteht PL2pro aus den Finger- (dunkelblau, links), Daumen- (cyan, rechts) und Handflächen-Subdomänen (hellblau, unten). Das katalytisch aktive Zentrum ist in der unteren Übersicht hervorgehoben, die mit dem Inhibitor interagierende Blockierungsschleife in der oberen Übersicht. Die S2-Bindungsstelle befindet sich an Phe69. Die Lage von S1 variiert je nach Substrat und ist daher nicht markiert. Die pinke Helix α7 interagiert mit der ISG15-Bindung an S1.
Urheber: Coronavirus Structural Task Force - Protein Imager, Lea C. von Soosten
Lizenz: cc-by-sa
Alternativbild:
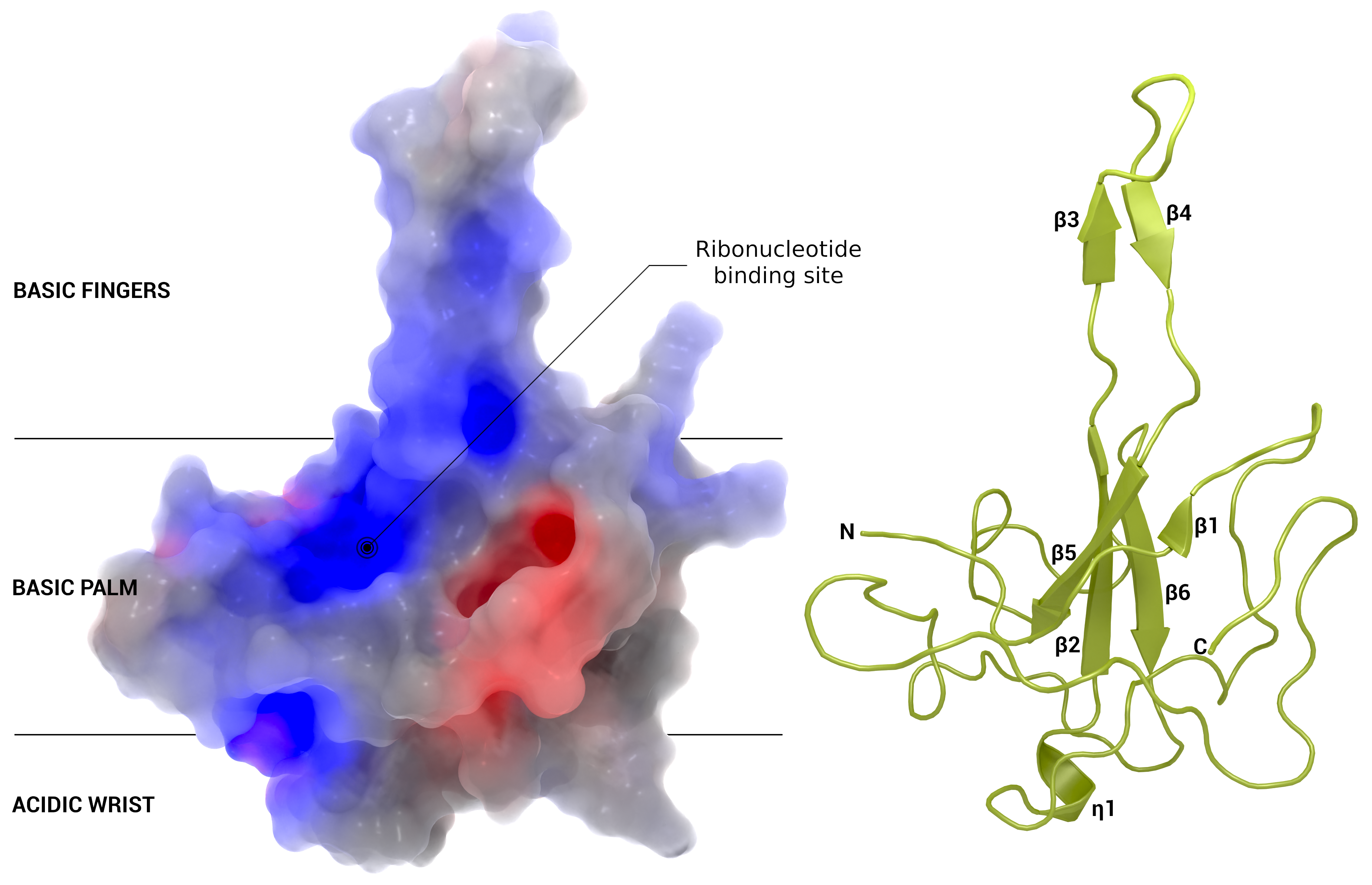
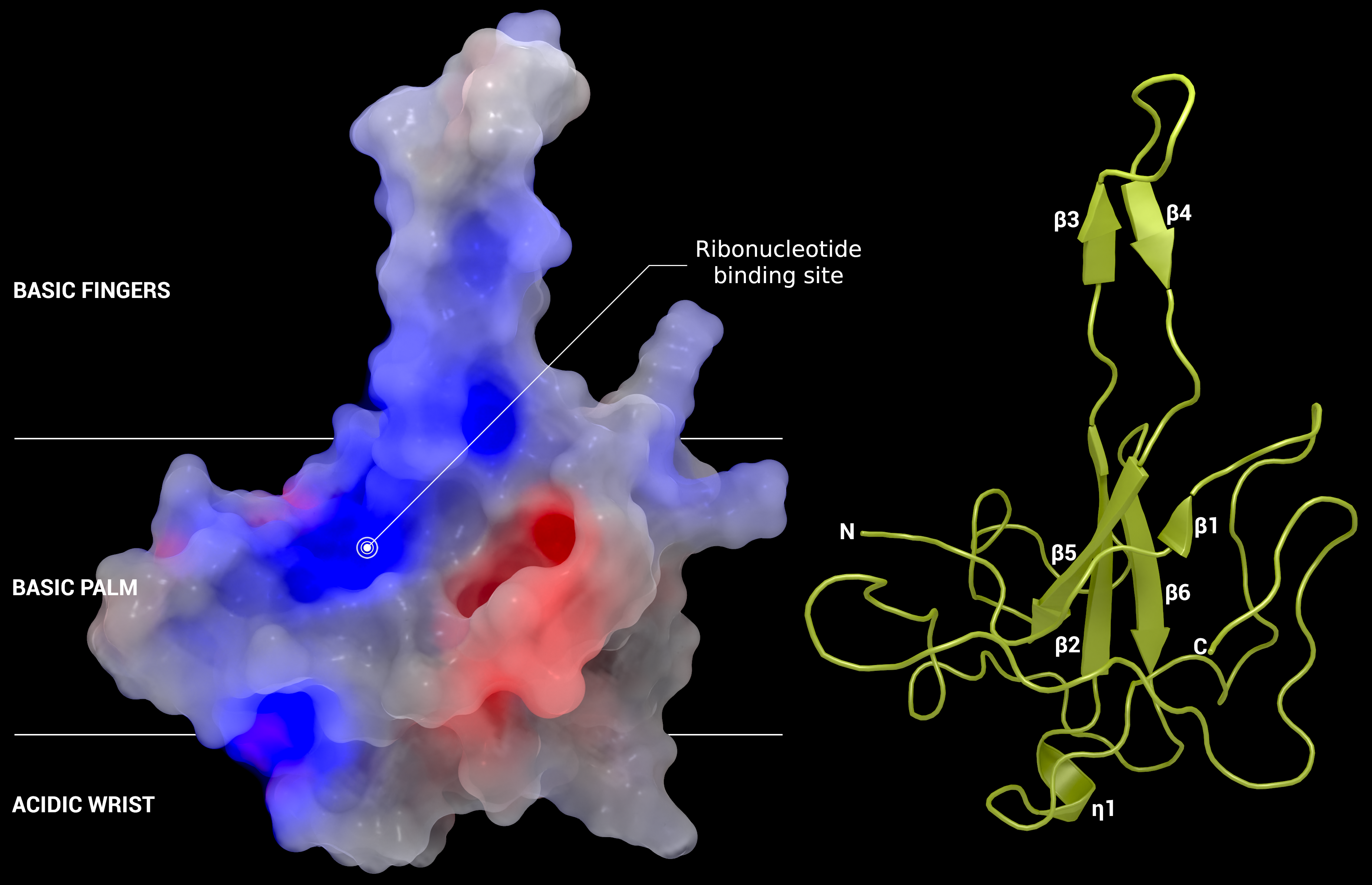
Nucleocapsid
Ausschnitt aus dem Nucleocapsid mit möglicher Bindungsstelle für Wirkstoffe
Urheber: Coronavirus Structural Task Force - Protein Imager, Oliver Kippes
Lizenz: cc-by-sa
