Überblick
Wie die meisten Viren besitzt auch das neuartige Coronavirus SARS-CoV-2 eine schützende Hülle (Envelope), die das wertvolle virale Erbgut umgibt. In dieser Hülle befinden sich verschiedene Strukturproteine, die essentielle Funktionen zur Vermehrung des Virus erfüllen. Ein großer Teil der wissenschaftlichen Forschung konzentriert sich dabei auf das charakteristische Spike-Glykoprotein als therapeutisches Ziel. Doch neben den Spikes enthält die Virushülle weitere Strukturproteine. Das Hüllenprotein (Envelope Protein/ E-Protein), welches aus nur 75 Aminosäuren besteht, ist das kleinste unter ihnen. Obwohl es ein integrales Membranprotein in der Virushülle ist, befindet sich das Hüllenprotein auch im ER, Golgi und ERGIC (ER-Golgi-Zwischenkompartiment) der Wirtszelle [1], wo es für die Virusbildung verantwortlich ist.
Interessanterweise kann die Erforschung dieses Proteins Aufschluss über den Ursprung des neuartigen Coronavirus geben, das derzeit den Alltag auf der ganzen Welt dominiert. Sequenzvergleiche verschiedener E Proteinen bestärken die Vermutung, dass SARS-CoV-2 aufgrund einer hohen Sequenzhomologie vom Bat-CoV (Fledermaus Coronavirus) oder Pangolin-CoV (Pangolin Coronavirus) stammen könnte [2]. Das E-Protein des "älteren Bruders" SARS-CoV weist ebenfalls eine nahezu identische Sequenz mit einer Homologie von 91% auf [2] und wurde durch experimentelle Kernspinresonanz (NMR)-Messungen strukturell bestimmt. Dennoch erweist sich die Aufklärung der 3D-Struktur des SARS-CoV-2 E-Proteins bisher als recht schwierig, weshalb bislang für das E Protein des neuen Coronavirus keine experimentellen Strukturen verfügbar sind [3].
Strukturvergleich mit dem SARS-CoV E Protein
Unser Ziel als Strukturbiologen ist es, die Strukturen möglichst vieler Proteine des neuen Virus aufzudecken und zu verbessern. Solange allerdings noch keine Strukturen des Hüllenproteins von SARS-CoV-2 gemessen und gelöst wurden, kann dies nur durch einen Vergleich mit den Strukturen des SARS-CoV E Proteins erreicht werden.
Topologie und Strukturmerkmale
Die Topologie des SARS-CoV E-Proteins ist hauptsächlich in drei Domänen unterteilt: Ein kurzer hydrophiler N-Terminus, mit einer identische Sequenz in SARS-CoV-2 [3], eine lange hauptsächlich hydrophobe Transmembrandomäne (TMD) und eine lange hydrophile C-terminale Domäne. Studien bezüglich der Frage, ob der C- und der N-Terminus luminal oder zytoplasmatisch sind, lieferten unterschiedliche Ergebnisse, was darauf hindeutet, dass die Topologie des E-Proteins je nach Funktion variieren könnte [3].
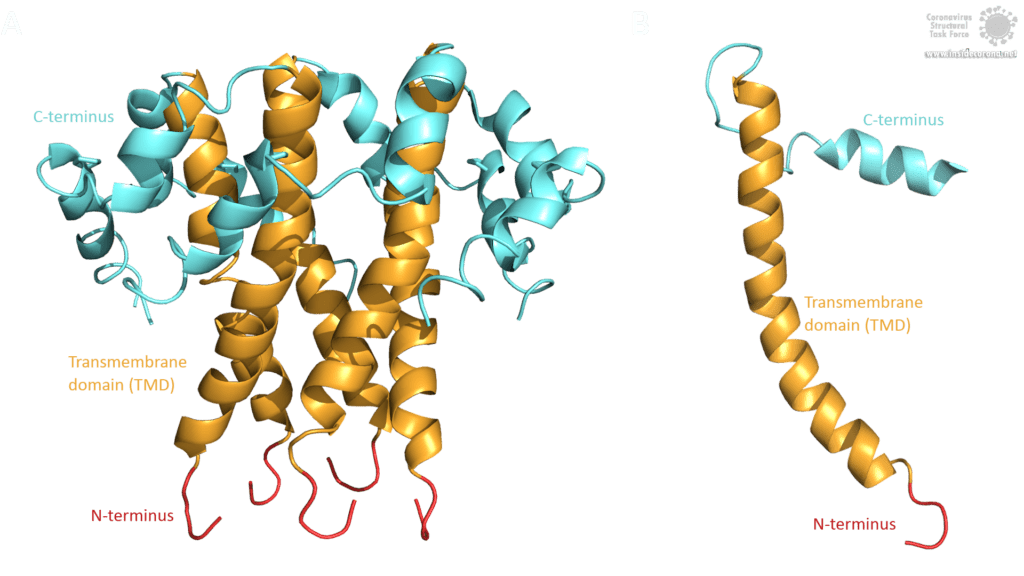
Bild von Luise Kandler
Das E-Protein von SARS-CoV weist mehrere interessante strukturelle Merkmale auf: Eine lange α-Helix mit amphipathischem Anteil bildet die Transmembrandomäne (TMD). Der C-Terminus enthält eine kurze α-Helix, von der man annimmt, dass sie in einem dynamischen Gleichgewicht mit einem seltener vorkommenden β-coil-β-Motiv ist. Beide Helices sind durch eine Windung [4] verbunden. Das β-coil-β-Motiv mit einem konservierten Prolinrest (Pro-54) fungiert möglicherweise als Golgi-Zielsignal und kann seine Konformation wechseln, um die Funktion des E-Proteins in der Wirtszelle zu verändern [4]. Darüber hinaus enthält der C-Terminus ein PDZ-bindendes Motiv (PBM) an der Stelle 73-76 [3]. Diese PBM-Domäne variiert unter Coronaviren leicht, aber die E-Proteine von SARS-CoV, Bat-CoV und SARS-CoV-2 weisen alle das gleiche DLLV-Motiv (Asp-Leu-Leu-Val) auf [2]. Leider sind keine PDB-Strukturen verfügbar, die das β-coil-β-Motiv oder die PBM-Domäne beinhalten.
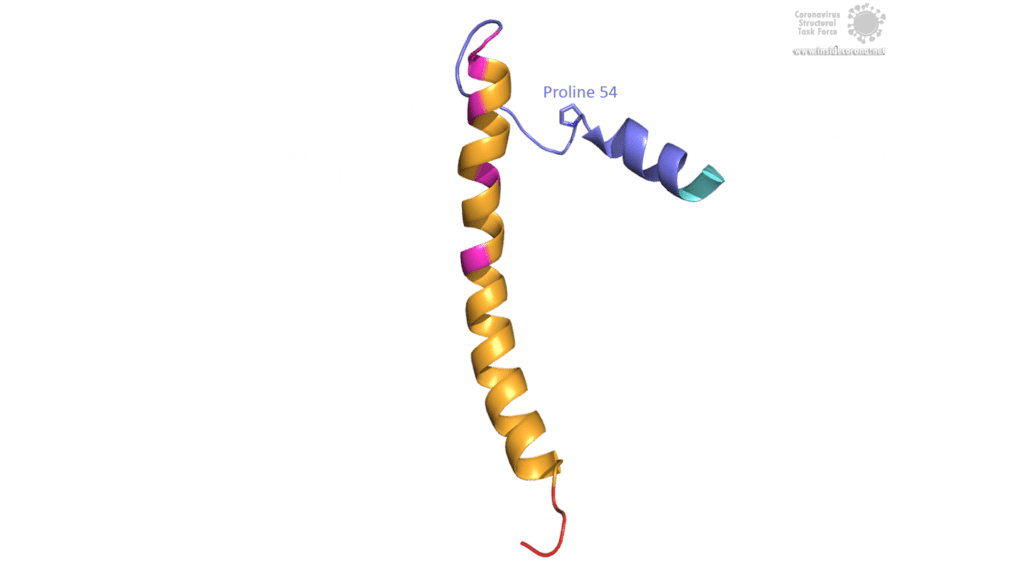
Bild von Luise Kandler
Strukturvarianten - Oligomerisierung und posttranslationale Modifikationen
Das E-Protein kommt in zwei verschiedenen Formen vor. Abgesehen von einer monomeren Struktur oligomerisiert das Protein auch zu einem pentameren Viroporin in der Golgi-Membran der Wirtszelle. Ob es sich bei den E-Proteinen in der Virushülle um Pentamere oder Monomere handelt, ist noch unklar. Die Oligomerisierung wird durch die amphipathische α-Helix der Transmembrandomäne (TMD) [3] induziert und wird hauptsächlich durch die Aminosäure Val-25 sowie die weniger beteiligte Aminosäure Asn-15 vermittelt [3]. Beide Aminosäuren sind auch in SARS-CoV-2 konserviert. Um die Pore in der Golgi-Membran zu verankern, sind die hydrophoben Aminosäuren der TMD den hydrophoben Phospholipiden der Membran zugewandt. Zusätzlich interferieren positiv geladene Aminosäuren über elektrostatische Wechselwirkungen mit den negativ geladenen Phospholipiden [3].
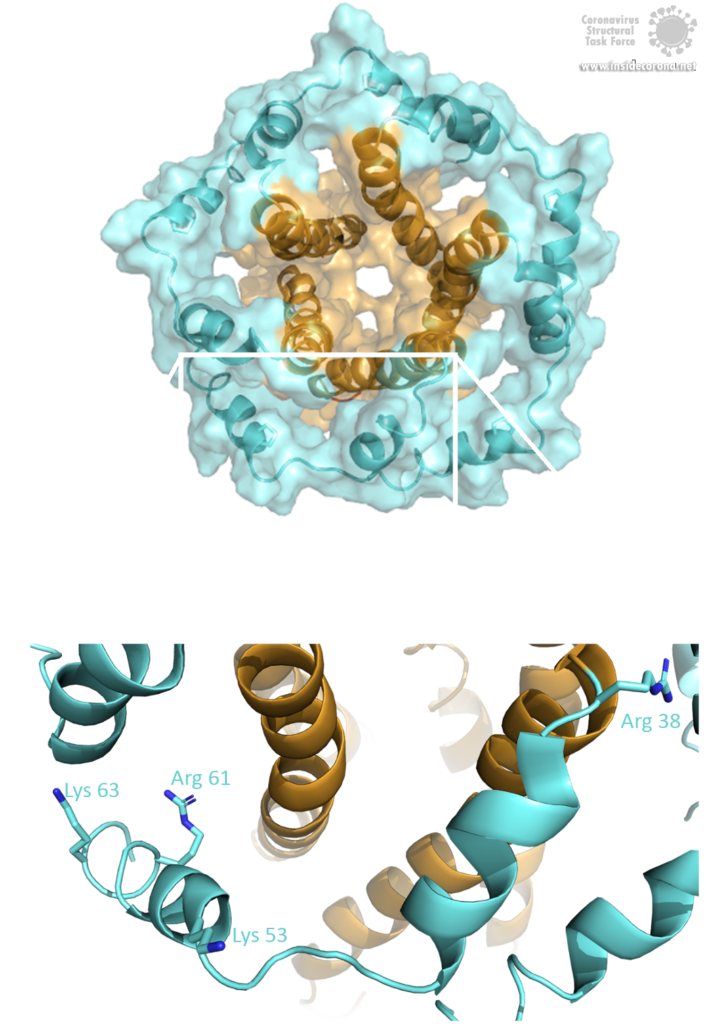
Bild von Luise Kandler
Andere Strukturvarianten entstehen durch posttranslationale Modifikationen, die im E-Protein von SARS-CoV und anderen Coronaviren nachgewiesen wurden. Palmitoylierung bezeichnet das Anheften von Palmitinfettsäure an Cysteinreste, was die Hydrophobie des Proteins erhöht. Folglich trägt Palmitoylierung zur Membranverankerung des E-Proteins bei und dient wahrscheinlich auch als zusätzliches Golgi-Zielsignal. Die Ubiquitinierung des E-Proteins löst dessen Abbau aus und könnte folglich als negative Regulation der E-Protein-Konzentration fungieren [3]. Es wurde festgestellt, dass der optimale E-Protein-Level in der Wirtszelle wichtig für eine erfolgreiche Produktion neuer Viren ist. Eine weitere Modifikation, die Glykosylierung, fügt Oligosaccharidfragmente zu Asparaginresten in einem bestimmten Motiv (Asn-X-Ser/Thr) hinzu, das ebenfalls im E Protein konserviert ist. Bei SARS-CoV wurde nachgewiesen, dass die in das Motiv Asn-Ser-Ser eingebettete Aminosäure Asn-66 glykosyliert wird. Die Glykosylierung könnte der Abwehr des Wirt-Immunsystems und der Rekrutierung von Chaperon-Proteinen dienen, die die korrekte Faltung neu synthetisierter Virus-Proteine unterstützen. Experimentelle Daten deuten außerdem darauf hin, dass die Glykosylierung von Asn-66 die monomeren Funktionen des E-Proteins fördert, indem sie die Oligomerisierung verhindert [3].
Die Struktur gibt Aufschluss über die verschiedenen Funktionen
Das Aufklären der biologischen Funktion eines Moleküls ist das Hauptziel der experimentellen Strukturbestimmung. Nur wenn die atomare Struktur eines viralen Proteins bekannt ist, kann ein passendes Medikament gefunden werden, das gezielt die Funktionen dieses Protein unterdrückt. Das E Protein kann diverse strukturelle Konformationen annehmen und somit viele verschiedene Funktionen erfüllen, sowohl als Monomer als auch als Pentamer.
Funktionen als Monomer: Golgi-Targeting und virale Assemblierung
Das E-Protein trägt ein Golgi-Ziel-Signal im β-coil-β-Motiv des C-Terminus und ein weiteres in der N-terminalen Domäne. Außerdem wird angenommen, dass die Palmitoylierung an dieser Funktion beteiligt ist. Dementsprechend wird das E-Protein nach seiner Translation am ER zur Golgi-Membran transportiert. Von dort beschafft sich das Virus die Membran für eine neue Virushülle [3]. Sobald sich das Protein am Golgi befindet, ist seine Hauptaufgabe als Monomer die virale Assemblierung, d.h. die Versammlung aller viralen Makromoleküle (Proteine und RNA-Genom) zur Bildung eines virusähnlichen Partikels (Virus-like Particle). Während der Assemblierung wächst der Virus-like Particle im Lumen des ERGIC und folgt dem Sekretionsweg der Wirtszelle. Mehrere Experimente bestätigen die Beteiligung des Hüllenproteins zusammen mit dem Membranprotein (M) an diesem Prozess. Es wird angenommen, dass das E-Protein dabei eher die Krümmung und Spaltung der Golgi-Membran induziert, während das M-Protein den Prozess koordiniert. Dennoch produzieren SARS-CoV-infizierte Zellen auch in Abwesenheit des E-Proteins Virus-like Particles, jedoch ist deren Transport zur Zelloberfläche und deren Sekretion behindert, was zu einer geringeren Anzahl reifer Virionen, einer atypischen Morphologie und einer höheren Rate an vermehrungsunfähigen Virionen führt [3]. Der genauen Mechanismus hinter der Membranbildung für die Virushülle ist allerdings noch unklar.
Nachdem die Virionen durch den Sekretionsweg transportiert wurden, werden sie als reife Virionen aus der Wirtszelle freigesetzt. Das Ablösen von der Wirtsmembran wird als „Scission“ (Spaltung) bezeichnet und wird entweder durch die viruseigenen Scission-Proteine oder durch die Spaltmaschinerie der Wirtszelle (ESCRT) koordiniert. Welcher Fall für SARS-CoV-2 zutrifft, ist noch unklar. Virus-infizierte Zellen, denen die Spaltmaschinerie fehlt, weisen eine „Perlenschnur“-Morphologie auf, bei der die Virionen in länglicher Form an der Wirtszellmembran kleben bleiben. Diese Morphologie wurde in Influenza-infizierten Zellen gefunden, denen das M2-Protein fehlt, was beweist, dass M2 in Influenza Viren am Scission-Prozess beteiligt ist. Da man davon ausgeht, dass das E-Protein von SARS-CoV aufgrund struktureller Ähnlichkeiten funktionell analog zu M2 ist, wird angenommen, dass das E-Protein ebenfalls am Spaltprozess beteiligt ist [3].
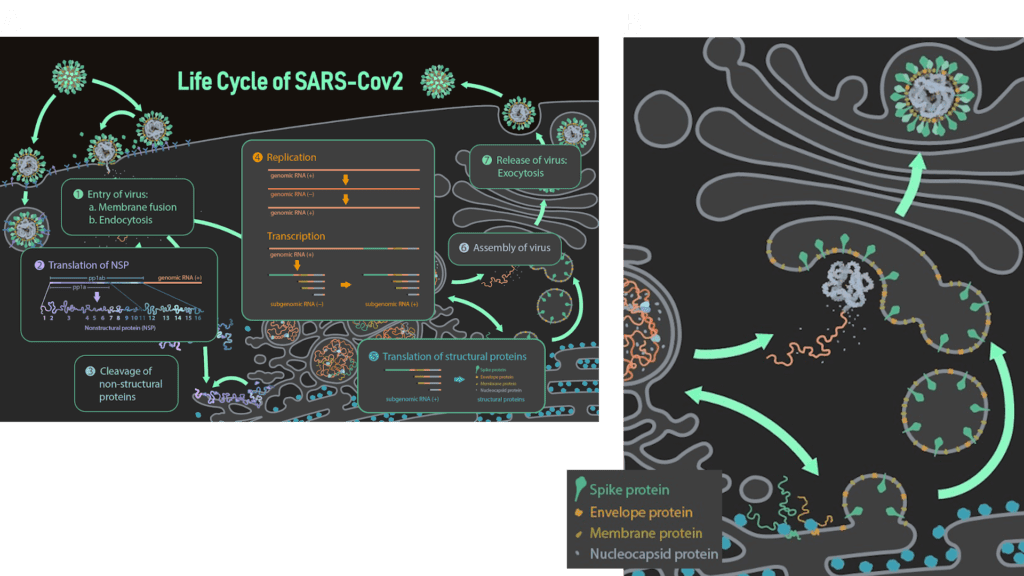
Bild von Ann (Hui) Liu, https://animationlab.utah.edu/ [5]
Funktionen als Pentamer: Ionen-Kanal-Aktivität
Einige E-Proteine oligomerisieren am Golgi und bilden pentamere Viroporine, die als Ionenkanäle fungieren. Sie transportieren hauptsächlich Na+ und K+, aber sind auch durchlässig für Ca2+- und H+-Ionen. Auch wenn der primäre Zweck des Kationentransports noch nicht klar ist, wird angenommen, dass der Ca2+-Transport die Entzündungsreaktion bei akutem Atemnotsyndrom auslöst[7].
Die Aminosäure Asn-15 wirkt dabei wahrscheinlich als eine Art „Filter“ für die Ionenselektivität [6], die zusätzlich durch die Ladung der Lipidkopfgruppe der Membran beeinflusst werden kann. Die Abwesenheit des E Proteins in seiner pentameren Form zeigt, dass seine Funktion als Ionenkanal für die virale Replikation zwar nicht essentiell ist, aber die Virulenz dennoch abschwächt [8].
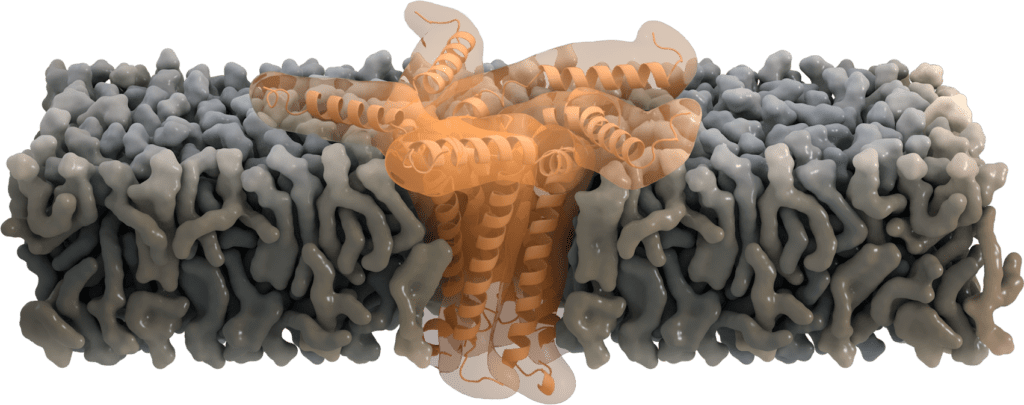
Bild von Thomas Splettstoesser, www.scistyle.com
Pathogenese und das E-Protein als potentieller Angriffspunkt für Medikamente
Interaktionen von viralen Proteinen mit Wirtszellproteinen deregulieren viele physiologische Prozesse. Bei Patienten, die mit SARS-CoV infiziert wurden, tragen diese deregulierenden Protein-Protein-Interaktionen wesentlich zur Pathogenese bei. Einige der SARS-CoV Symptome sind auch bei SARS-CoV-2-infizierten Patienten zu beobachten.
Interaktionen des Hüllenproteins mit Proteinen der Wirtszelle werden durch das PDZ-bindende Motiv (PBM) am Ende des C-Terminus vermittelt. Das Motiv bindet an die PDZ-Domäne von Adaptorproteinen, die anschließend von weiteren zellulären Proteinen gebunden werden, wodurch eine Signalkaskade aktiviert wird, die zur Pathogenese auf zellulärer Ebene beitragen kann. Von einigen dieser Interaktionen weiß man, dass sie Symptome wie Lymphopenie [9], Veränderungen des Blutdrucks und der Wasserhomöostase sowie Gewebeschäden, Ödeme und das akute Atemnotsyndrom (ARDS) [10] auslösen. Grund dafür ist eine ausgelöste Signalkaskade, die zu einer Überexpression von entzündlichen Zytokinen (die ebenfalls durch das Leitprotein nsp1 reguliert werden) führt. Eine weitere Protein-Protein-Interaktion zerstört die „Tight Junctions“ der Lungenepithelzellen. Folgen sind das Versagen der epithelialen Barriere, das Durchbrechen der Alveolarwand durch Virione und schließlich eine systemische Infektion [11]. O. Wittekindt schreibt [12]: "Der Zusammenbruch der epithelialen Barriere ist charakteristisch für Atemnotsyndrome (...)". Darüber hinaus aktiviert die Ionenkanal-Funtion des E-Proteins durch Ca2+-Transport den entzündungsfördernden Signalweg, was bei infizierten Mäusen zu Lungenschäden führte [7]. Die Hemmung des E-Protein-Viroporins durch Hexamethylenamilorid (HMA) [8] reduziert die Aktivierung des Inflammasoms, was den Ionenkanal des E-Proteins zu einem potenziellen therapeutischen Ziel macht. Als Teil der viralen Abwehr wird von der infizierten Zelle die ER-Stress-Reaktion aktiviert, sobald die Proteinfaltungskapazität des ER durch zusätzliche Expression viraler Proteine überlastet wird. Dies kann zur Apoptose der Wirtszelle führen. Experimente bestätigen jedoch, dass das E-Protein zur Pathogenese beiträgt, indem es die ER-Stress-Reaktion unterdrückt, um das Überleben der Wirtszelle zu sichern [3].
Als potenzielles Ziel für eine medikamentöse Behandlung sind die Protein-Protein-Interaktionen des E-Proteins vielversprechend. Seine PBM-Domäne kann zelluläre Proteine binden, die an der Pathogenese beteiligt sind. Eine experimentelle Verkürzung dieser Domäne zeigt, dass es möglich sein könnte, einen Lebendimpfstoff mit einer mutierten, aber intakten PBM-Domäne und folglich abgeschwächter Pathogenität zu finden. Die Identifizierung weiterer Interaktions-Partnern ist dennoch wichtig, um eine gezieltere Therapie zu ermöglichen. Das Fehlen des gesamten E-Proteins führt darüber hinaus zu unvollständiger Virusreifung und vermehrungsdefekten Nachkommen [3], sodass E-Protein-defiziente Virionen ebenfalls ein potenzieller Impfstoffkandidat sind.
Zusammenfassend kann man sagen, dass das E-Protein von SARS-CoV-2 ein weiterer wertvoller Angriffspunkt für medikamentöse Behandlungen ist. Zwar gibt uns sein „älterer Bruder“, das SARS-CoV E-Protein, schon eine gute Vorhersage über mögliche Strukturen und Funktionen, aber dennoch wäre eine experimentelle Strukturbestimmung des SARS-CoV-2 E-Proteins erstrebenswert. Doch bis dahin bleibt das E Protein von SARS-CoV-2 eine kleine, mysteriöse Struktur.
Die besten PDB-Einträge
- 2MM4: Diese NMR-Struktur zeigt das SARS-CoV E-Protein als Monomer. Die Struktur beinhaltet die Transmembran-Domäne komplett und den C- sowie den N-Terminus teilweise. Die Struktur ist an der Membranbiegung, Membranspaltung und an der viralen Assemblierung beteiligt.
- 5X29: Diese NMR-Struktur zeigt das SARS-CoV E-Protein als Oligomer. Sie ist ein Pentamer aus fünf identischen Monomeren und fungiert als membran-verankerter Ionenkanal. Die Struktur beinhaltet die Transmembran-Domäne komplett und den C- sowie den N-Terminus teilweise.
Literatur
[1] J. Nieto-Torres, M. DeDiego, E. Álvarez, J. Jiménez-Guardeño, J. Regla-Nava, M. Llorente, et al.: Subcellular location and topology of severe acute respiratory syndrome coronavirus envelope protein, Virology, 2011
[2] M. Bianchi, D. Benvenuto, M. Giovanetti, S. Angeletti, M. Ciccozzi, S. Pascarella: Sars-CoV-2 Envelope and Membrane proteins: differences from closely related proteins linked to cross-species transmission, Preprint, 2020
[3] D. Schoeman, B. Fielding: Coronavirus envelope protein: current knowledge, Virology Journal, 2019
[4] Y. Li, W. Surya, S. Claudine, J. Torres: Structure of a Conserved Golgi Complex-targeting Signal in Coronavirus Envelope Proteins, The Journal Of Biological Chemistry, 2014
[5] Ann (Hui) Liu, in https://animationlab.utah.edu/
[6] K. Pervushin, E. Tan, K. Parthasarathy, X. Lin, F. Jiang, D. Yu, A. Vararattanavech, T. Soong, D. Liu, J. Torres: Structure and Inhibition of the SARS Coronavirus Envelope Protein Ion Channel, PloS Pathogens, 2009
[7] J. Nieto-Torres, C. Verdiá-Báguena, J. Jimenez-Guardeño, J. Regla-Nava, C. Castaño-Rodriguez, R. Fernandez-Delgado, et al.: Severe acute respiratory syndrome coronavirus E protein transports calcium ions and activates the NLRP3 inflammasome, Virology, 2015
[8] J. Nieto-Torres, M. DeDiego, C. Verdiá-Báguena, J. Jimenez-Guardeño, J. Regla-Nava, R. Fernandez-Delgado, et al.: Severe acute respiratory syndrome coronavirus envelope protein ion channel activity promotes virus fitness and pathogenesis, PLoS Pathogens, 2014
[9] Y. Yang, Z. Xiong, S. Zhang, Y. Yan, J. Nguyen, B. Ng, et al.: Bcl-xL inhibits T-cell apoptosis induced by expression of SARS coronavirus E protein in the absence of growth factors, Biochemical Journal, 2005
[10] J. Jimenez-Guardeño, J. Nieto-Torres, M. DeDiego, J. Regla-Nava, R. Fernandez-Delgado, C. Castaño-Rodriguez, et al.: The PDZ-binding motif of severe acute respiratory syndrome coronavirus envelope protein is a determinant of viral pathogenesis, PLoS Pathogens, 2014
[11] K. Teoh, Y. Siu, W. Chan, M. Schlüter, C. Liu, J. Peiris, et al.: The SARS coronavirus E protein interacts with PALS1 and alters tight junction formation and epithelial morphogenesis, Mol Biol Cell, 2010
[1] J. Nieto-Torres, M. DeDiego, E. Álvarez, J. Jiménez-Guardeño, J. Regla-Nava, M. Llorente, et al.: Subcellular location and topology of severe acute respiratory syndrome coronavirus envelope protein, Virology, 2011
[2] M. Bianchi, D. Benvenuto, M. Giovanetti, S. Angeletti, M. Ciccozzi, S. Pascarella: Sars-CoV-2 Envelope and Membrane proteins: differences from closely related proteins linked to cross-species transmission, Preprint, 2020
[3] D. Schoeman, B. Fielding: Coronavirus envelope protein: current knowledge, Virology Journal, 2019
[4] Y. Li, W. Surya, S. Claudine, J. Torres: Structure of a Conserved Golgi Complex-targeting Signal in Coronavirus Envelope Proteins, The Journal Of Biological Chemistry, 2014
[5] Ann (Hui) Liu, in https://animationlab.utah.edu/
[6] K. Pervushin, E. Tan, K. Parthasarathy, X. Lin, F. Jiang, D. Yu, A. Vararattanavech, T. Soong, D. Liu, J. Torres: Structure and Inhibition of the SARS Coronavirus Envelope Protein Ion Channel, PloS Pathogens, 2009
[7] J. Nieto-Torres, C. Verdiá-Báguena, J. Jimenez-Guardeño, J. Regla-Nava, C. Castaño-Rodriguez, R. Fernandez-Delgado, et al.: Severe acute respiratory syndrome coronavirus E protein transports calcium ions and activates the NLRP3 inflammasome, Virology, 2015
[8] J. Nieto-Torres, M. DeDiego, C. Verdiá-Báguena, J. Jimenez-Guardeño, J. Regla-Nava, R. Fernandez-Delgado, et al.: Severe acute respiratory syndrome coronavirus envelope protein ion channel activity promotes virus fitness and pathogenesis, PLoS Pathogens, 2014
[9] Y. Yang, Z. Xiong, S. Zhang, Y. Yan, J. Nguyen, B. Ng, et al.: Bcl-xL inhibits T-cell apoptosis induced by expression of SARS coronavirus E protein in the absence of growth factors, Biochemical Journal, 2005
[10] J. Jimenez-Guardeño, J. Nieto-Torres, M. DeDiego, J. Regla-Nava, R. Fernandez-Delgado, C. Castaño-Rodriguez, et al.: The PDZ-binding motif of severe acute respiratory syndrome coronavirus envelope protein is a determinant of viral pathogenesis, PLoS Pathogens, 2014
[11] K. Teoh, Y. Siu, W. Chan, M. Schlüter, C. Liu, J. Peiris, et al.: The SARS coronavirus E protein interacts with PALS1 and alters tight junction formation and epithelial morphogenesis, Mol Biol Cell, 2010
[1] J. Nieto-Torres, M. DeDiego, E. Álvarez, J. Jiménez-Guardeño, J. Regla-Nava, M. Llorente, et al.: Subcellular location and topology of severe acute respiratory syndrome coronavirus envelope protein, Virology, 2011
[2] M. Bianchi, D. Benvenuto, M. Giovanetti, S. Angeletti, M. Ciccozzi, S. Pascarella: Sars-CoV-2 Envelope and Membrane proteins: differences from closely related proteins linked to cross-species transmission, Preprint, 2020
[3] D. Schoeman, B. Fielding: Coronavirus envelope protein: current knowledge, Virology Journal, 2019
[4] Y. Li, W. Surya, S. Claudine, J. Torres: Structure of a Conserved Golgi Complex-targeting Signal in Coronavirus Envelope Proteins, The Journal Of Biological Chemistry, 2014
[5] Ann (Hui) Liu, in https://animationlab.utah.edu/
[6] K. Pervushin, E. Tan, K. Parthasarathy, X. Lin, F. Jiang, D. Yu, A. Vararattanavech, T. Soong, D. Liu, J. Torres: Structure and Inhibition of the SARS Coronavirus Envelope Protein Ion Channel, PloS Pathogens, 2009
[7] J. Nieto-Torres, C. Verdiá-Báguena, J. Jimenez-Guardeño, J. Regla-Nava, C. Castaño-Rodriguez, R. Fernandez-Delgado, et al.: Severe acute respiratory syndrome coronavirus E protein transports calcium ions and activates the NLRP3 inflammasome, Virology, 2015
[8] J. Nieto-Torres, M. DeDiego, C. Verdiá-Báguena, J. Jimenez-Guardeño, J. Regla-Nava, R. Fernandez-Delgado, et al.: Severe acute respiratory syndrome coronavirus envelope protein ion channel activity promotes virus fitness and pathogenesis, PLoS Pathogens, 2014
[9] Y. Yang, Z. Xiong, S. Zhang, Y. Yan, J. Nguyen, B. Ng, et al.: Bcl-xL inhibits T-cell apoptosis induced by expression of SARS coronavirus E protein in the absence of growth factors, Biochemical Journal, 2005
[10] J. Jimenez-Guardeño, J. Nieto-Torres, M. DeDiego, J. Regla-Nava, R. Fernandez-Delgado, C. Castaño-Rodriguez, et al.: The PDZ-binding motif of severe acute respiratory syndrome coronavirus envelope protein is a determinant of viral pathogenesis, PLoS Pathogens, 2014
[11] K. Teoh, Y. Siu, W. Chan, M. Schlüter, C. Liu, J. Peiris, et al.: The SARS coronavirus E protein interacts with PALS1 and alters tight junction formation and epithelial morphogenesis, Mol Biol Cell, 2010
[12] O. Wittekindt: Tight junctions in pulmonary epithelia during lung inflammation, Springer Verlag, 2016
