Für diesen Beitrag steht leider keine deutsche Übersetzung zur Verfügung.
Introduction
Before I started writing this article, the first thing I did was to google the name of my protein “NendoU” and was greeted by Figure 1. Needless to say, this is not what I was expecting. So, if you’re an anime fan looking for Riki Nendou, a dutiful yet dull-witted boy who likes helping people, particularly prioritising the weak, from The Disastrous Life of Saiki K: I’m afraid you have come to the wrong place. However, now that you’re here, maybe you’d like to learn about an interesting protein involved in SARS-CoV-2 viral replication? It can bind to and process six RNA molecules at a time! Six!
After that interlude, I should get this blog post back on track! So… viruses and proteins. SARS-CoV-2 is an enveloped coronavirus with a non-segmented positive-sense RNA genome, in English this means the RNA genome in SARS-COV-2 can be used “as is” to make viral proteins without prior modification. SARS-CoV-2 has one of the largest RNA genomes among RNA viruses, made up of a replicase gene encoding non-structural proteins (nsps), as well as various structural and accessory genes. During viral replication, depending on the starting point (a.k.a. a ribosomal frame shift), the replicase gene can produce one of two poly-protein chains, which are then cleaved to produce 15-16 individual viral nsps (non-structural Proteins). These nsps then form a large membrane-bound replicase complex with multiple enzymatic activities, like a tiny viral Voltron.
What’s in a Name?
This blog post will focus on SARS-CoV-2 Nsp15, a nidoviral RNA uridylate‐specific endoribonuclease (NendoU). That is a very long and complicated name which conveys a lot of information, so let’s break it down into its individual parts, like when Voltron separates to become several small robots. It’s possible I’ve watched too many cartoons during lockdown:
- Nidoviral – An order of RNA viruses which infect vertebrates and invertebrates.
- RNA – Genetic material used to produce proteins
- Uridylate-specific – Cuts Uridine (U) in RNA, not Cytosine (C), Adenine (A) or Guanine (G)
- Endo – A Greek word meaning inside or within
- Ribonuclease – An enzyme that cuts RNA into smaller pieces.
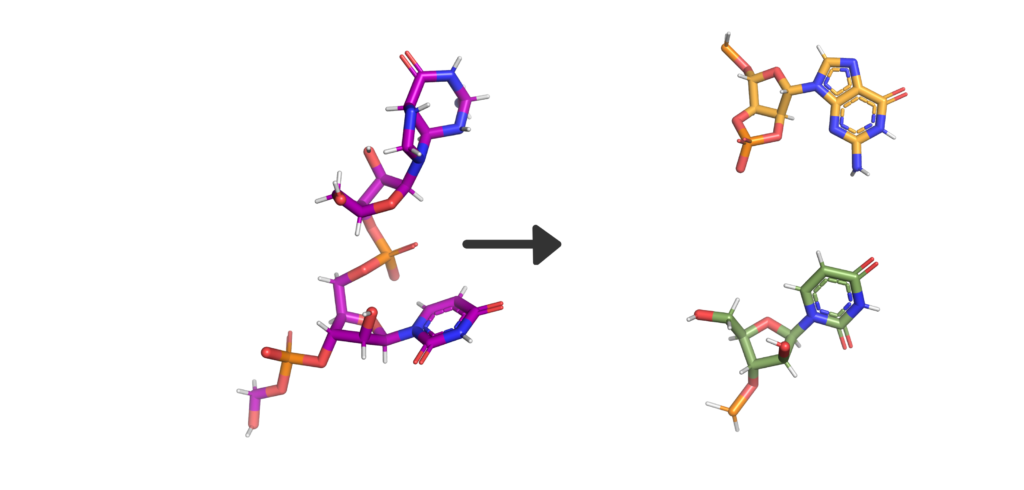
So, what’s in a name? Well, Nsp15 is a viral enzyme that likes to cut at uridine (a building block of RNA) in the middle of an RNA sequence. Quite a lot really. The final bit of the name “NendoU” goes into even more specifics on our protein, as it defines a common family of proteins which share certain traits. The first is that when Nsp15 cuts RNA, it gives a 2′‐3′ cyclic phosphodiester and 5′‐hydroxyl terminus. If we look at Figure 2, you’ll see a purple RNA chain made of two bases linked by an orange phosphate in the middle. When RNA is cleaved by Nsp15, a 2′‐3′ cyclic phosphodiester is made: in the two resulting molecules, the phosphate ion has been incorporated into a 5-membered ring (orange), and the other half of the RNA has a 5′‐hydroxyl, or and OH- group on another 5-membered ring (green). The second thing being a member of the NendoU family tells us is that the catalytic domain of the protein (the business end) is found on the C-terminal end of the protein (the latter half) as this is a shared trait within the NendoU family.
Domains
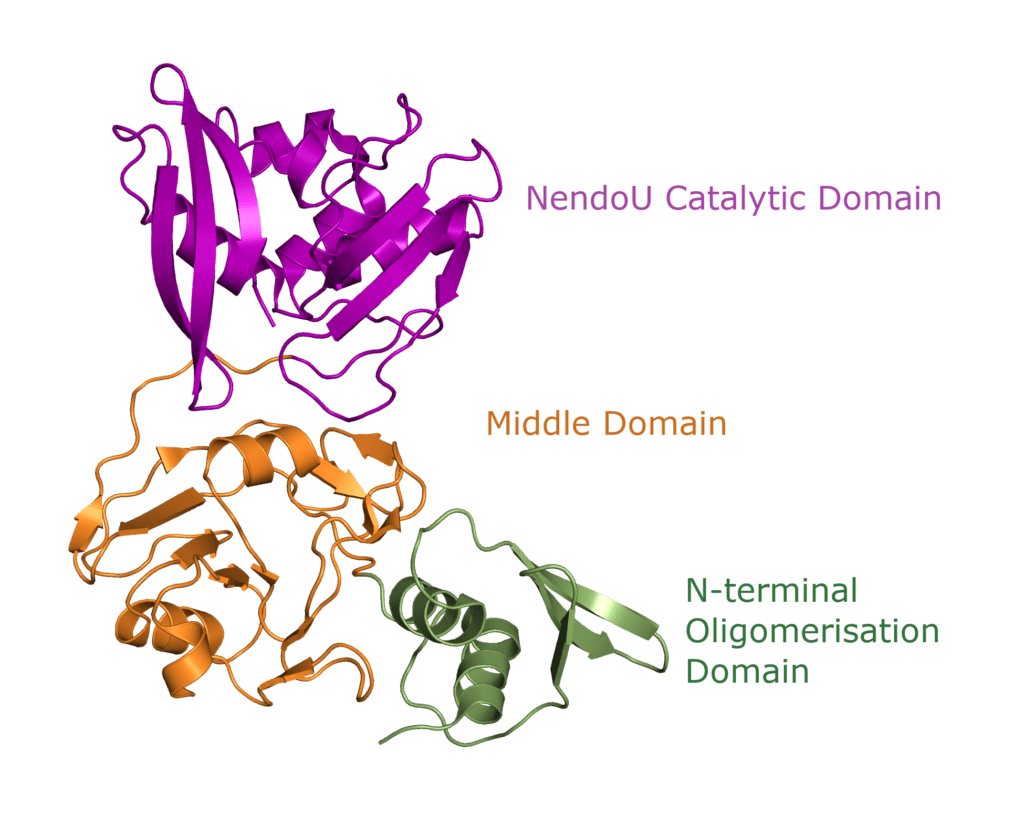
One Nsp15 monomer is made up of three distinct domains, the aforementioned N-terminal oligomerisation domain (green), a middle domain in… well, the middle (orange), and the catalytic NendoU domain at the C-terminal (purple, Figure 3b). Overall SARS-CoV-2 Nsp15 shows high sequence identity with SARS-CoV Nsp15 (88%) and, somewhat lower identity with MERS-CoV (51%) (Youngchang 2020), but the overall structural similarity is very high between the three viruses. For a more detailed breakdown of the secondary structure that makes up individual Nsp15 domains, check out our proteopedia entry!
Tertiary Structure
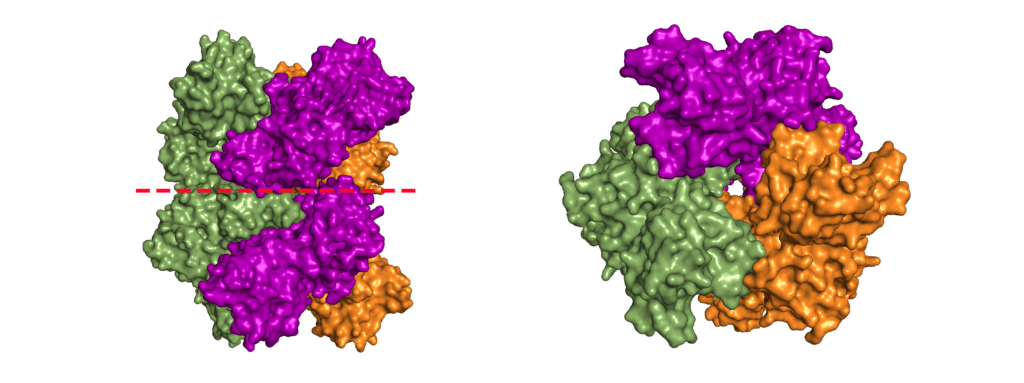
Nsp15 forms a double-ring hexamer made up of a dimer of trimers stabilised by an N-terminal oligomerisation domain. So, three monomers form a trimer which then binds another trimer of monomers. However, If you open a crystal structures this can be confusing as you might not be presented with the whole complex. A crystal is composed of an infinite array of identical (or near enough) molecules related to each other by symmetry. To eliminate the need to store an infinite number of atoms on your computer the PDB file gives you just enough of the crystal to define the unique part. You are then expected to remember that the rest are generated by symmetry. This subset is called the asymmetric unit. Should you want to try and generate the whole crystal you can try, but your computer will likely grind to a halt on its way to infinity (and beyond).
For most structures the asymmetric unit is the interesting part. Often, when the biologically relevant complex has symmetry itself, like Nsp15 does, only part of the complex will be present in the file from the PDB. In the case of the PDB model 6X4I the molecules of each trimer obey the crystal’s three-fold symmetry. The file you download contains two molecules, one monomer from each trimer, and you must generate the symmetry related molecules (shown in green and orange in figure 3) to build the entire complex. These six monomers all come together to form the active enzyme, a 100 Å long and 10-15 Å wide channel, open to solvent from the top, bottom, and three separate side openings in the middle of the hexamer (Figure 4). Formation of the hexamer has been shown to be essential for enzymatic activity, making the oligomerisation interfaces a potential target for structure-based drug design. I’m not sure if I should be proud or disappointed that I didn’t mention Voltron once back there.
The Active Site
SARS-CoV-2 Nsp15 is a Mn2+ dependent endoribonuclease, meaning it relies on the coordination of manganese to perform the transesterification reaction (cutting RNA). Unfortunately, the structure of SARS-CoV-2 Nsp15 has not been solved with manganese present, but we do have a structure with 3’ uridine monophosphate in the active site (PDBID: 6X4I). It has been proposed that the presence of manganese help stabilise the active site and substrate, but it is yet to been seen. Based on sequence alignment against related enzymes from other viruses we know the active site is made up of six conserved residues that sit in a shallow groove between two β-sheets (His235, His250, Lys290, Thr341, Tyr343, and Ser294), as shown in Figure 5. His235, His250, and Lys290 are predicted to act as a catalytic triad, His235 as a general acid, and His250 as a base with Lys290 governing U specificity.
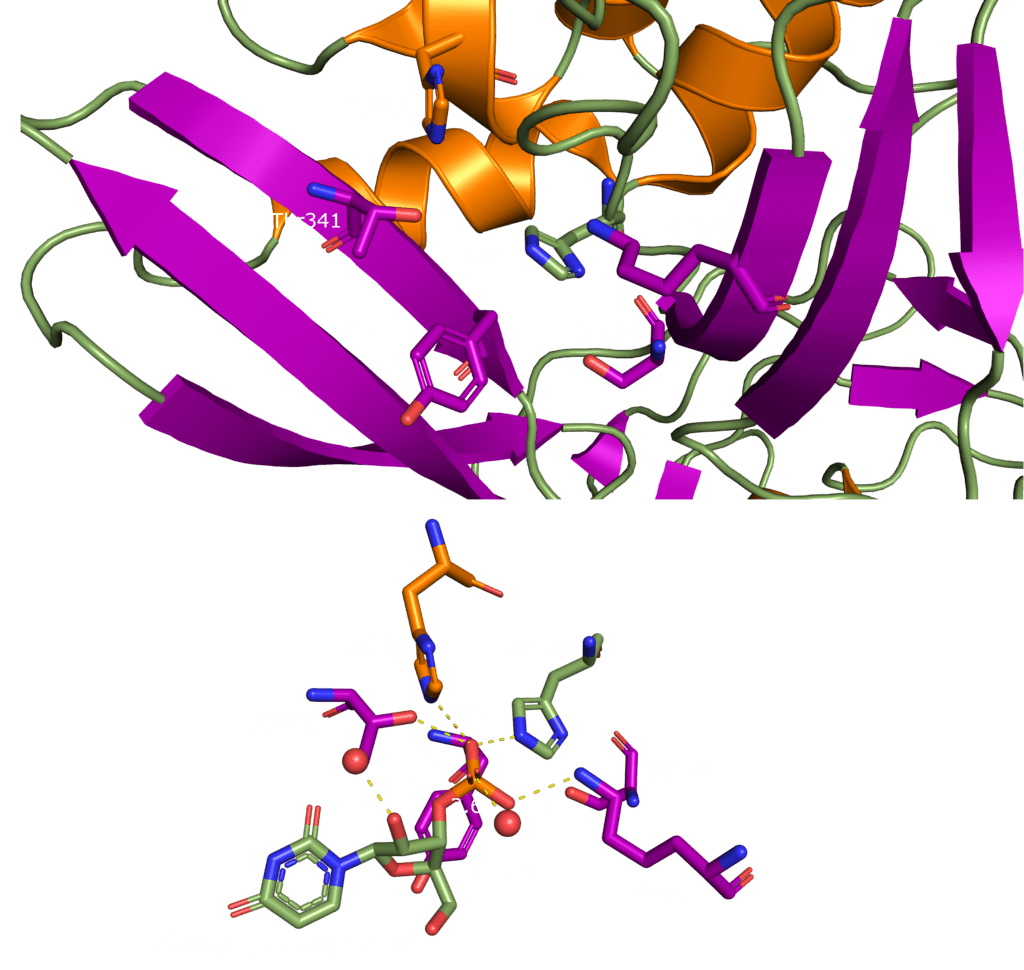
But What Does it do?
After all that we have a pretty clear picture of what Nsp15 NendoU looks like, but what does it actually do? The fact that it cuts RNA would immediately suggest a role in viral replication, but Nsp15 deficient coronaviruses are still able to replicate. So maybe not, at least it's not essential for replication. Another suggestion is that Nsp15 is involved in interfering with the hosts innate immune response, but other studies suggest this is independent of Nsp15 activity. Finally, it has been suggested that Nsp15 degrades viral RNA as a means of hiding viral infection from the host immune system. So why does coronavirus bother with Nsp15? I’m afraid we don’t exactly know yet, but we’re working on it.
With that I’m going to leave you with one final Voltron reference for making it to the end. Good job, you earned this.

Alexander Matthew Payne und Binisha Karki
Einleitung
Haben Sie schon gehört, dass das Corona-Virus „mutiert“? Oder dass weltweit „mehr als ein Stamm“ dieses Virus existiert? Das klingt ziemlich erschreckend, nicht wahr? In Wirklichkeit mutiert aber wirklich alles - alle Organismen, angefangen bei Bakterien bis hin zum Menschen, verändern im Laufe der Zeit ihr Erbgut. Dies kann auch passieren, wenn die DNA (Deoxyribonucleic Acid, zu Deutsch: Desoxyribonukleinsäure) UV-Licht (wie dem der Sonne!) ausgesetzt wird oder während der Replikation der DNA. Bei diesem Vorgang verwendet eine Zelle einen der beiden DNA-Stränge als Vorlage für eine neue, komplementäre Kopie des anderen Strangs. Mutationen treten bei allen Lebewesen (und Viren) auf und sind die Triebkraft der Evolution. In diesem Beitrag befassen wir uns mit der Replikation von Coronaviren unter besonderer Berücksichtigung der beteiligten Proteine.
Wie vermehrt sich das Coronavirus?
SARS-CoV-2 verwendet zur Kodierung seines Genoms nicht DNA, sondern einsträngige Ribonukleinsäure (Ribonucleic Acid, RNA) und wird deshalb der Klasse der „einsträngigen RNA-Viren“ zugeordnet. Aus diesem Grund muss das Virus zur Vervielfältigung seines Genoms einen anderen Weg beschreiten als „normale“ Zellen: Das für die Replikation der RNA zuständige Virusprotein wird als „RNA-abhängige RNA-Polymerase“ (RdRp) bezeichnet. Dieses Protein verwendet die virale RNA als Vorlage für neue Virus-RNA-Kopien; dabei reiht es einzelne Ribonukleotide wie Perlen auf einer Schnur aneinander. Dieser Vorgang wird als Polymerisation bezeichnet.
Eine Studie des AG Mors an der Texas A&M University hat gezeigt, dass die SARS-CoV-2 RNA-Polymerase eine bemerkenswerte Ähnlichkeit mit der RNA-Polymerase von SARS-CoV (> 95 %) und MERS-CoV [1] hat, dem Virus, das das Middle East Respiratory Syndrome verursacht. Wir könnten also vielleicht von den Erkenntnissen aus der SARS- und MERS-Epidemien bei der Bekämpfung von SARS-CoV-2 profitieren. Mit kontinuierlichen Mitteln für die Coronavirus-Forschung in den letzten Jahren hätten wir vielleicht noch mehr über RdRp lernen können. In jedem Fall könnte RNA-Polymerase ein interessanter Ansatzpunkt für Medikamente zur Eindämmung von COVID-19 und zur Senkung der Sterblichkeitsrate sein…
Struktur der RNA-abhängigen RNA-Polymerase
Um ein Medikament so zu optimieren, dass es an dieses Protein bindet und es blockiert, muss man zunächst die Struktur von RdRp bestimmen und seine Funktionsweise umfassend verstehen – und in den vergangenen Monaten wurden hierzu verschiedene Strukturen der SARS-CoV-2 RNA-Polymerase aufgeklärt.
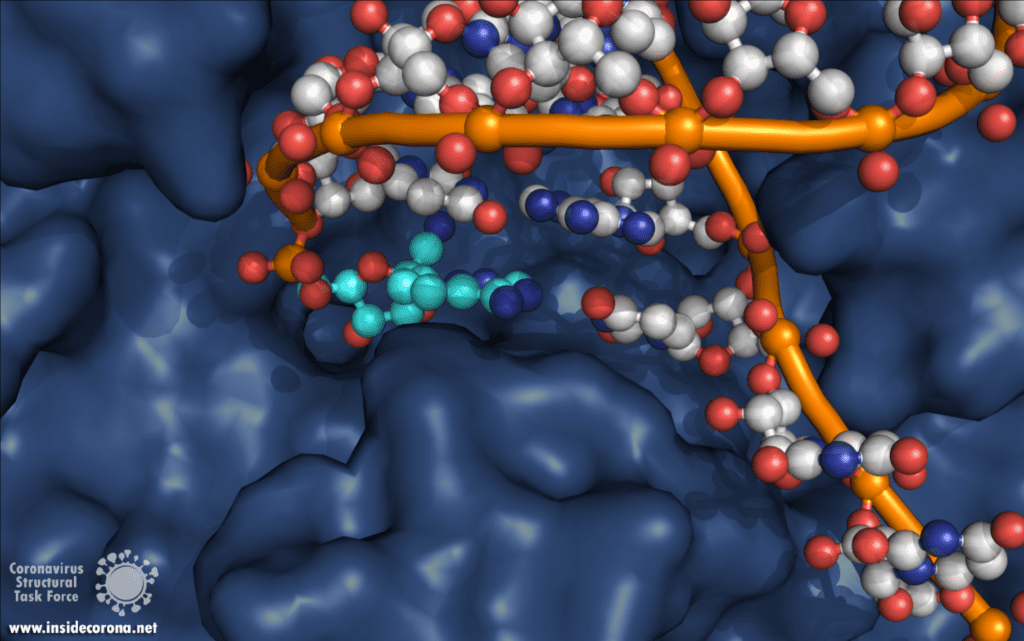
Eine interessante Struktur zeigt die RNA-Polymerase in Aktion: dabei ist zu erkennen, wie ein RNA-Strang verlängert wird (siehe Abbildung 1). Es ist deutlich zu sehen, dass die Polymerase einen Komplex mit kleineren Proteinen, den so genannten Nichtstrukturproteinen 7 und 8 (nsp7 und nsp8), bildet. Diese Proteine stabilisieren die Bindung der RNA-Polymerase an die RNA-Vorlage und erhöhen die „Prozessivität“, also die Zeit, die das Enzym an der Vorlage kleben bleibt, bis es sich wieder ablöst. [3]
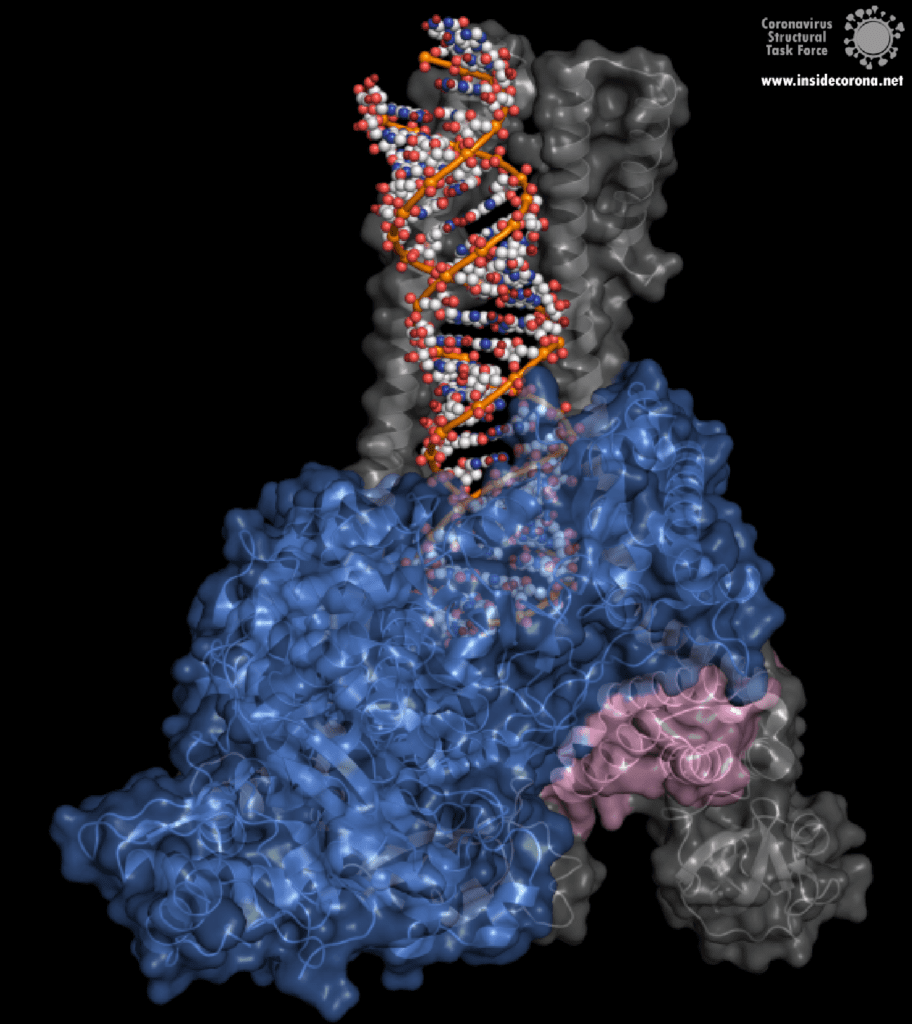
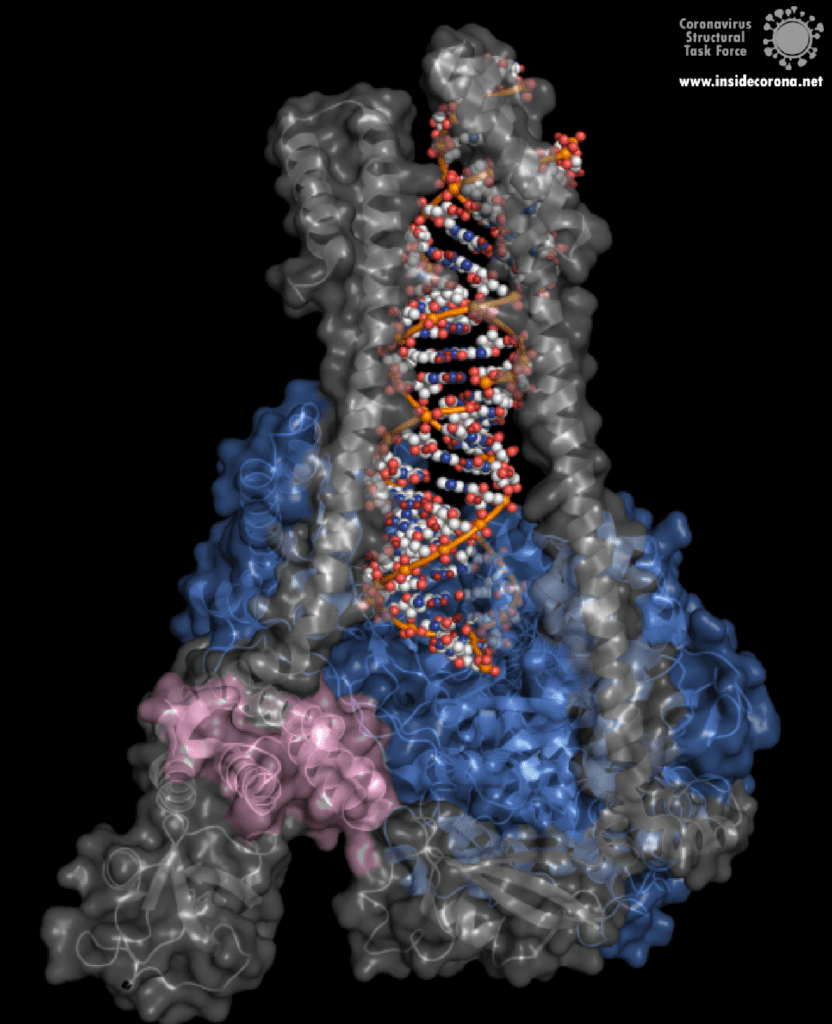
In der Mitte des Proteins befindet sich das „aktive Zentrum“, in dem der eigentliche Synthesevorgang stattfindet. Die Aminosäuren der Polymerase im aktiven Zentrum ermöglichen eine besonders hohe Polymerisationsrate. Die Polymerase kann bis zu 100 Nukleotide pro Sekunde aneinanderreihen [3]. RNA-Moleküle, die für die Produktion der RNA-Kette benötigt werden, gelangen durch ein Fenster in das aktive Zentrum. An dieser Stelle können nun antivirale Medikamente ansetzen:
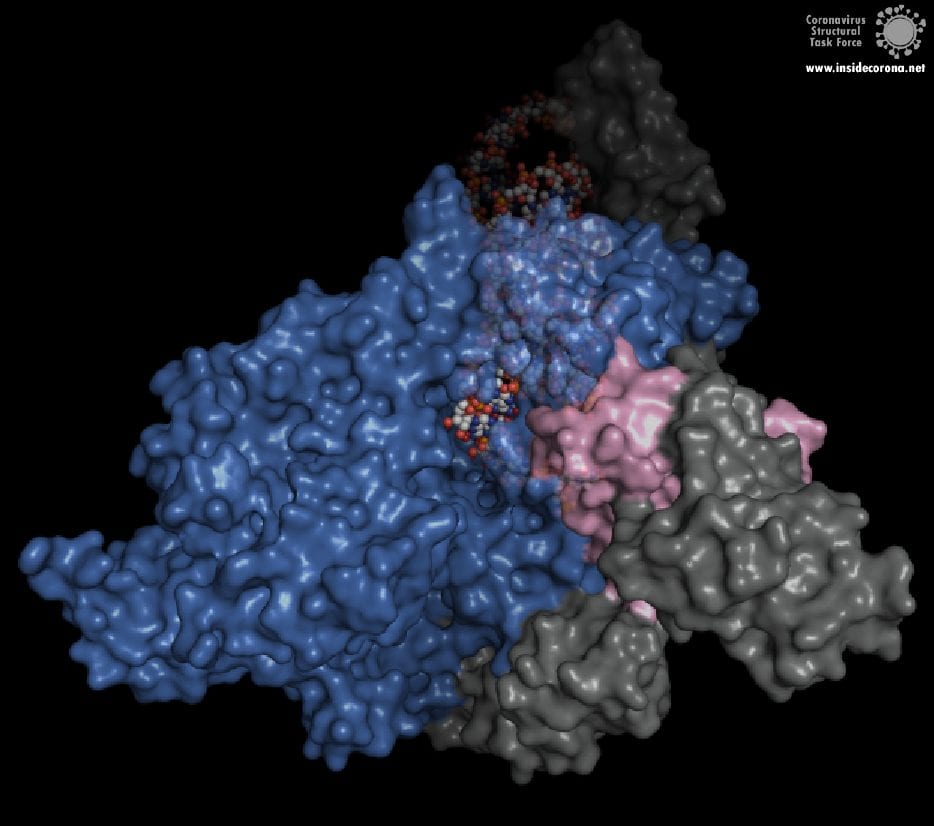
Wie können antivirale Medikamente RNA-abhängige RNA-Polymerase hemmen?
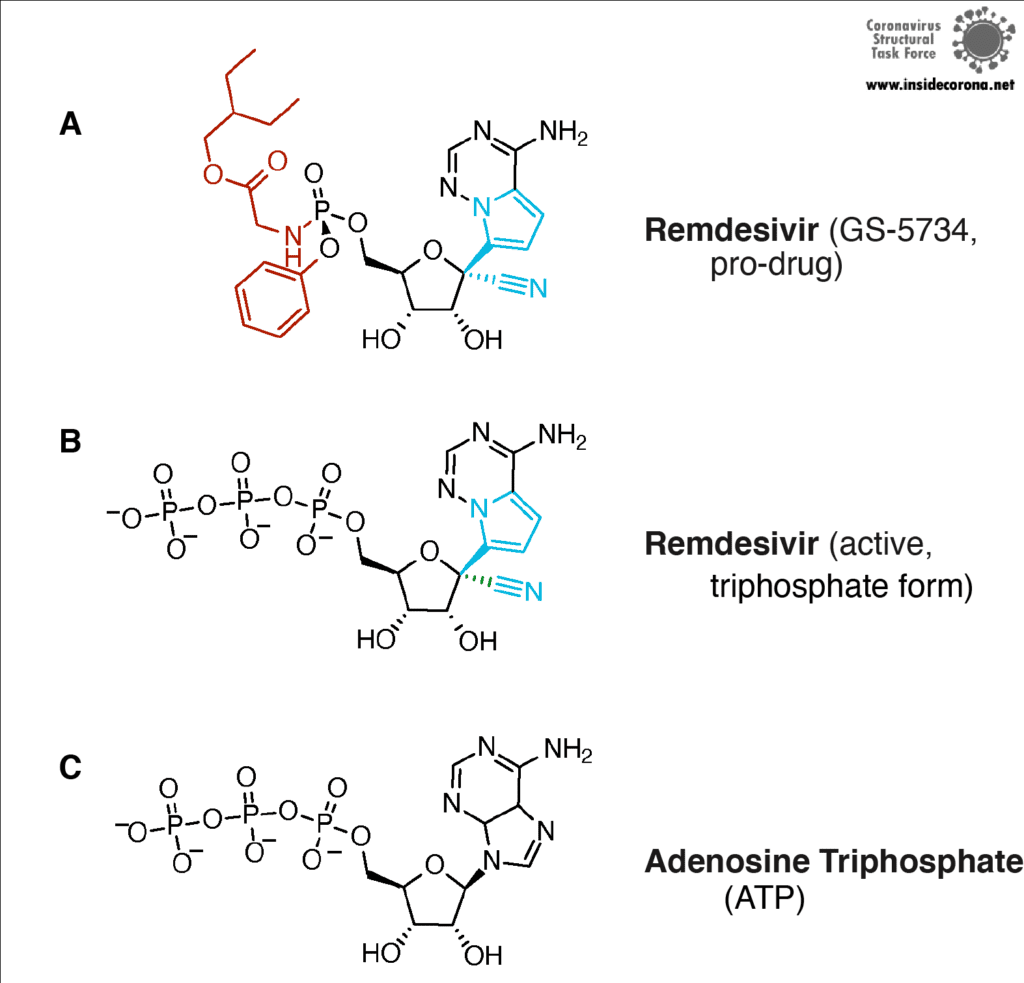
Remdesivir ist das von der FDA zugelassene Medikament des Herstellers Gilead, welches im Fokus der Suche nach einem Heilmittel gegen COVID-19 steht. Remdesivir (das mit dem Entwicklungscode GS-5734 unter dem Markennamen Veklury vertrieben wird) ist ein „Nukleotid-Analogon“, also eine synthetisch hergestellte Substanz, die in Form und Chemie den Bausteinen von RNA und DNA (Nukleotiden) ähnelt (siehe Abbildung 4).
Remdesivir wurde ursprünglich als generelles antivirales Medikamente entwickelt und zeigte in Labortests an Zellen und in klinischen Tests an Affen seine Wirksamkeit gegen das Ebolavirus [4]. Doch ist die Forschung dahingehend noch sehr jung und die Mühlen der Wissenschaft mahlen so langsam, dass bis zur COVID-19-Pandemie keine groß angelegten klinischen Studien mit Remdesivir durchgeführt wurden. Wissenschaftler und Ärzte wollten das Medikament schnell gegen COVID-19 einsetzen, um zu sehen, ob es auch hier half. Sowohl die USA als auch Japan erteilten dem Medikament bereits im Mai eine „Zulassung für die Anwendung in Notfallsituationen“ für Patienten mit schwerem COVID-19-Verlauf [5], [6].Woraufhin Remdesivir im Juli von der Europäischen Arzneimittelbehörde eine „bedingte Zulassung“ erhielt (welche für Medikamente vergeben wird, die eine medizinische Versorgungslücke schließen, aber über nicht genügend Daten für eine normale Zulassung verfügen). Damit kann Remdesivir bis zum nächsten Jahr bei Patienten mit schweren COVID-19-Verläufen eingesetzt werden [7].
Wie ist es denn aber möglich, dass ein Medikament gegen Ebola, Influenza oder eine andere Viruserkrankung auch bei COVID-19 hilft? Ich habe mir diese Frage immer und immer wieder gestellt, als die Studien über die Medikamentenwirksamkeit veröffentlicht wurden – und ich stand damit ganz sicher nicht alleine...
Die simple Antwort lautet: Alle diese Viren müssen ihr RNA-Genom vervielfältigen und verwenden hierzu das im Wesentlichen gleiche Werkzeug, nämlich eine RNA-abhängige RNA-Polymerase. Und alle Medikamente aus der Gruppe der Nukleotid-Analoga bemühen den gleichen Trick: sie geben sich als Ribonukleotide (also die eingangs beschriebenen „Perlen“) aus und gelangen so in das aktive Zentrum der RNA Polymerase. Im Inneren angekommen, docken sie dauerhaft am aktiven Zentrum an und bringen die Polymerase-Maschine zum Stillstand. Weil dieser Trick bei jeder viralen RNA-Polymerase funktionieren sollte, könnte man diese Wirkstoffe bei allen RNA-Viren als antivirale Medikamente einsetzen. In der Praxis sieht es natürlich etwas anders aus, weil zwischen den verschiedenen RNA-Polymerasen eben doch Unterschiede bestehen. Aber es ist ein guter Ausgangspunkt. Wenn wir über einsatzbereite allgemeine Virostatika gegen SARS-CoV-2 verfügen, sind wir vermutlich besser für einen künftigen Coronavirus-Ausbruch gerüstet!

Abbildung 3. Wahrscheinlich sehen wir alle nur, was wir sehen wollen.
Die Chemie von Remdesivir
In seiner Struktur ähnelt Remdesivir dem Nukleotid Adenosin, doch einige ausgeklügelte chemische Anhänge machen es zu einem besonders raffinierten Medikament (vielen Dank an die medizinische Chemie!). Wird Remdeivir injiziert, gelangt es in unsere Zellen, die es als Fremdkörper erkennen und zu verdauen versuchen. Dabei entfernen die Zellen allerdings nur die chemischen Anhänge. Was übrig bleibt, halten sie fälschlicherweise für ein normales Adenosin. In infizierten Zellen bindet nun die virale RNA-abhängige RNA-Polymerase dieses Moleküle und baut es anstelle von Adenosinmolekülen in den neu synthetisierten RNA-Strang ein. Remdesivir wird Teil der RNA, blockiert die Polymerase und verhindert, dass das Virus weitere Kopien seines Erbguts anfertigen kann. Dadurch kommt die Virusreplikation letztlich zu einem Stillstand und hilft dem Patienten, das Virus zu bekämpfen.
Ein anderes Medikament, das die RNA-Polymerase-Aktivität hemmt, ist Favipiravir, das unter den Markennamen Avigan, Abigan und FabiFlu vertrieben wird. Favipiravir wurde von Toyama Chemical Co. Ltd. in Japan entdeckt und wirkt ähnlich wie Remdesivir, allerdings imitiert es statt eines Adenosin-Nukleotids ein Guanosin- Nukleotid [8]. Dieses Medikament wurde bereits 2014 in Japan für den Einsatz bei resistenten Fällen von Influenza A und B zugelassen, wartet in den USA (erst in der klinischen Phase II und III) und in Großbritannien allerdings noch auf seine Freigabe [9]. Außerdem wird das Medikament in 43 Ländern für den Einsatz getestet. Die Zulassung von Favipiravir als COVID-19-Medikament erfolgte in einigen Ländern sehr rasch. So erteilte China am 15. März 2020 die Freigabe, Russland am 3. Juni 2020 und Indien am 20. Juni 2020 [10], [11]. Andere Länder, wie Japan, befinden sich noch in unterschiedlichen klinischen Teststufen und die Ergebnisse werden bis Ende Juli 2020 erwartet [10].
Heißt das, wir haben ein Heilmittel gegen SARS-CoV-2?
Leider noch nicht. Zwar hat Remdesivir die klinischen Studien mit beispielloser Geschwindigkeit durchlaufen, doch ist noch mehr Arbeit nötig, um sicher sein zu können, dass es unbedenklich und vorallem auch wirksam ist. Insgesamt wurden noch nicht besonders viele Menschen mit Remdesivir behandelt, so dass wir nicht wirklich sagen können, welche Nebenwirkungen es haben kann; zuletzt gab es Hinweise auf eine Schädigung von Leber und Niere [12, 13]. Die häufigsten Nebenwirkungen sind Übelkeit (10% bzw. 9% der Patienten), Verdauungsstörungen (7 %) und erhöhte Transaminasewerte (6 % bzw. 8 %). In einer Studie mussten 3,6 % der Patienten in einer 10-Tage-Studie die Therapie aus letzterem Grund abbrechen. Allerdings können auch schwere Virusinfektionen Leberschäden verursachen. Die Trennung der beiden Ursachen stellt eine große Herausforderung dar. Außerdem ist Remdesivir kein Allheilmittel. In einer Studie verkürzte sich unter Remdesivir die Genesungszeit von 15 auf 11 Tage; bei Patienten mit leichter bis mittelschwerer Erkrankung zeigte es jedoch keine Wirkung und bei Patienten, die bereits beatmet wurden, konnte kein Unterschied in der mittleren Genesungszeit festgestellt werden [14]. Da das Medikament über mehrere Tage als Infusion verabreicht werden muss, gibt es ein ziemlich kleines Zeitfenster, in dem Remdesivir tatsächlich helfen kann.
Wie auch Remdesivir hat Favipiravir seine eigenen Nebenwirkungen. Dazu zählen unter anderem Die Schädigung von Leber und Niere, erhöhte Harnsäurewerte, Hautallergien usw. [15]. Aus diesem Grund soll es bei Patienten mit schwerer Diabetes und schweren Herzerkrankungen nicht eingesetzt werden. Zudem ist es für schwangere Frauen nicht geeignet, da es Missbildungen oder ein Absterben des Fötus verursachen kann. Es hat sich gezeigt, dass Favipiravir nur im Anfangsstadium einer SARS-CoV-2-Infektion wirkt, wenn das körpereigene Immunsystem noch gut funktioniert. Bei schwer erkrankten Patienten kann es einen „Zytokinsturm“ auslösen (bei dem das Immunsystem lebensbedrohlich entgleist). Ein universelles Medikament gegen COVID-19 müsste aber für alle Menschen sicher sein.
Diese Medikamente sind jedoch besser als nichts. Und indem Wissenschaftler die beteiligten Mechanismen verstehen lernen, können sie vorhandene Medikamente zum Nutzen aller weiter verbessern. Während die meisten an der RNA-Polymerase ansetzenden „allgemeinen Virostatika“ bei SARS-CoV-2 versagt haben, war Remdesivir hingegen manchmal erfolgreich. Die Wissenschaft vermutet, dass seine begrenzte Wirksamkeit an einem SARS-CoV-2-eigenen Korrekturprotein namens Exonuklease liegt. Sobald die RNA-Polymerase neue RNA gebildet hat, prüft die Exonuklease diese auf Richtigkeit. Eine Studie hat gezeigt, dass ein anderer RNA-simulierender Arzneistoff namens Ribivarin unmittelbar nach der RNA-Synthetisierung erkannt und anschließend durch die Exonuklease wieder aus der RNA entfernt wird. Die Reparaturmechanismen scheinen für Remdesivir jedoch nicht gut zu funktionieren, was erklärt, warum es erfolgreicher ist als andere Medikamente [17], [18].
Wie nsp14 die Integrität und Virulenz von SARS-CoV-2 aufrechterhält, lesen Sie in einem zukünftigen Blog-Eintrag!

Abbildung 6. Haben wir das nicht alle schon erlebt?
Empfohlene Strukturen
Wer die Strukturen eingehender zu studieren möchte, findet diese in unserem GitHub-Repository, zusammen mit Informationen zur Validierung und, soweit vorhanden, zu verbesserten Strukturen. Für einen hochauflösenden Vergleich des aktiven Zentrums mit und ohne Remdesivir wurden 7BV2 bzw. 7BV1 zusammen bei 2,5 bzw. 2,8 Å Auflösung veröffentlicht. In der wachsenden Struktur des oben gezeigten Komplexes (6YYT) sind sowohl die Polymerase als auch die Kofaktoren und die RNA mit wenig „fehlender“ Dichte und einer Auflösung von 2,9 Å sehr gut aufgelöst. Sie ist wahrscheinlich 6M71 und 7BTF vorzuziehen, die mit einer ähnlichen Auflösung veröffentlicht wurden, wobei allerdings der Komplex weniger detailliert ist und keine RNA vorliegt. Für Interessierte: 7C2K und 7BZF (bei 2,93 Å und 3,26 Å) zeigen den an die RNA gebundenen Komplex vor und nach der Translokation.
Quellen
[1] J. S. Morse, T. Lalonde, S. Xu, and W. R. Liu, “Learning from the Past: Possible Urgent Prevention and Treatment Options for Severe Acute Respiratory Infections Caused by 2019-nCoV,” ChemBioChem, vol. 21, no. 5, pp. 730–738, Mar. 2020, doi: 10.1002/cbic.202000047.
[2] H. S. Hillen, G. Kokic, L. Farnung, C. Dienemann, D. Tegunov, and P. Cramer, “Structure of replicating SARS-CoV-2 polymerase,” Nature, May 2020, doi: 10.1038/s41586-020-2368-8.
[3] W. Yin et al., “Structural basis for inhibition of the RNA-dependent RNA polymerase from SARS-CoV-2 by remdesivir,” Science, p. eabc1560, May 2020, doi: 10.1126/science.abc1560.
[4] R. T. Eastman et al., “Remdesivir: A Review of Its Discovery and Development Leading to Emergency Use Authorization for Treatment of COVID-19,” ACS Cent. Sci., May 2020, doi: 10.1021/acscentsci.0c00489.
[5] O. of the Commissioner, “Coronavirus (COVID-19) Update: FDA Issues Emergency Use Authorization for Potential COVID-19 Treatment,” FDA, May 04, 2020. https://www.fda.gov/news-events/press-announcements/coronavirus-covid-19-update-fda-issues-emergency-use-authorization-potential-covid-19-treatment (accessed Jul. 08, 2020).
[6] A. Sternlicht, “Japan Approves Remdesivir For Use On Severe COVID-19 Patients,” Forbes. https://www.forbes.com/sites/alexandrasternlicht/2020/05/07/japan-approves-remdesivir-for-use-on-severe-covid-19-patients/ (accessed Jul. 08, 2020).
[7] D. CZARSKA-THORLEY, “First COVID-19 treatment recommended for EU authorisation,” European Medicines Agency, Jun. 25, 2020. https://www.ema.europa.eu/en/news/first-covid-19-treatment-recommended-eu-authorisation (accessed Jul. 10, 2020).
[8] E. De Clercq, “New Nucleoside Analogues for the Treatment of Hemorrhagic Fever Virus Infections,” Chem. Asian J., vol. 14, no. 22, pp. 3962–3968, Nov. 2019, doi: 10.1002/asia.201900841.
[9] K. Shiraki and T. Daikoku, “Favipiravir, an anti-influenza drug against life-threatening RNA virus infections,” Pharmacol. Ther., vol. 209, p. 107512, May 2020, doi: 10.1016/j.pharmthera.2020.107512.
[10] T. Hornyak, “Japan sending Fujifilm’s flu drug favipiravir to over 40 countries for Covid-19 trials,” CNBC, May 04, 2020. https://www.cnbc.com/2020/05/04/fujifilms-flu-drug-favipiravir-sent-to-43-nations-for-covid-19-trials.html (accessed Jul. 14, 2020).
[11] G. P. Ltd, “Glenmark Becomes the First Pharmaceutical Company in India to Receive Regulatory Approval for Oral Antiviral Favipiravir, for the Treatment of Mild to Moderate COVID-19.” https://www.prnewswire.com/in/news-releases/glenmark-becomes-the-first-pharmaceutical-company-in-india-to-receive-regulatory-approval-for-oral-antiviral-favipiravir-for-the-treatment-of-mild-to-moderate-covid-19-855346546.html (accessed Jul. 14, 2020).
[12] Goldman, J. D. et al. Remdesivir for 5 or 10 Days in Patients with Severe Covid-19. N. Engl. J. Med. (2020) doi:10.1056/NEJMoa2015301
[13] Remdesivir Safety Forecast: Watch the Liver, Kidneys | MedPage Today. https://www.medpagetoday.com/infectiousdisease/covid19/86582
[14] J. H. Beigel et al., “Remdesivir for the Treatment of Covid-19 — Preliminary Report,” N. Engl. J. Med., vol. 0, no. 0, p. null, May 2020, doi: 10.1056/NEJMoa2007764.
[15] Sandhya Ramesh, “Favipiravir, Japanese drug that’s the new Covid treatment hope your chemist will soon stock,” ThePrint, Jun. 25, 2020. https://theprint.in/health/favipiravir-japanese-drug-thats-the-new-covid-treatment-hope-your-chemist-will-soon-stock/447987/ (accessed Jul. 14, 2020).
[16] F. Ferron et al., “Structural and molecular basis of mismatch correction and ribavirin excision from coronavirus RNA,” Proc. Natl. Acad. Sci., vol. 115, no. 2, pp. E162–E171, Jan. 2018, doi: 10.1073/pnas.1718806115.
[17] C. J. Gordon, E. P. Tchesnokov, J. Y. Feng, D. P. Porter, and M. Gotte, “The antiviral compound remdesivir potently inhibits RNA-dependent RNA polymerase from Middle East respiratory syndrome coronavirus,” J. Biol. Chem., Feb. 2020, doi: 10.1074/jbc.AC120.013056.
[18] L. Zhang et al., “Role of 1’-Ribose Cyano Substitution for Remdesivir to Effectively Inhibit both Nucleotide Addition and Proofreading in SARS-CoV-2 Viral RNA Replication,” bioRxiv, p. 2020.04.27.063859, Apr. 2020, doi: 10.1101/2020.04.27.063859.
Das Coronavirus unkompliziert selbst drucken und zusammenbauen – wir haben ein 3D-Modell dafür entworfen!
Abhängig vom User und dem jeweiligen 3D-Drucker sind die Details natürlich unterschiedlich. Die Methoden, die wir angewandt habenkönnen aber als Anhaltspunkt dienen. Nutzer ohne eigenen 3D-Drucker können die STL-Daten aber auch dafür verwenden, den Druck bei einem externen Dienstleister zu beauftragen. Wir hoffen, mit diesem Projekt nicht nur private Nutzer zu erreichen, sondern auch bessere Möglichkeiten für die Lehre und das öffentliche Verständis des Virus zu schaffen.
Unser Entwurf basiert auf aktuellesten wissenschaftlichen Erkenntnissen bezüglich der Proteinstrukutur und Größenverhältnisse. Mehr dazu hier.
Mit dem ausgedruckten und zusammengebauten Modell bekommt man eine Vorstellung, wie das Virion aussehen würde, wenn es um eine Million vergrößert wäre (1 mm des Models stellt 1 nm (10 Å) dar). Die RNA, das Erbgut des Virus, wäre dann zehn Meter lang und einen Millimeter dick.
Zusätzlich haben wir ein Modell eines menschlichen Antikörpers im selben Maßstab entworfen, welches zusätzlich zu den Strukuren des Virions gedruckt und je nach Wunsch an das Spike-Protein angehägt werden kann. Um das Drucken, Bemalen und Zusammenbauen zu erleichtern, haben wir die Virusstruktur in vier einzelne Komponenten zerlegt:

Bis jetzt wurden die Strukturen erfolgreich auf verschiedenen Schmelzschicht-Druckern (FDM), einem Rostok MAX v2 und einem Prusa I3 MK3 Drucker getestet. Mit anderen Methoden, wie Stereolithographie, wäre eine noch höhere Qualität durchaus möglich.
Jeder Teil ist im STL-Format verfügbar und sollte mit jeder geeigneten Slicer-Software druckbar sein.
Beim Zusammenbauen und Bemalen des fertigen Drucks geht man am besten nach eigenem Gutdünken vor. Die exakten Details unterschieden sich schließlich je nach Equipment und nach den Einstellungen.
Wir zeigen hier trotzdem unseren Aufbau in knapper Zusammenfassung.
Druck der Komponenten:
Der erste Schritt ist das Drucken der einzelnen Bestandteile. Die Virion-Kugel ist schnell gedruckt, da durch die flache Oberfläche keine weiteren Träger oder Verbindungen benötigt werden.
Dieser Teil kann mit einem Minimum an Füllung und Trägern gedruckt werden, aus Gründen der Stabilität empfehlen wir jedoch eine Füllung von mindestens 10%.
Die anderen Teile (Spikeproteine und Antikörper) stellen hierbei eine größere Herausforderung dar.
Das Spikeprotein muss für das fertige Model mindestens 95mal gedruckt werden. Hierzu können entweder individuelle Einstellungen genutzt oder die 25x STL-Datei 4mal gedruckt werden.
Es ist empfehlenswert das Spike-Protein mit dem Kopf in Richtung Druckbett zu drucken. Das erhöht die Stabilität und benötigt weniger Verbindungen und Vernetzungen zwischen den einzelnen Trägern.
Diese müssten sonst mit Fingerspritzengefühl vom sensiblen Stamm der Spikes entfernt werden. Wie viele Träger zusätzlich hinzugefügt werden, kann je nach Nutzer und der jeweiligen Situation entschieden werden.
Ein Dual-Extruder-Drucker ist für das Herstellen der Spikes ideal, da die stabilisierenden Verbindungen zwischen den Spikes aus einem wasserlöslichen Plastik gedruckt und somit einfach zu entfernen sind. Auf jeden Fall erzeugt ein individueller Druck der Spikes oder zumindest eine geringere Anzahl pro Block ein besseres Ergebnis. Die Verarbeitung dieser Spikes ist dann einfacher, auch wenn der Druck zeitaufwändiger wird. Generell muss ein guter Kompromiss zwischen der Druckgenauigkeit, der Geschwindigkeit und dem Aufwand beim Aufarbeiten der Modelle gefunden werden.
Die Details dieses Prozesses hängen vor allem von der Art des Druckers, dem Aufbau und der Drucktechnik ab. Wir nutzten die bekannteste Technik: Schmelzschicht-Druck (FDM), als Plastik wurde Polylactide (PLA) verwendet, was die folgende Aufreinigung erleichterte.
Aufarbeitung
Um die Objekte möglichst sauber zusammensetzen und bemalen zu können, ist eine Aufarbeitung der Einzelteile notwendig. Die Stabilisierungsstücke können mit einer Zange entfernt werden, während kleinere Artefakte einen Abschliff benötigen. Auch ein Zahnstocher hat sich als hilfreich erwiesen.

Für PLA erwies sich Ethylacetat als die beste Reinigungsmethode um Oberflächen zu glätten und Überbleibsel der Träger zu entfernen. Das Ethylacetat löst das Plastik auf und zerstört somit kleine Unebenheiten auf den Oberflächen, wenn es bedacht angewendet wird. Hierbei kann unterschiedlich vorgegangen werden, wobei die schonendste Methode das Aussetzen in eine Ethylacetat-Dampf Umgebung in einem geschlossenen Gefäß ist. Es entsteht eine glatte Oberfläche mit genauen Details, der Prozess nimmt jedoch oft viele Stunden oder sogar einige Tage in Anspruch.
Die schnellere Methode , die ebenfalls zufriedenstellende Resultate liefert, ist das Eintunken der Objekte in Ethylacetat für 10-30 Sekunden. Anschließend werden sie abgetupft und zum Trocknen ausgelegt. Oft ist ein zweiter Reinigungsgang nötig. Für die größeren Virusteile kann es helfen ein Tuch, welches mit Ethylacetat getränkt ist, bis zum gewünschten Ergebnis über die Oberfläche zu reiben. Mit dieser Methode lassen sich die beiden Virionhälften auch hervorragend zusammenkleben. Eine kleine Menge Ethylacetat wird auf jeder Fläche der Hälften verteilt und die Hälften zusammengedrückt, bis sie zu einem einzigen Stück verschmolzen sind. Auch die Naht kann dann mit einem Ethylacetat-Tuch gut geglättet werden. Hierfür stellt Aceton-freier Nagellackentferner eine ausgezeichnete, frei käufliche Alternative dar, die die gleichen Ergebnisse erzielen dürfte. Bei der Handhabung dieser Chemikalien sollte immer geeignete Schutzausrüstung getragen werden ( Schutzbrille, Handschuhe etc.).

Übrigens: Aceton erzielt für das andere häufig genutzte Druckmaterial, Acrylnitril-Butadien-Styrol (ABS) die gleiche Wirkung wie Ethylacetat für PLA.
Bemalen und Zusammensetzen
Wie beim Druck, sind auch das Bemalen und die jeweiligen Malmethoden dem Nutzer individuell überlassen. Hier zeigen wir die Variante des Würzburger Modells, bei der wir versucht haben, der Illustration von Thomas Splettstösser möglichst treu zu bleiben.

Die Einzelteile wurden zu Beginn mit einem Primer überzogen, um die Farbe besser an das Modell zu binden. Außerdem wirkt dieser wie eine gleichmäßige Grundierung. Beim Auftragen des Primers und der Nutzung einer Airbrush muss auf die Sicherheit geachtet werden, um das Einatmen der schädlichen Substanzen zu vermeiden. Ein gut belüfteter Raum, ein Abzug und eine Zirkulation weg vom Körper sind zu empfehlen. Das Tragen von Handschuhen, einer Schutzbrille und einer Maske sollte für zusätzlichen Schutz sorgen.
In unserem Fall wurde das Modell hauptsächlich mit einer Airbrush bemalt und wir empfehlen diese Methode für die kleinen Oberflächendetails und komplexen Strukturen. Natürlich können auch alle Teile mit dem Pinsel angemalt werden, dies ist jedoch deutlich zeitaufwendiger und erfordert genaueres Arbeiten. Alle genutzen Farben, Verdünner, Primer und Lack sind von Citadel-Painting. Hier eine Liste der genutzten Farben und Amterisleien die für unser Modell verwendet wurden:
- Grün: “Moot green”
- Gelb: “Yriel Yellow”
- Grau: “Dawnstone”
- Braun: “Baneblade Brown”
- Dunkelbraun: “Doombull Brown”
- Hellblau (Aqua): “Gauss Blaster Green”
- Türkis: “Kabalite Green”

Um den Effekt einer natürlichen Lichtquelle zu erzeugen wurden die Spikes in vier Gruppen unterteilt und unterschiedlich hell bemalt.
Wenn das Modell nicht für die feste Ausstellung auf einer Halterung oder Ähnlichem geplant ist, ist dieser Schritt nicht notwendig. Jedes Spike-Protein wurde mit einem helleren Limettengrün hervorgehoben (Highlighting), um einen stärkeren Kontrast zu erzeugen und die Oberfläche besser zu differenzieren. Anschließend wurde das Highlighting mit einem "Dry-brush" der hellblauen (Aqua) Farbe vollendet.

Nachdem das Virusmodell samt Spikes bemalt war, wurde die Farbe mit Glanzlack und einem matten Finish versiegelt. Dieser Schritt ist ebenfalls optional, aber zum Schutz gegen Abnutzung der Farben bei häufiger Handhabung des Modells zu empfehlen.
Nach all diesen Schritten kommt es endlich zum langersehnten Zusammensetzen der Einzelteile. Falls die Spike Proteine verschiedene Highlights bekommen haben, ist darauf zu achten, sich auf eine „Lichtquelle“ festzulegen und die Spikes dementsprechend anzuordnen und am Modell zu befestigen (Auf einem Ständer: Unten dunkler, nach oben heller). Um die Spikes an ihrer Position zu befestigen haben wir normalen Modellbaukleber verwendet. Starker Bastel-Kleber oder Ethylacetat können hierfür ebenfalls benutzt werden, sowie kleine Magnete für besondere Tüftlerinnen und Tüftler. Da unser Modell auf einer Halterung präsentiert werden soll, wurde hierfür ein Loch an der Unterseite des Modells freigelassen, in dem dann die Stange befestigt werden kann.

Hoffentlich hat euch unser kleines Abenteuer gefallen und inspiriert, euch an euer eigenes 3D-Coronamodell zu wagen. Die beschriebenen Arbeitsschritte haben insgesamt etwas mehr als eine Woche in Anspruch genommen. Das Drucken dauert etwa einen Tag. Aufreinigung und Verfeinerung benötigten mehr als zwei Tage und das Bemalen des Modells ein ganzes Wochenende.
Die Dateien sind öffentlich auf Thingiverse verfügbar und das Modell ist lizensiert als Creative Commons BY-NC: Frei Verwendung und Veränderung für nicht-kommerzielle Zwecke und unter Nennung der "Coronavirus Structural Task Force" als Urheber.

Wie bei jedem 3D-Modell, gibt es weit mehr als einen Weg, diese Aufgabe anzugehen und zu vollenden. Wir freuen uns, darauf, Eure Modelle zu sehen und mit Euch über Herangehensweisen und Techniken zu diskutieren - hier in den Kommentaren, auf Thingiverse oder Twitter!
Die Autoren:
Wir möchten hervorheben, dass dieser Artikel eine Zusammenarbeit mehrerer Leute ist:
Dale Tonrud und Thomas Splettstösser haben zusammen die STL Dateien für das 3D Modell erstellt und verfeinert. dale hatte die Idee, ein Modell zu drucken und diese wurde dann von Andrea Thorn aufgegriffen. Thomas und Dale sorgten dann dafür, das Modell möglichst realistisch und gleichzeitig gut für Handhabung und Druck in Einzelteilen zu gestalten. Dale druckte das erste Design des Modells aus.
Matt Reeves war für die Optimierung und den Druck des Würzburger Modells zuständig. Er fand heraus, wie das Modell am besten nachbearbeitet wird und trug zusammen mit dem Rest des Teams zur Verbessung des Modells bei.
Kristopher Nolte arbeitete zusammen mit Ferdinand Kirsten das gedruckte Modell auf und reinigte es. Kristopher war zudem für die filigrane Arbeit des Bemalens und Zusammensetzens des fertigen Virions verantwortlich.
Dieser Artikel ist übersetzt von Ferdinand Kirsten, Pairoh Seeliger und Kristopher Nolte, nach dem originalen Artikel in Englisch von Kristopher Nolte, Dale Tonrud und Matt Reeves.
Für die Bekämpfung der Lungenkrankheit COVID-19 und um eine weitere Ausbreitung der Pandemie zu stoppen, ist die Entwicklung neuer Medikamente essenziell. Ein vielversprechendes Angriffsziel für neue Wirkstoffe gegen den Erreger, SARS-CoV-2, ist die 3-Chymotrypsin-like Protease (3CL-protease), auch bekannt als Hauptprotease oder Mpro.
Damit neue Viren in der Wirtzelle gebildet werden können, muss die ssRNA des Virus in Proteine translatiert werden. Ein Großteil der Virusproteine werden als lange Polyproteine translatiert. Damit funktionale Proteine daraus entstehen können, müssen diese von Proteasen wie der Hauptprotease zerschnitten werden. Die Prozessierung der Polyproteine ppa1a und ppa1ab erfolgt an 11 Stellen in Richtung des C-terminalen Endes (abwärts von Nsp4). Für die Bildung von neuem Viruserbmaterial wird der RNA-abhängige RNA-Polymerasekomlex benötigt, der ein Teil dieser Polyproteine ist (nämlich Nsp7, Nsp8, Nsp12 und Nsp14). Seine Bildung kann durch eine gezielte Inhibition der Hauptprotease gestoppt werden, und damit die RNA-Replikation.
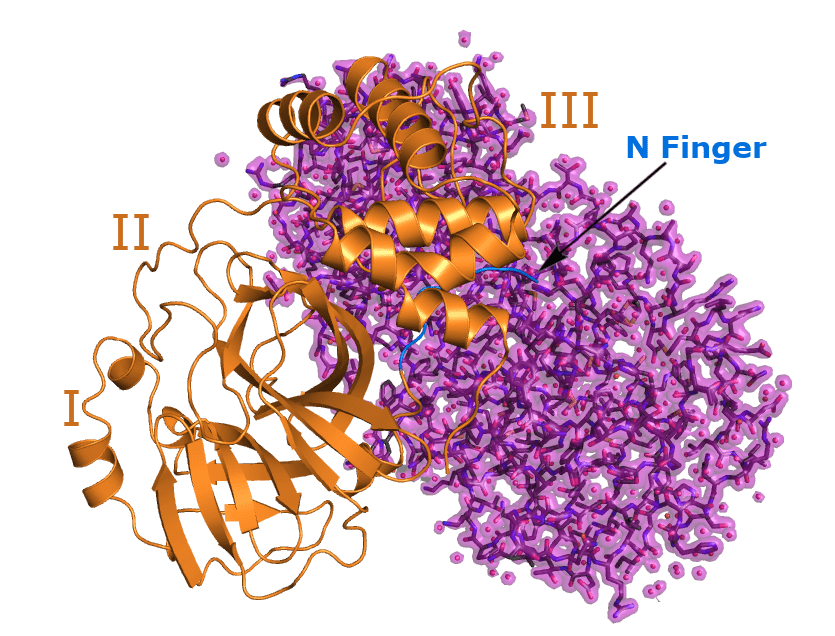
Die Hauptprotease ist eine Cysteinprotease, für die eine katalytische Dyade, gebildet aus den Aminosäuren Cystein und Histidin, typisch ist. Die Hauptprotease besteht aus zwei identischen senkrecht zueinander stehenden Protomeren, welche sich aus je drei Domänen zusammensetzen. An ihrem N-terminalen Ende liegen die Domänen I und II. Sie formen je eine antiparallele, chymotrypsinartige β-Tonnenstruktur, in der sich die Substratbindungsstelle befindet (siehe Bild weiter unten). Die Domäne III am C-terminalen Ende des Protomers besteht aus fünf α-Helices, die sich zu einem Cluster zusammenlagern und durch Ausbildung einer Salzbrücke zwischen Glu-290 des einen und Arg-4 des anderen Protomers zur Dimerisierung beitragen.
Die beiden Protomere werden auch durch den sogenannten "N-Finger" verbunden. Er besteht aus den letzten 7 Aminosäureresten am N-Terminus und stellt einen Kontakt zur Domäne II des jeweils anderen Protomers her, wodurch eine Kontaktfläche von ~1394 Å2 entsteht. Die Bildung des Homodimers ist unerlässlich für die Protease-Aktivität, da der Ser-1 Rest am N-terminalen Ende mit dem Glu-166 Rest des anderen Protomers interagiert und so die Substratbindungsstelle in Form hält. Diese Substratbindungsstelle besteht aus der katalytischen Dyade Cys-145 und His-41. Direkt daneben befindet sich die Substratbindungstasche S1, die aus den aus den Aminosäureresten von Phe-140 und His-163, sowie den Hauptkettenatomen von Glu-166, Asn-142, Gly-143 und His-172, besteht. Die Substratbindungsstelle bindet spezifisch Glutamin im Motiv Leu-Gln↓(Ser,Ala,Gly) des zu schneidenden Protein-Substrats, da das Carbonyl-Sauerstoffatom des Glutamins durch die Aminosäuren Gly-143 und Cys-145 stabilisiert wird.
Durch die Entwicklung von Inhibitoren, die gezielt an die Substratbindungsstelle der Protease binden, könnte die Spaltung der Polyproteine gehemmt, und somit die Virus RNA-Replikation gestoppt werden, so dass keine neuen Viren entstehen. Der Vorteil hierbei ist, dass bis heute keine menschlichen Proteasen mit ähnlicher Spezifität bekannt sind, weshalb eine Toxizität der Inhibitoren unwahrscheinlich ist. Potenzielle Inhibitoren lassen sich aufgrund ihrer chemischen Struktur in zwei Klassen unterteilen. Die erste Klasse umfasst Peptide, die spezifisch an die katalytische Stelle des Enzyms binden und durch kovalente Bindung mit Cys-145 die Substratbindung verhindern. Die zweite Klasse besteht aus kleinen organischen Molekülen, welche an Gruppen in der Substratbindungsstelle des Enzyms binden und so kompetitiv den Substrateintritt in den Hohlraum des aktiven Zentrums verhindern. Ein solcher Stoff, der als Wirkstoff zur Therapie von COVID-19 eingesetzt werden könnte, ist der HIV1-Protease-Inhibitor Lopinavir. Sollte sich die Wirksamkeit von Lopinavir gegen SARS-CoV-2 bestätigen, hätte das den Vorteil, dass Lopinavir bereits als HIV-Medikament für den Menschen zugelassen ist - und somit schneller zum Einsatz kommen könnte.
