Das Genom von SARS-CoV-2 codiert für ein langes Polyprotein ORF1a/ ORF1ab (ORF = offener Leserahmen). Es umfasst 16 nicht-strukturelle und vier strukturelle Proteine. Durch Leserasterverschiebungen (Engl: „Frame Shifts“) entstehen zusätzliche ORFs, die für sogenannte akzessorische Proteine codieren. Diese Gruppe von Proteinen steht im Verdacht einen großen Beitrag an der Pathogenese von SARS-CoV-2 zu leisten. Man nimmt an, dass eines dieser Proteine, das akzessorische Protein 7a, durch Induktion von apoptotischen Prozessen in menschlichen Wirtszellen maßgeblich an der Erkrankung Covid-19 beteiligt ist1.
Struktur von SARS-7a
Bislang ist es Forschern noch nicht gelungen die vollständige Proteinstruktur und Funktion von SARS-7a des neuen Coronavirus zu entschlüsseln. Das Protein besitzt aber in seiner Sequenz eine 85%ige Übereinstimmung und 95.2%ige Ähnlichkeit mit einem bereits bekannten Protein in SARS-CoV2. Somit kann angenommen werden, dass sich beide Proteine in Struktur sowie Funktion ähneln. Aus der Sequenzanalyse von SARS-CoV geht hervor, dass ORF7a für ein Typ I Transmembranprotein mit 122 Aminosäuren codiert3. Der N-Terminus umfasst ein Signalpeptid, bestehend aus sieben β-Strängen, die sich zu einem kompakten immunoglobulinartigen β-Sandwich, bestehend aus zwei β-Faltblättern, zusammenlagern (siehe Abbildung 1). Das erste β-Faltblatt umfasst die β-Stränge A, G, F, C und das zweite die Stränge B, E, D (Abbildung 1, links)4.
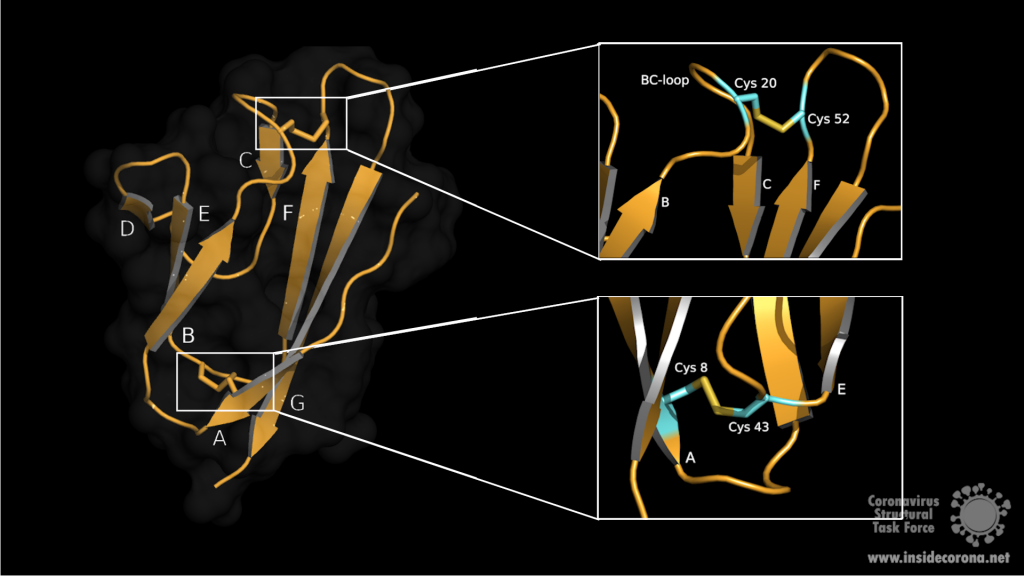
Die beiden amphipathischen Faltblätter sind eng aneinander gepackt, wobei die hydrophobe Seite innen liegt. Oberseitig wird die Ektodomaine über die β-Schleifen BC, DE und FG definiert. Die Unterseite bilden die Schleifen AB, CD und EF. Die Stabilisierung des β-Sandwichs erfolgt über zwei Disulfidbrücken. Die erste Disulfidbrücke liegt am unteren Ende der Faltblätter und verknüpft die Aminosäuren Cys8 auf Strang A mit Cys43 auf Strang E. An der Oberseite, auf der BC Schleife, liegt Cys20, welches über die zweite Disulfidbrücke mit dem Cys52 am Ende von Strang F verknüpft ist (siehe Abbildung 1, rechts). Oberhalb des BED-Faltblatts ragt die DE-Schleife aus dem β-Sandwich hervor und bildet mit den β-Strängen C und D eine Höhle. In dem Zentrum dieser hauptsächlich hydrophoben Vertiefung sitzt ein Glu18, das zur negativen Ladung am Boden der Höhle beiträgt. Aufgrund des negativen elektrostatischen Potentials ist es denkbar, dass diese Vertiefung eine potenzielle Interaktionsstelle für Liganden darstellt4.
Interaktionspartner von SARS-7a
Experimente mit Zellkulturen zeigen, dass SARS-7a vielfältige biologische Funktionen hat und auf unterschiedlichen Wegen in Zellprozesse eingreifen könnte5. Eine mögliche Schlüsselrolle ist die Zellzykluskontrolle. Die Überexpression von SARS-7a in HEK273 Zellen führte zu einer Inhibition des Zellwachstums und zum Zellzyklusarrest in G0/G1 Phase. Dieser Zellzyklusarrest kann die Virusreplikation begünstigen und die durch das Virus induzierte Pathogenität verstärken. Über die Interaktion von 7a mit einem Protein namens „B-cell lymphoma-extra large“ (Bcl-xL) kann Apoptose in menschlichen Nierenepithelzellen ausgelöst werden. Bcl-XL gehört zur B-cell lymphoma-2 Familie (Bcl-2), einer Gruppe von sogenannten „pro-survival“ Proteinen, die die Induktion der Apoptose inhibieren und so das Überleben der Zelle fördern. Durch die Wechselwirkung zwischen SARS-7a und der C-Terminalen Transmembrandomäne von Bcl-XL könnte dessen überlebenserhaltende Funktion unterdrückt und die Apoptose durch den Caspase-abhängigen Signalweg eingeleitet werden6,7.
SARS-7a kann auch eine Interaktion mit einer sogenannten Ap4a- Hydrolase, die in Prozesse der Zellproliferation, DNA-replikation, Apoptose und RNA-prozessierung involviert ist, eingehen und so deren Aktivität einschränken. Dieses Herunterregulieren der Hydrolaseaktivität führt zu einer gesteigerten Apa4 (Diadenosintetraphosphat) Produktion, wodurch es ebenfalls zu Apoptotischen Prozessen in der Zelle kommen kann5. Diese wirtszellenspezifische Regulation der Apoptose ermöglicht es dem Virus der Immunantwort zu entgehen und sich über weitere Organe auszubreiten.
Eine andere mögliche Funktion von ORF7a besteht in der Hemmung des Knochenmark-Matrix-Antigens 2 (BST-2), dass die Virusfreisetzung und so auch dessen Verbreitung, durch physisches Anheften der Virionen an die Plasmamembran, einschränken könnte. ORF7a antagonisiert diese einschränkende Wirkung durch die Bindung der extrazellulären Domäne von BST-2 wodurch dessen Glykosylierung verhindert wird. Wenn ein Wirkstoff gefunden wird, der in die ORF7a-BST-2 Interaktion eingreift, könnte dieser die Virusausbreitung verlangsamen oder sogar ganz stoppen8.
Zusammenfassung
Es lässt sich sagen, dass SARS-7a auf verschiedensten Wegen zur Pathogenität von SARS-CoV-2 beiträgt. Aus diesem Grund könnte die Entwicklung eines oder mehrerer Wirkstoffe, welche SARS-7a und dessen Interaktionen inhibieren, dabei helfen die Virusausbreitung zu verlangsamen und schwere Krankheitsverläufe verhindern.
6W37: Röntgenkristallstruktur des akzessorischen Proteins 7a, welches durch den Offenen Leserahmen ORF7a von SARSCoV-2 codiert wird.
1xak: Kristallstruktur des akzessorischen Proteins 7a von SARS-CoV. Das einzigartige typ I Transmembranprotein mit unbekannter Funktion hat eine kurze cytoplasmische Fraktion und eine Transmembrandomäne.
1y04: Röntgenkristallstruktur des akzessorischen Proteins X4, welches auch unter dem Namen 7a, U122 oder X4 bekannt ist. Das Protein lagert sich zu einem immunoglobulinartigen Betasandwich zusammen.
Weiterführende Literatur
- 1.Michel CJ, Mayer C, Poch O, Thompson JD. Characterization of accessory genes in coronavirus genomes. Virol J. Published online August 27, 2020. doi:10.1186/s12985-020-01402-1
- 2.Yoshimoto FK. The Proteins of Severe Acute Respiratory Syndrome Coronavirus-2 (SARS CoV-2 or n-COV19), the Cause of COVID-19. Protein J. Published online May 23, 2020:198-216. doi:10.1007/s10930-020-09901-4
- 3.Fielding BC, Tan Y-J, Shuo S, et al. Characterization of a Unique Group-Specific Protein (U122) of the Severe Acute Respiratory Syndrome Coronavirus. JVI. Published online July 15, 2004:7311-7318. doi:10.1128/jvi.78.14.7311-7318.2004
- 4.Hänel K, Stangler T, Stoldt M, Willbold D. Solution structure of the X4 protein coded by the SARS related coronavirus reveals an immunoglobulin like fold and suggests a binding activity to integrin I domains. J Biomed Sci. Published online November 23, 2005:281-293. doi:10.1007/s11373-005-9043-9
- 5.Vasilenko N, Moshynskyy I, Zakhartchouk A. SARS coronavirus protein 7a interacts with human Ap4A-hydrolase. Virology Journal. Published online 2010:31. doi:10.1186/1743-422x-7-31
- 6.Tan Y-J, Fielding BC, Goh P-Y, et al. Overexpression of 7a, a Protein Specifically Encoded by the Severe Acute Respiratory Syndrome Coronavirus, Induces Apoptosis via a Caspase-Dependent Pathway. JVI. Published online December 15, 2004:14043-14047. doi:10.1128/jvi.78.24.14043-14047.2004
- 7.Tan Y-X, Tan THP, Lee MJ-R, et al. Induction of Apoptosis by the Severe Acute Respiratory Syndrome Coronavirus 7a Protein Is Dependent on Its Interaction with the Bcl-XL Protein. JVI. Published online April 11, 2007:6346-6355. doi:10.1128/jvi.00090-07
- 8.Taylor JK, Coleman CM, Postel S, et al. Severe Acute Respiratory Syndrome Coronavirus ORF7a Inhibits Bone Marrow Stromal Antigen 2 Virion Tethering through a Novel Mechanism of Glycosylation Interference. García-Sastre A, ed. J Virol. Published online September 16, 2015:11820-11833. doi:10.1128/jvi.02274-15
