Die 3D Struktur eines Proteins zu kennen, erlaubt es Wissenschaftlern deren Form, Stabilität und mögliche Funktion zu untersuchen. Außerdem kann die Struktur, sofern sie von einem Krankheitserreger stammt, helfen mögliche Medikamente zu entwickeln. Jedoch ist das Lösen solcher Strukturen keine einfache Aufgabe, da Proteine zu klein sind, um mit optischen Mikroskopen untersucht zu werden. Zudem dauert die Vorbereitung der Proteinproben und die Datenverarbeitung auf dem Weg zur fertigen Struktur mehrere Wochen bis hin zu Monaten, was auch zu hohen Kosten führt. Doch 2021 änderte sich alles, als AlphaFold2 veröffentlich wurde – eine neue Software zur Vorhersage von Proteinstrukturen, welche nicht nur unglaublich akkurat, sondern auch einfach in der Handhabung ist. Sofort dominierte ein Hype rund um AlphaFold die Nachrichten, doch kann dieses Programm wirklich die Strukturbiologie verändern und wird es die gängigen Methoden obsolet machen?
Proteinstrukturen sichtbar machen
Doch bevor wir uns um die Lösung eines alten Problems der Biologie kümmern, frischen wir noch einmal die Grundkenntnisse auf. Also was waren Proteine noch mal? Kurzgesagt, sind Proteine kleine Nano-Maschinen, welche alle möglichen Aufgaben in unserem Körper erledigen. Während manche Proteine die Replikation von Zellen organisieren, verdauen andere unsere Mahlzeiten. Weitere Proteine sind der Hauptbestandteil unserer Haare und zahlreiche andere Dinge funktionieren nur mit Proteinen.
Was genau ein Protein macht und kann, wird durch seine Form bestimmt, weshalb wir überhaupt erst an der exakten 3D Struktur interessiert sind. Aber leider sind Proteine kleiner als sichtbares Licht und lassen sich daher nicht mit optischen Mikroskopen beobachten. Moment, kleiner als Licht? Geht das überhaupt? Nun, dieses Bild sollte bei der Erklärung helfen:
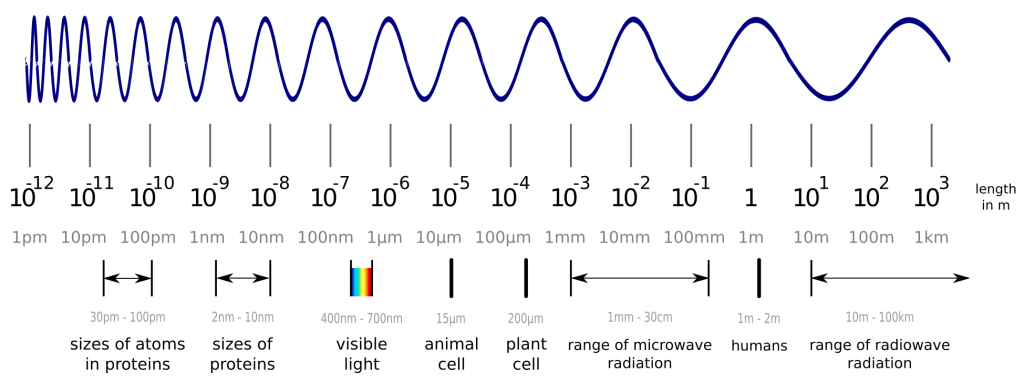
Während sichtbares Licht ohne Weiteres mit Objekten interagiert, die größer sind als die Wellenlänge des Lichts, interagiert es praktisch nicht mit Objekten die kleiner sind, wie z.B. einzelne Atome oder Proteine. Glücklicherweise können wir auch “Licht” im Spektrum dieser Größenordnung erzeugen, wobei wir anstelle unserer Augen einen passenden Detektor nutzen. Eine gängige Methode zur Lösung von Proteinstrukturen ist die Röntgen-Kristallographie. Die Details sind kompliziert und würden den Rahmen dieses Beitrags sprengen, doch die wichtigsten Schritte sind die Herstellung des gewünschten Proteins durch Bakterien und dessen Aufreinigung, die Zucht von Proteinkristallen (ja, man auch daraus Kristalle züchten!) und die Bestrahlung besagter Kristalle mit Röntgenstrahlen. Als Ergebnis erhalten wir Daten vom Detektor, mit denen wir ein 3D-Model des Proteins konstruieren können, welches zwar nicht exakt die Realität widerspiegelt, aber genau genug ist, um uns neue Erkenntnisse zu bringen. Mehr zu diesen 3D-Modellen haben wir bereits in diesem Blogpost erläutert.
Doch leider kann jede Menge schiefgehen: Das Protein könnte das Bakterium, welches es für uns herstellen soll, töten. Kristalle bilden sich nicht unter allen Bedingungen und auch die Datenverarbeitung ist kein Kinderspiel. Insgesamt kann uns diese Methode die gewünschten Ergebnisse bringen, doch sie kostet viel Zeit und Geld.
Das Proteinfaltungsproblem
Proteine bestehen aus langen Ketten von Aminosäuren, wobei nahezu alle Lebewesen, uns mit eingeschlossen, zwanzig verschiedene Aminosäuren nutzen. Jede von ihnen besitzt andere Eigenschaften und auch in der Größe können sie sich unterscheiden. Positiv geladene Aminosäuren zum Beispiel streben es an, von negativen Molekülen oder Wasser umgeben zu sein. Andere Aminosäuren besitzen gar keine Ladung und finden sich meist im Inneren des Proteins wieder, fern vom Wasser, welches das Protein umgibt.
Die Aminosäuren sind immer an denselben Stellen aneinander gekettet und ergeben so eine Sequenz, wobei die Eigenschaften der einzelnen Kettenglieder die Form und somit die Faltung des Proteins bestimmt. Würde man also eine Aminosäure der Kette durch eine andere mit deutlich anderen Eigenschaften ersetzen, so würde dies die Faltung des Proteins beeinflussen. Tatsächlich haben einzelne Änderungen meist keine große Auswirkung auf die gesamte Form, doch ändert man gleich mehrere Aminosäuren in der Sequenz, so erhält man schon wesentlich stärkere Unterschiede. Da die Form und Faltung eines Proteins auch dessen Funktion bestimmt, könnten solch veränderte Proteine auch ihre eigentliche Funktion verlieren oder neue Funktionen dazugewinnen. Die Kernaussage hierbei ist: Die Information über die 3D-Struktur eines Proteins liegt in der Sequenz der Aminosäuren versteckt. Doch wie genau die Aminosäurenkette sich zum fertigen Protein faltet, bleibt seit je her ein Mysterium und konnte bisher nicht in Simulationen repliziert werden. Dieses Mysterium ist heute bekannt als das Proteinfaltungsproblem.
Die versteckte Information in der Aminosäurensequenz motivierte Wissenschaftlerinnen weltweit an der Entschlüsselung der Proteinfaltung und an Programmen zur Vorhersage von Proteinstrukturen zu arbeiten. Schon bald traten die Wissenschaftlerinnen in der CASP Competition gegeneinander an – in der "Critical Assessment of Techniques for Protein Structure Prediction"-Competition. Seit 1994 wurden alle 2 Jahre die neusten Vorhersagetechniken miteinander verglichen, doch für Jahrzehnte blieben selbst die besten Ansätze zu fehlerhaft und unzuverlässig. Erst vor kurzem während der CASP13 im Jahr 2018 schaffte es Deepminds AlphaFold einen Großteil der gesuchten Strukturen nur anhand der gegebenen Eingabesequenzen mit hoher Qualität vorherzusagen. Und dennoch gab es viel Raum zur Verbesserung. Im Jahr 2020 folgte schließlich AlphaFold2, welches erstmalig in der Geschichte Strukturvorhersagen lieferte, die kaum von den Strukturen aus konventionellen Methoden zu unterscheiden sind. Das Proteinfaltungsproblem ist nun für mehr als 50 Jahre bekannt, aber erst mit der heuten Technik und neuen Methoden wie dem Einsatz von Deep Learning konnte die Strukturvorhersage endlich realisiert werden.
Wie genau ist AlphaFold?
Wenn von AlphaFold gesprochen wird, wird normalerweise AlphaFold2 gemeint. Während Wissenschaftlerinnen Formeln und Berechnungen nutzen, um die Ähnlichkeit der Vorhersagen und der experimentell gelösten Strukturen zu bestimmen, ist ein Bild wohl der beste Weg AlphaFolds Leistungen zu demonstrieren:
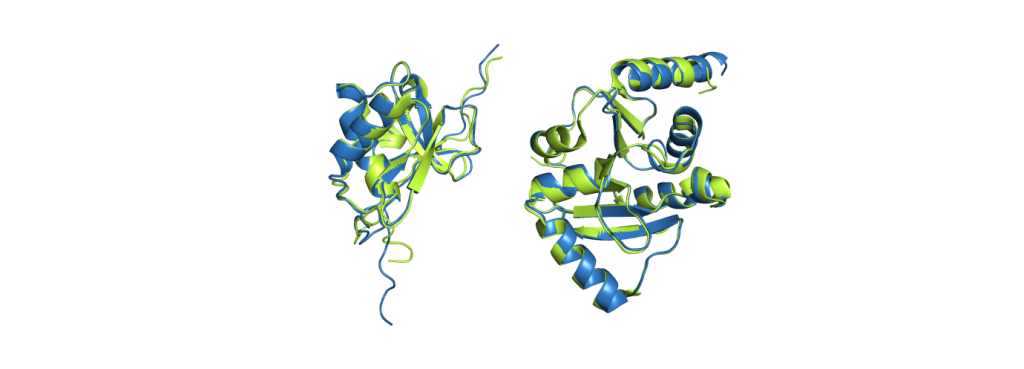
Die grünen Strukturen sind das Ergebnis experimenteller Methoden, die blauen hingegen sind die Vorhersagen von AlphaFold2. Allein vorherzusagen, welche Regionen der Aminosäurenkette sich in Helices und Faltblätter anordnen (die Spiralen und flachen Pfeile) ist schon eine herausfordernde Aufgabe. Doch AlphaFold2 sagt sogar deren relative Anordnung zueinander korrekt vorher und auch die Bereiche zwischen solcher Elemente stimmen häufig mit denen aus konventionell gelösten Strukturen überein. Die größten Unterschiede zwischen den Strukturen liegen gewöhnlich in den Enden der Kette. Obwohl AlphaFold2 keine exzellenten Resultate für alle Proteine erzeugt, so arbeitet es dennoch zuverlässig und gibt zudem einen Indikator über die Sicherheit der Vorhersage, was das Erkennen von potenziell fehlerhaften Regionen deutlich vereinfacht.
Und um den Durchbruch von AlphaFold2 in Relation zu setzen: Eine gängige Methode um die Ähnlichkeit zweier Strukturen zu messen ist der GDT, der Global Distance Test, welcher auch bei CASP zum Einsatz kommt. Dieser gibt einen Wert von 0% (absolut keine Ähnlichkeit) bis hin zu 100% (identische Strukturen) wieder. Während es zuvor keine Methoden bei der CASP Competition gab, welche die 60% Marke überschritten, schaffte es AlphaFold2 konsistent Vorhersagen mit einem GDT von über 90% zu erzeugen.
Wird AlphaFold konventionelle Methoden ersetzen?
Wie wir gesehen haben, sind die Vorhersagen von AlphaFold2 kaum von den Strukturen aus der Röntgenkristallographie zu unterscheiden. Heißt das also, dass wir die zeit- und kostenintensiven experimentellen Methoden nicht mehr benötigen? Nun, aus mehreren Gründen ist das nicht der Fall.
Der erste und wohl wichtigste Punkt: Vorhersagen sind nicht die Realität. Sie können einige Arbeitsschritte vereinfachen und uns näher ans Ziel bringen, aber sie können nicht alle Details realer Biologie mit berücksichtigen. AlphaFold2 betrachtet lediglich die Eingabesequenz, doch in der Realität sind Proteine umgeben von Wasser, weiteren Kleinmolekülen und auch anderen Proteinen, wobei alles miteinander interagiert und auch die Faltung beeinflussen kann. Abhängig von der Umgebung und diesen Interaktionen können Proteine sogar zwischen mehreren stabilen Faltungen wechseln. Daher bilden die Vorhersagen nur einen kleinen Teil vom Gesamtbild ab.
Ein weiteres Problem stellen die sogenannten Membranproteine dar. Proteine dieser Klasse verankern sich selbst in Membranen, wie z.B. die Wand einer Zelle und obwohl AlphaFold2 einzelne Teile solcher Proteine vorhersagen kann, scheitert es an der korrekten Zusammensetzung der Einzelteile. Dieses Problem taucht auch bei sehr großen Proteinen auf, die aus mehreren kleineren gefalteten Einheiten bestehen.
Zu guter Letzt gibt es auch Proteinstrukturen, die generell nicht korrekt vorhergesagt werden und somit auch grundsätzlich noch Verbesserungen übrig lassen.
Erwähnenswert ist auch die Tatsache, dass AlphaFold anhand der PDB, einer Datenbank für experimentell gelöste Strukturen, trainiert wurde. Ohne weitere experimentellen Daten könnte eine Vorhersage-Software wie AlphaFold gar nicht weiter verbessert werden.
Zusammenfassend lässt sich daher sagen, dass AlphaFold2 wirklich gut ist, aber noch lange nicht perfekt.
In jedem Fall sind die neuen Vorhersagen nicht nur gut um einen ersten Eindruck der Struktur zu erhalten, sondern unterstützen auch die konventionellen Methoden.
Erinnert ihr euch daran, dass die Zucht von Proteinkristallen einige Probleme bereiten kann? Nun, manchmal sind bestimmte Regionen daran schuld, welche sich nicht stabil falten lassen. AlphaFold2 kann solche Regionen vorhersagen und kann somit helfen, erfolgreichere Experimente zu planen.
Im Großen und Ganzen ist es ein unglaubliches Werkzeug, welches nicht nur neues Wissen innerhalb von Stunden statt von Tagen schafft, sondern auch noch von jedem genutzt werden kann und keinen Laborzugang erfordert. Ebenso wenig muss man ein Experte in der Strukturbiologie sein und lernt die Auswertung der Ergebnisse schon in wenigen Tagen. Und das ist nur der Anfang. Wir können neugierig auf die Zukunft der Strukturvorhersage blicken, da AlphaFold2 bereits heute Wissenschaftlern rund um die Welt die strukturellen Mysterien zahlreicher Proteine offenbart.

Hallo Max,
vielen Dank für diesen Artikel. Ich bin ganz begeistert, weil er eine so komplexe Materie so verständlich beschreibt, ohne in blumige Halbwahrheiten abzugleiten.
Ich kann mich nicht erinnern, wann ich so einen großartigen Text zuletzt auf Deutsch gelesen hätte.