Form folgt Funktion
Proteine sind große Moleküle, die aus 400 bis 20.000 Atomen bestehen können. Sie sind die Arbeitstiere der lebendigen Welt – Proteine verarbeiten Nahrung, bauen Muskeln, organisieren Zellteilung und Zellunterteilung und sie bilden Haut und Haare. Ihre Grundbausteine sind Aminosäuren, die eine lange Kette bilden. Diese Kette wird durch komplexe Faltung und Verknüpfung zum funktionellen Molekül. Die Sequenz der Aminosäuren - von denen es 20 verschiedene gibt – entscheidet hierbei über die Faltung des fertigen Moleküls, die man nicht immer vorhersagen kann. Aufgrund der (noch) zu hohen Komplexität müssen die Formen der Moleküle experimentell ermittelt werden.
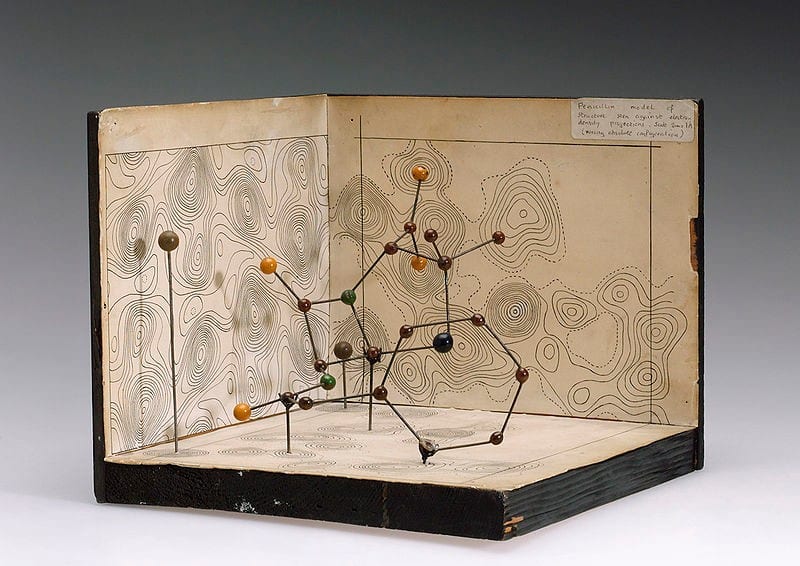
Eine Proteinstruktur verstehen heißt, zu verstehen wie sie funktioniert und was sie tut. Das Coronavirus trägt den genetischen Code für seine eigenen Proteine in sich, welche dann von der infizierten Wirtszelle produziert werden. Diese Proteine interagieren mit den eigentlichen menschlichen Proteinen. Es ist also wichtig, dass wir diese Proteine verstehen: Die Beeinflussung viraler Proteine oder die Beeinflussung der Interaktion mit der Wirtszelle kann eine Infektion stoppen und uns einen entscheidenden Vorteil im Kampf gegen den unsichtbaren Feind verschaffen - denn er ist jetzt sichtbar.
Aber wie misst und visualisiert man überhaupt etwas so Kleines wie ein Molekül, selbst ein großes? Gute Frage!
Nach der schwierigen Aufgabe, einen Kristall aus so einem großen (und irgendwie labberigem) Molekül zu formen, es wie ein(e) Irre(r) mit Röntgenstrahlen zu beschießen und die Streuung zu messen, werden die Daten mit einem Modell der Struktur interpretiert.
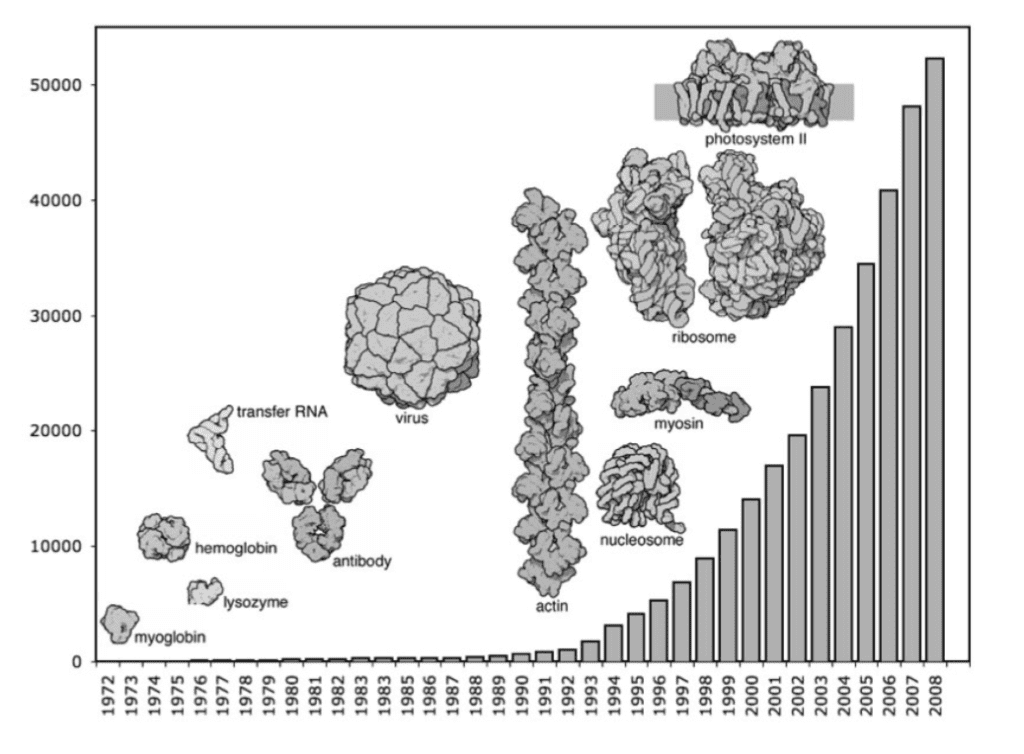
Aber auch mit einem Modell ist es oft kaum möglich, irgendetwas zu erkennen. Es gibt zu viele Atome. Das stete Wachstum der bekannten Strukturen erfordert eine bessere Darstellung zur Analyse und zum Vergleich. Einen großen Teil zur Lösung dieses Problems konnte Jane Richardson von der Duke Universität beitragen, als sie 1980 das Ribbon-Diagramm, oder Bändermodell, entwickelte.
Das Bändermodell
"Ich kann nicht sehen, wie man eine Proteinstruktur mit 1000 Wörtern beschreiben könnte, aber mit einem Bild kann man der Sache ziemlich nahekommen."
- Jane Richardson
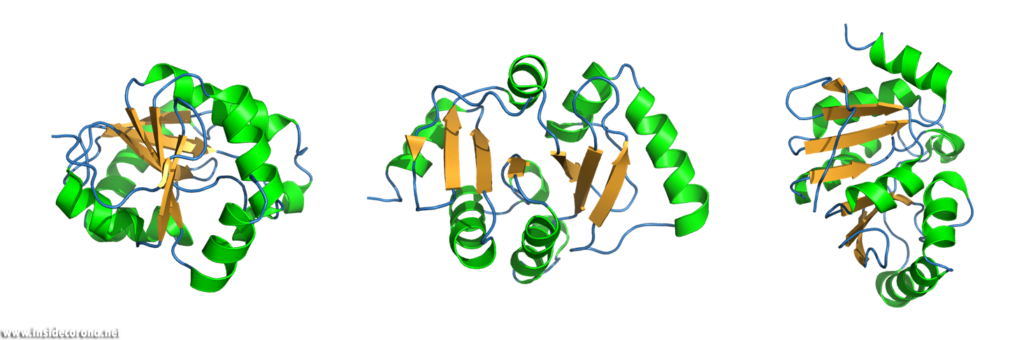
Das Bändermodell, eine dreidimensionale, schematische Darstellung, ist heutzutage die meistgenutzte Darstellungsform makromolekularer Strukturen. Das „Band“ zeigt das Rückgrat (= Aminosäurekette) des Proteins. Abhängig von Faltung, die durch sogenannte Wasserstoffbrückenbindungen erreicht wird, kann die Aminosäurekette in drei Kategorien, die Sekundärstrukturen, unterteilt werden: α-Helices, β-Faltblätter und Schleifen.
Diese werden dann als Band-Helices oder Pfeile dargestellt. Wenn es sich um um parallele Pfeile handelt, zeigen beide in die gleiche Richtung. Bei anti-parallelen, zeigen sie in die entgegengesetzte Richtung.
Schleifen werden als einfacher Schlauch, mit geringerem Durchmesser als Helices oder Faltblätter gezeigt. Diverse Merkmale können noch zusätzlich zum Bild hinzugefügt werden.
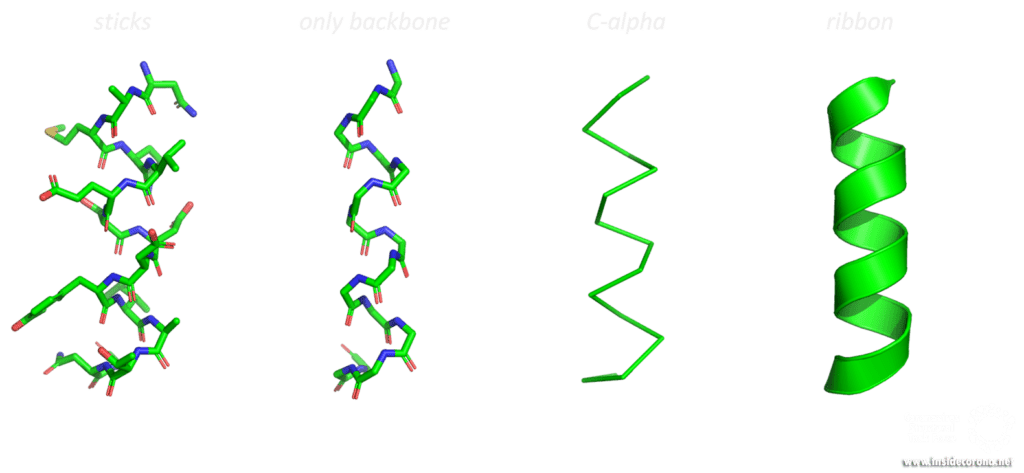
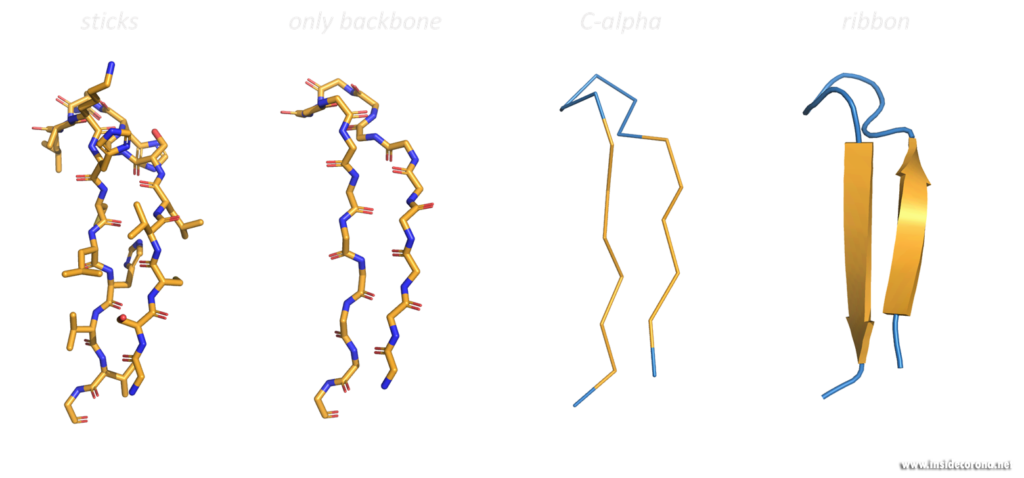
Strukturen mithilfe von Visualierungsmethoden wie dem Bändermodell zu untersuchen kann einen ausgezeichneten Überblick über die Faltung, Symmetrie und mögliche Interaktions- und Bindestellen im Protein verschaffen. Und strukturelle Eigenschaften bewirken verschiedene Funktionen in Proteinen.
Eine einheitliche Repräsentation der gewonnenen Daten ist der Schlüssel, um diese kleinen, fleißigen Maschinen Stück für Stück besser verstehen und miteinander vergleichen zu können.
Zum aktuellen Problemkind: Das SARS-Coronavirus-2 (oder hCoV2019).
Hier können Bändermodelle viraler Proteine zahlreiche Einblicke in virale Strukturen, ihre Funktion und ihre Rolle bei den Infektionen von Wirtszellen ermöglichen.
Es ist eine Kunst für sich, ein atomares Modell anhand von experimentellen Daten zu bauen. Diese Kunst und die Ideen von Jane Richardson und die anderer Strukturbiologinnen und -biologen beeinflussen tagtäglich das Denken von jungen Wissenschaftlern und unsere Sicht auf das Leben im molekularen Maßstab.
Mehr Informationen:
https://iubmb.onlinelibrary.wiley.com/doi/full/10.1002/bmb.2002.494030010005
https://research.duke.edu/ribbon-diagrams
https://blogs.sciencemag.org/pipeline/archives/2018/11/05/hail-to-the-ribbon
https://stories.duke.edu/sciences-mother-of-ribbon-diagrams-celebrates-50-years-at-duke

Ich habe vermutlich nur die Hälfte von dem Verstanden. Und das vermutlich auch nur halb so gut, wie es bei deiner Beschreibung angemessen gewesen wäre. Dennoch war es interessant zu lesen 🙂