Proteine sind komplexe und empfindliche, große Moleküle (sogenannte Makromoleküle), deren Strukturanalyse uns sehr viel über ihre Funktion verraten kann. Doch die Messung ist komplizierter als man annehmen mag. Man kann nicht einfach einen Blick durch ein Mikroskop werfen, fokussieren und das Protein sehen. Man kann es sich wie das Röntgenbild eines Arms vorstellen, mit dem kleinen Unterschied, dass der Arm hierfür abgenommen, tausendfach vervielfältigt und kristallisiert werden müsste, bevor er mit Röntgenstrahlen beschossen wird. Nachdem man sich dann noch mit viel Mathematik herumgeschlagen hat, würde man so das fertige Bild eines Arms erhalten.
Unterschiedliche Methoden
Die experimentell ermittelten Molekülstrukturen aus dem Coronavirus können aus drei Quellen stammen: Röntgenkristallographie, Kryo-Elektronenmikroskopie (Kryo-EM) oder Kernspinresonanz in Lösung (NMR). Jede dieser Methoden bringt dabei ihre ganz eigenen Vor- und Nachteile mit sich. Kombiniert man diese Methoden miteinander und mit weiteren Techniken wie Massenspektrometrie, chemischer Quervernetzung, fluoreszentem Resonanzenergietransfer und einigen Berechnungen, können die genauen Details der Molekülstruktur Stück für Stück ans Licht gebracht werden.
Kryo-Elektronenmikroskopie (Kryo-EM)
Wenn es darum geht, eine „große“ (aber immer noch sehr kleine) Molekülstruktur aufzulösen, kann die Elektronenmikroskopie einem einen ausgezeichneten strukturellen Überblick bieten. Im Gegensatz zu den anderen Techniken wird das Molekül direkt abgebildet. Das geschieht mit Hilfe eines Elektronenstrahls und eines Systems von Linsen. Der schwierige Teil ist dann die Umwandlung dieser 2D-Abbildungen in dreidimensionale Strukturen. Dafür bildet man das Objekt tausende Male aus verschiedenen Perspektiven ab, um es dann dreidimensional rekonstruieren zu können. Auch wenn Elektronenmikroskopie lange Zeit als Methode mit niedriger Auflösung galt, ermöglicht moderne Technologie eine immer höhere Auflösung, die fast den Detailreichtum der Röntgenkristallographie erreichen kann. Es können sogar Aminosäure-Seitenketten, Wassermoleküle auf den Oberflächen der Makromoleküle und Liganden erkannt werden.
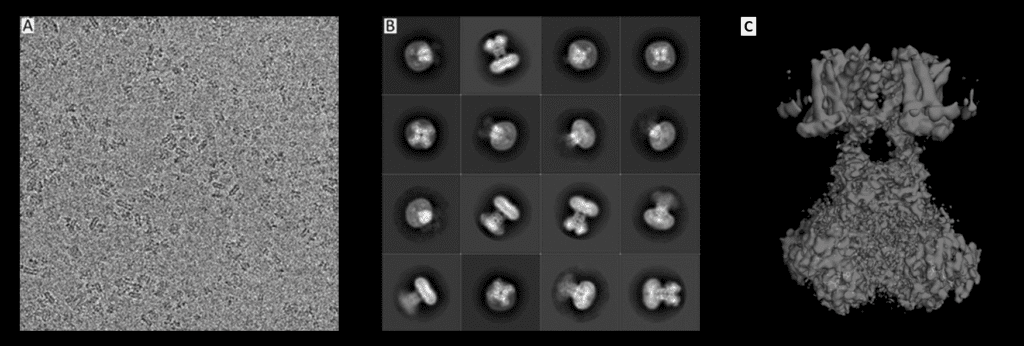
Original Bilder aus: Matthies, D., Bae, C., Toombes, G.E., Fox, T., Bartesaghi, A., Subramaniam, S., Swartz, K.J. (2018) Life 2018;7:e37558, bearbeitet von Ferdinand Kirsten, Lizenz: CC BY-ND 2.0
NMR-Spektroskopie
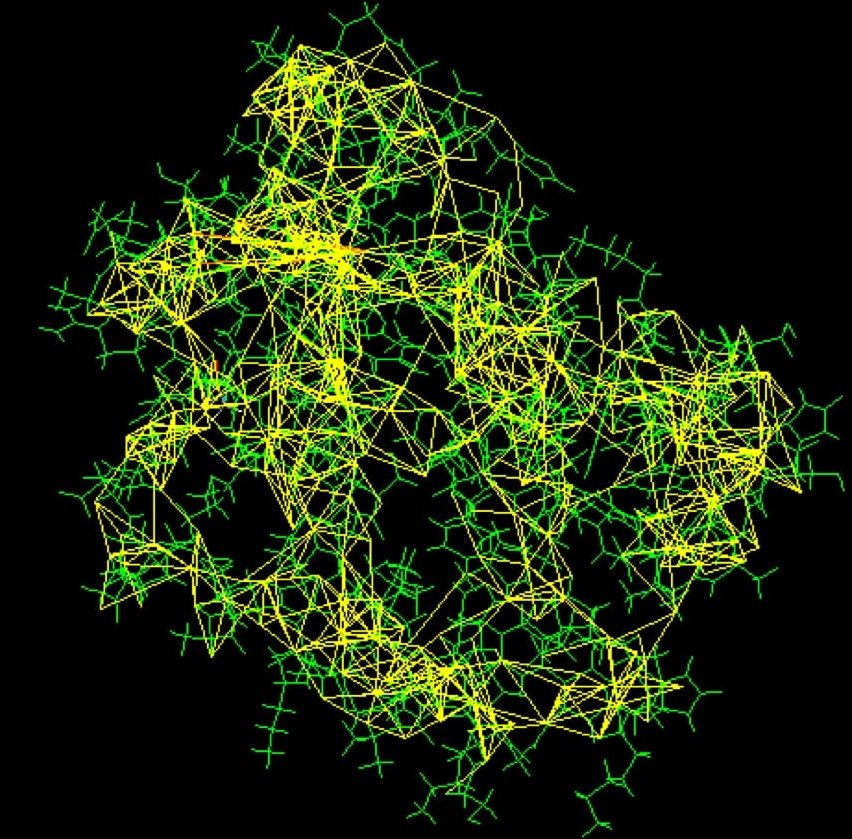
Ein wichtiger Teil der NMR-Spektroskopie ist die sogenannte Isotopenanreicherung. Während beim altbekannten MRT beim Arzt lediglich die Position der Atomkerne eines bestimmten Gewebes bestimmt werden kann, gestattet diese Methode eine genauere Analyse der Verteilung der Kohlenstoffatome. Dazu müssen sich die Kohlenstoffatome jedoch voneinander unterscheiden: Deshalb werden im Laufe des Aufarbeitungsprozesses verschiedene Isotope mit unterschiedlicher Neutronenzahl im Kern in das Proteinrückgrat eingebaut. Nach dieser Aufarbeitung wird das Protein einem starken Magnetfeld ausgesetzt und mit Radiowellen gemessen. Die entstehenden spezifischen Resonanzen jeder Substanz werden dann analysiert und man erhält Informationen über die Position jedes Kohlenstoff-Atomkerns im Verhältnis zu den anderen. Durch diese Positionsbestimmung kann man auf Distanzen oder mögliche chemische Bindungen schließen. Jetzt gilt es, das sudokuartige Rätsel richtig zu lösen, um ein atomares Modell des Moleküls erstellen zu können. Diese Methode eignet sich nur für kleine oder mittelgroße Moleküle, da größere Strukturen zu viel Überlappung in den Resonanzspektren verursachen. Die NMR-Spektroskopie kann jedoch damit punkten, dass die Messung flexibler Proteine in Lösung möglich ist. Sie ist an kein festes Stadium gebunden, das molekulare Bewegungen einschränken würde.
Röntgenkristallographie
Die Röntgenkristallographie ist, von den hier behandelten Methoden, die am häufigsten genutzte. Insgesamt sind 145252 der Strukturen in der Protein-Datenbank mittels Röntgen-Kristallographie gemessen worden. Die NMR-Spektroskopie (12965 Strukturen) und die Kryo-Elektronenmikroskope (4926 Strukturen) sind weitaus seltener vertreten. Das große Manko der Röntgenkristallographie ist die Notwendigkeit eines Proteinkristalls, aber sie liefert enormen Detailreichtum. Mit ihr kann man die Atome jeder Aminosäure und sogar von Liganden, Inhibitoren, Ionen oder anderen Molekülen bestimmen. Gleichzeitig schränkt aber der aufwändige Prozess der Kristallisation die Möglichkeiten ein, welche Proteine gemessen werden können. Die Aufarbeitung der Proteine für die Kristallisation bleibt eine anspruchsvolle Aufgabe. Nach der Aufarbeitung kann die Produktion eines messbaren Proteinkristalls einige Zeit, mitunter Jahre, in Anspruch nehmen. Ein besonderes Problem stellen dabei sehr flexible Proteine dar. Als Enzyme oder Rezeptoren besitzen Proteine häufig bewegbare Teile oder liegen in verschiedenen Konformationen vor, um vollständig funktionsfähig zu sein. Genau diese flexiblen Proteine sind leider oft die Interessantesten.
Ist der Kristall einmal hergestellt, wird er mit flüssigem Stickstoff gekühlt und mit einem intensiven Röntgenstrahl beschossen. Dieser Prozess ist vergleichbar mit einem Kristall, der ins Licht gehalten wird. Er wirft Reflektionen an eine Wand und diese werden dann angesehen. Die Röntgenstrahlen treffen den Kristall und werden in einem spezifischen Muster reflektiert. Diese Reflektionen sind kein direktes Bild des Kristalles, sondern müssen erst richtig interpretiert werden, um daraus auf die Kristallstruktur, also die molekulare Struktur im Kristall, schließen zu können. Die Verteilung der Elektronen kann aus den Reflektionen errechnet werden und liefert eine Elektronen-Dichtekarte, mit der die Position jedes einzelnen Atoms abgeschätzt werden kann.
Kristalle von Andrea Thorn, Diffraktionsmuster von Sabrina Stäb, Grafik von Ferdinand Kirsten.
Und dann...?
Die molekularen Modelle, die aus diesen Methoden gewonnen werden, eröffnen zahlreiche Möglichkeiten: Struktur-basiertes-Wirkstoffdesign, computerbasierte, dynamische Simulationen und natürlich Antworten auf wichtige biologische Fragen. Aber wie interpretieren wir diese Strukturen richtig, um alle biologischen Informationen zu bekommen? Das wird das Thema des nächsten Blog-Eintrags!
Zum Nachlesen (Englisch):
- https://pdb101.rcsb.org/learn/guide-to-understanding-pdb-data/methods-for-determining-structure
- https://proteopedia.org/wiki/index.php/Resolution
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4465431/
- https://pdb101.rcsb.org/learn/guide-to-understanding-pdb-data/r-value-and-r-free
- https://pdb101.rcsb.org/learn/guide-to-understanding-pdb-data/biological-assemblies
- https://www.rcsb.org/stats/summary

Die Aussen-Elektronen an den Virus-Spikes stellen atomphysikalisch
Energiewirbelketten dar mit eigenen Frequenzen ihrer Schwingungen.
So habe ich es beim Lesen von mod. Lit. zur Atomphysik verstanden.-
Die optimale Energieübertragung (Informationsübertragung) des Virus
auf eine Wirtszelle haben wir bei Resonanzfrequenz.
Mit Störfrequenzen könnte man das Virus hemmen bzw. eliminieren.
Beispiel: UV-Licht. Abwehrzellen im Immunsystem züchten, die UV -Impulse abgeben können, eine Art Fluoreszenz.
?? Utopischer Gedanke meinerseits?