Das SARS-CoV-2 Virus bringt riesige Einschränkungen für uns und unser Leben mit sich. Im krassen Kontrast dazu steht, wie winzig das Virus tatsächlich ist. Diese verschwinden geringe Größe des Virus ist auch das, was unserem Verständnis des Erregers, und damit auch unserer Möglichkeit ihn zu bekämpfen, im Wege steht. In Zeiten von Fake-news, Politikern, die die Krise ausnutzen um Falschinformationen und Missgunst zu sähen, sowie Zweifel und Unsicherheiten bezüglich der Virusmutationen und Impfungen, ist es durchaus legitim die Grundlagen zu hinterfragen:
„Wie können wir uns der Existenz von Viren sicher sein, wenn sie selbst zu klein für ein Mikroskop sind.“
Diese Frage beschreibt ein naturwissenschaftliches Problem, vor dem Mikrobiologen und Ärzte bereits im 19. Jahrhundert standen.
Hier werden wir uns auf die Suche nach den Beweisen für die Existenz von Viren machen, und auf dem Wege gleich die beiden Methoden betrachten, die die Wissenschaftler anwandten, um die Viren zu finden und ihre Eigenschaften zu bestimmen.
Methode 1: Beweis durch Kausalität
Lasst uns zunächst einen Blick auf das Tollwutvirus werfen: Übertragen wird es üblicherweise durch den Biss von befallenen Tieren. Bis die Krankheit daraufhin im gebissenen Menschen ausbricht vergehen ein bis drei Monate. Nachdem dann die ersten Symptome aufgetreten sind, weist Tollwut eine Tödlichkeit von 100% auf. Diese Symptome reichen anfangs von Fieber, intensiven Schmerzen und einem wildgewordenen Verdauungstrakt bis im späteren Verlauf zu Hyperaktivität und Hydrophobie. Dieses letzte Symptom beschreibt eine starke Abneigung gegenüber Wasser: die infizierte Person möchte den eigenen Speichel nicht mehr schlucken und fühlt sich generell unwohl in der Nähe von Flüssigkeiten. Tollwut ist eine schreckliche Krankheit, und Ärzte wie Wissenschaftler haben jahrelang nach der Ursache gesucht. Und selbst, nachdem Bakterien gefunden und als Erreger (oder auch Pathogene) für viele Krankheiten identifiziert wurden, blieb die Ursache für Tollwut ein Mysterium.
Die Wissenschaftler damals mussten sich also fragen: Wenn kein Bakterium als Ursache für Tollwut gefunden werden konnte, was ist dann die Ursache? Ist das Pathogen den Bakterien zumindest ähnlich, oder einfach so andersartig, dass es mit nichts Bekanntem vergleichbar ist? Hat Tollwut überhaupt eine physische Ursache?

Luis Pasteur, ein französischer Mikrobiologe, ist bei seinen Versuchen den Antworten zu diesen Fragen bereits im 19. Jahrhundert auf der Spur gewesen und einen großen Schritt näher gekommen. Erfolge mit Milzbrand und Geflügelcholera hatten ihn bereits als eine wichtige Instanz der Medizin und Mikrobiologie etabliert, und doch war auch seine Suche nach dem Tollwutpathogen durch die Begrenzungen der damaligen Mikroskopietechniken zum Scheitern verurteilt.
Die Schwere und der immer tödliche Verlauf der Tollwutkrankheit veranlassten Pasteur aber dennoch dazu nach einem Impfstoff zu suchen. Einem Impfstoff gegen ein Pathogen, für dessen Existenz er außer den Krankheitssymptomen keinen Hinweis hatte. Aber indem er aus seinen Erfahrungen mit bakteriellen Erregern schöpfte schaffte er es sogar: Er umging die Notwendigkeit den Erreger zu isolieren, indem er stattdessen infiziertes Nervengewebe verwendete, um die Krankheit schrittweise an unterschiedliche Säugetierspezies wie Hasen oder Hunde weiterzugeben. Mit jedem dieser Infektionszyklen verringerte sich die Schädlichkeit und Infektiosität (also Virulenz) des Virus gegenüber dem Menschen. Im Jahre 1885, nur wenige Jahre nachdem er den entwickelten Impfstoff an Hunden testete, rettete Pasteur mit der Impfung das Leben des neunjährigen Jungen Joseph Meister, welcher dadurch vermutlich die erste Person war, die jemals gegen Tollwut immun war.

Die Tatsache, dass Pasteurs Impfstoff - ohne dass die wahre Natur des Tollwuterregers bekannt war - Menschen (und Hunde) vor einem Ausbruch der Tollwut schützen konnte, zeigte, dass die Art und Weise mit der ein Tollwuterreger mit unserem Körper kommuniziert zumindest damit vergleichbar ist, wie Bakterien es tun. Was gleichzeitig beweist, dass der Erreger physisch, und daher auffindbar sein muss.
Die Entwicklung des Chamberland Filters von Pasteurs damaligem Assistenten Charles Chamberland, grenzte die möglichen Eigenschaften des Tollwuterregers weiter ein. Das Herzstück des Filters ist eine einseitig geöffnete Porzellanröhre, durch die eine Flüssigkeit gefiltert werden kann, wenn sie mit genügend Druck in die Röhre hineingeführt wird.
Dieser Filter war in der Lage, alle bekannten und beobachtbaren Mikroorganismen, inklusive pathogener Bakterien, von einer Flüssigkeit zu trennen. Der russische Biologe Dimitry Iwanowski, der sich mit der Mosaikkrankheit von Pflanzen beschäftigte, filterte 1892 den extrahierten Saft infizierter Blätter mit einem solchen Filter. Die gefilterte Flüssigkeit - unter dem Mikroskop scheinbar frei von jeglichen Partikeln – war nach Iwanowskis Beobachtung jedoch immer noch in der Lage, gesunde Pflanzen zu infizieren. Außerdem beobachtete er, dass anders als ein isolierter Bakterienstamm, der nach einiger Zeit in der Flasche ohne Nährstoffe abstirbt und seine Infektiosität verliert, das Filtrat für viele weitere Monate infektiös blieb. Diese Versuche enthüllten die nervige Fähigkeit der Viren ohne Stoffwechsel zu „überleben“.

Dieses Experiment wurde mit vielen anderen Krankheiten wiederholt, ebenso auch mit Tollwut. Der Holländische Mikrobiologe Martinus Beijerinck führte 1898 zunächst das Wort „Virus“ für diese infektiösen Flüssigkeiten ein (vom lateinischen – virus: „Gift, Schleim“). Und auch wenn die Wissenschaftler sich nicht sicher waren, ob die Erreger flüssig oder partikelartig sind, wusste man zumindest, dass sie in Wasser löslich sein müssen und klein genug, um durch den Chamberland Filter zu passen. Dies ist ein sehr gutes Bespiel dafür, wie Wissenschaftler die Existenz einer unbekannten Sache demonstrieren, ohne sie zu sehen, sondern nur durch die Beobachtung der Auswirkungen, die diese unbekannte Sache auf ihre Umwelt hat. Ein Beweis durch Kausalität. Sogar einige Annahmen zur wahren Natur der unbekannten Sache waren den Wissenschaftlern in diesem Beispielfall möglich.
Aber „Der Mensch glaubt nur das, was er sieht.“, daher lasst uns mit unserer Suche fortfahren, diesmal nach einem visuellen Beweis:
Methode 2: Beweis durch Visualisierung.
In den Jahren nach der Entdeckung dieser infektiösen Flüssigkeiten – „Viren“ genannt – hat sich die Mikroskopietechnik immer weiterentwickelt, bis zu dem Punkt, an dem geklumpte Häufchen von Viruspartikeln tatsächlich sichtbar gemacht werden konnten. Einige Wissenschaftler sahen diese Partikelklumpen vermutlich, ohne sie als die Erreger identifizieren zu können, die sie waren. Alles in Allem waren Vergrößerung und Auflösung noch nicht gut genug, um Details oder Strukturen zu erkennen. Dies liegt an dem natürlichen physikalischen Auflösungslimit von Lichtmikroskopen. Um dies zu erläutern und den anschließenden Schritt besser zu verstehen, müssen wir einen kurzen Exkurs machen und uns anschauen, wie Licht und Materie interagieren:
Licht kann als Welle betrachtet werden. Reflektion, Brechung und Beugung sind, neben Absorption und Emission, die Hauptinteraktionen zwischen Wellen und Materie und erlauben uns letztendlich zu Sehen. Was all diese Mechanismen allerdings gemeinsam haben ist, dass sie zu einem gewissen Grad von dem Größenunterschied zwischen der Wellenlänge und dem angestrahlten Objekt abhängig sind. Objekte, die deutlich kleiner sind als die Wellenlänge des Lichts, werden von den Lichtwellen einfach „ignoriert“. Da Wellen jeglicher Art viele Eigenschaften teilen, können wir uns hier zur Veranschaulichung den Vergleich mit Wasserwellen zu Nutze machen: Stellt euch einige, etwa einen Meter große Felsen in der Brandung am Strand vor. Betrachtet man lediglich die Wellen, die am Strand ankommen, könnte man bereits Annahmen über Größe und Position der Felsen im Wasser treffen. Stellen wir uns nun eine 20 m hohe Riesenwelle vor wie sie auf die Felsen und den Strand zurollt. Sie würde einfach über die Felsen hinwegfegen, ohne dass man danach anhand der Form der Welle auf die Existenz der Felsen schließen könnte. Ähnlich verhält sich dies auch bei Lichtwellen und Objekten, die viel kleiner als die Wellenlänge sind.* Um Zahlen zu nennen: Das Spektrum von Licht, das in der Lichtmikroskopie genutzt wird, reicht von Wellenlängen ab 700 nm bis ca. 400 nm. Wenn wir die Wellen-Objektwechselwirkungen und die Lichtführung in einem Mikroskop berücksichtigen und etwas Mathematik hinzuziehen, dann können wir das theoretische Auflösungslimit eines Lichtmikroskops zu etwa 250 nm berechnen. Das heißt, dass unter einem hochmodernen Mikroskop Strukturen ab einer Größe von 250 nm voneinander unterschieden werden können. Aber selbst heutzutage ist es sehr kostspielig und ressourcenintensiv, diesem Auflösungslimit nahe zu kommen. Und die meisten Viren sind ohnehin kleiner. Das SARS-CoV-2 Virus hat einen Durchmesser von ungefähr 120 nm, das ist weniger als die Hälfte des theoretisch möglichen Auflösungslimits.
Nun verstehen wir, warum uns das Lichtmikroskop nicht bei der Visualisierung von Viren helfen kann. Aber, welche anderen Instrumente stehen uns zur Verfügung?
Ich stelle vor: das Elektronenmikroskop!

Dieses revolutionäre Gerät wurde von den deutschen Wissenschaftlern Max Knoll und Ernst Ruska im Jahre 1931 entworfen. Kurz zuvor wurde die Hypothese des französischen Physikers Lois de Broglie, darüber das Partikel auch Wellencharakter annehmen können, weltweit bekannt. Diese Hypothese besagt außerdem, dass die Wellenlänge des Partikels sich mit zunehmender Geschwindigkeit weiter verkürzt. Sehr viel kürzere Wellenlängen als die 250 nm des Lichtmikroskops waren somit plötzlich möglich. Mit diesem Konzept arbeitet das Elektronenmikroskop und umgeht das Problem der „zu großen“ Wellenlängen von Licht, indem es sich den Wellencharakter stark beschleunigter Elektronen zu Nutze macht. Die optischen Linsen aus dem Lichtmikroskop werden hier durch magnetische Linsen – also Spulen - ersetzt, die Elektronen mit magnetischen Feldern leiten und fokussieren. Nachdem die Elektronen die Probe passiert haben, werden sie auf einem Fluoreszenzschirm aufgefangen, der die Elektronen detektiert und in für uns sichtbare Signale übersetzt.
Da Elektronen theoretisch bis nahe an die Lichtgeschwindigkeit beschleunigt werden können, könnten auch ihre Wellenlängen weiter und weiter verkürzt werden, so dass das theoretische Auflösungslimit unbegrenzt ist. Relativistische Effekte, exponentiell steigende Energiekosten für Beschleunigungen nahe der Lichtgeschwindigkeit und technische Aspekte der magnetischen Linsen beschränken das Auflösungslimit jedoch zu immer noch unfassbar kleinen 0,1 – 0,2 nm.
Die Entwicklung des Elektronenmikroskops öffnete der Wissenschaft die Türen zu kleineren Ebenen des Mikrokosmos, und wie der Zufall es wollte, war Ernst Ruskas Bruder, Helmut Ruska, ein Arzt und Biologe. Sehr früh bereits bemerkte dieser das Potential der Erfindung seines Bruders für die Mikrobiologie, und zusammen mit einem Kollegen, Bodo von Borries, publizierten sie im Jahr 1938 das erste mit einem Elektronenmikroskop aufgenommene Bild eines Virus.
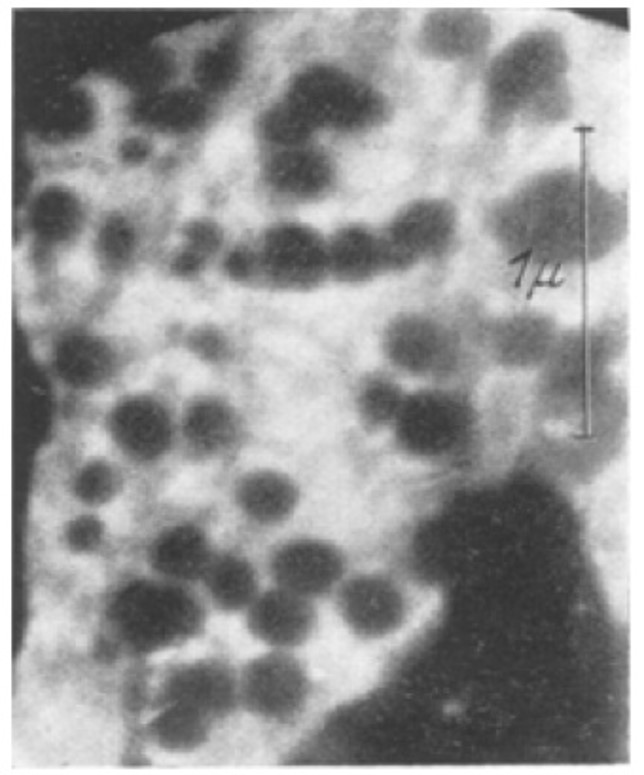
Es folgte ein “Goldenes Zeitalter“ für die Virologie, in dem das Elektronenmikroskop nach und nach die wahre Natur der Viren enthüllte und den Wissenschaftlern erlaubte deren Funktionsweise zu verstehen und wie sie zu klassifizieren sind.
Und das neue Coronavirus?
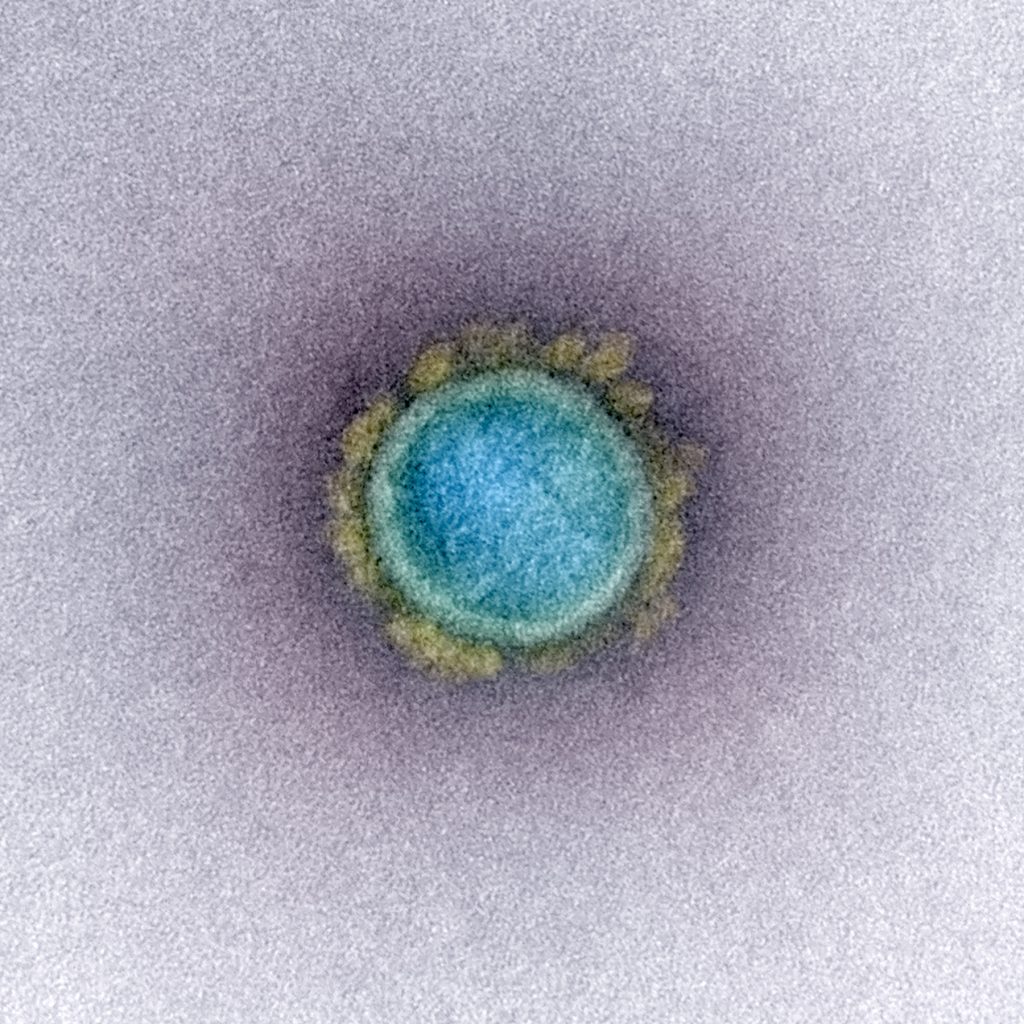
Wir springen ins Jahr 2020: Die Weiterentwicklung des Elektronenmikroskops wurde in den letzten 90 Jahren vorangetrieben, neue Techniken entwickelt und bestehende Techniken verfeinert. Nach dem Aufkommen des Coronavirus zu Beginn des Jahres war es also nur eine Frage der Zeit, bis EM-Bilder des SARS-CoV-2 Virus veröffentlicht wurden. Ein unschlagbarer Beweis für die Existenz des Virus!
Weiterlesen und Quellen:
Von Borries, B., E. Ruska, und H. Ruska. "Bakterien und Virus in übermikroskopischer Aufnahme." Klinische Wochenschrift 17.27 (1938): 921-925.
Ackermann, Hans-W., und Hans-W. Ackermann. "The first phage electron micrographs." Bacteriophage 1.4 (2011): 225-227.
Knoll, Max, und Ernst Ruska. "Das elektronenmikroskop." Zeitschrift für physik 78.5-6 (1932): 318-339.
Kruger, D. H., Peter Schneck, und H. R. Gelderblom. "Helmut Ruska and the visualisation of viruses." The Lancet 355.9216 (2000): 1713-1717.
Horzinek, Marian C. "The birth of virology." Antonie van Leeuwenhoek 71.1-2 (1997): 15-20.
- *Diese Analogie ist zwar nicht zu 100% genau, aber konzeptuell richtig, und reicht hier aus.

[…] Frage wurde uns in den vergangenen Monaten immer wieder gestellt: Existiert das Virus wirklich? Kann man beweisen, dass es wirklich Zellen befällt und so der Auslöser für COVID-19 ist? Hier […]
Solange ein Virus nicht isoliert wird und gemäss Henle Koch'sche Postulate bewiesen wird, ist alles nach wie vor unklar.
https://insidecorona.net/de/volle-kanne-nachgefragt-die-vier-postulate-des-herrn-koch/