Für die Bekämpfung der Lungenkrankheit COVID-19 und um eine weitere Ausbreitung der Pandemie zu stoppen, ist die Entwicklung neuer Medikamente essenziell. Ein vielversprechendes Angriffsziel für neue Wirkstoffe gegen den Erreger, SARS-CoV-2, ist die 3-Chymotrypsin-like Protease (3CL-protease), auch bekannt als Hauptprotease oder Mpro.
Damit neue Viren in der Wirtzelle gebildet werden können, muss die ssRNA des Virus in Proteine translatiert werden. Ein Großteil der Virusproteine werden als lange Polyproteine translatiert. Damit funktionale Proteine daraus entstehen können, müssen diese von Proteasen wie der Hauptprotease zerschnitten werden. Die Prozessierung der Polyproteine ppa1a und ppa1ab erfolgt an 11 Stellen in Richtung des C-terminalen Endes (abwärts von Nsp4). Für die Bildung von neuem Viruserbmaterial wird der RNA-abhängige RNA-Polymerasekomlex benötigt, der ein Teil dieser Polyproteine ist (nämlich Nsp7, Nsp8, Nsp12 und Nsp14). Seine Bildung kann durch eine gezielte Inhibition der Hauptprotease gestoppt werden, und damit die RNA-Replikation.
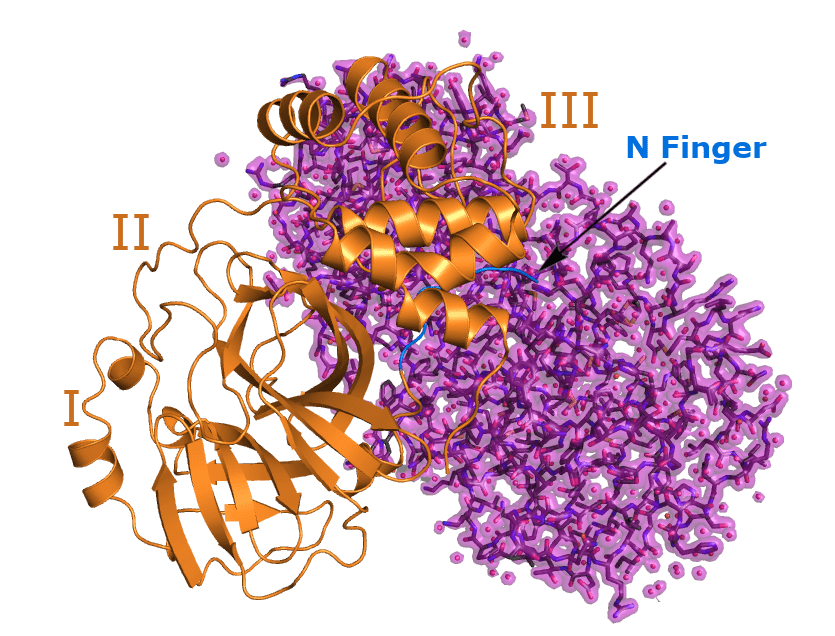
Die Hauptprotease ist eine Cysteinprotease, für die eine katalytische Dyade, gebildet aus den Aminosäuren Cystein und Histidin, typisch ist. Die Hauptprotease besteht aus zwei identischen senkrecht zueinander stehenden Protomeren, welche sich aus je drei Domänen zusammensetzen. An ihrem N-terminalen Ende liegen die Domänen I und II. Sie formen je eine antiparallele, chymotrypsinartige β-Tonnenstruktur, in der sich die Substratbindungsstelle befindet (siehe Bild weiter unten). Die Domäne III am C-terminalen Ende des Protomers besteht aus fünf α-Helices, die sich zu einem Cluster zusammenlagern und durch Ausbildung einer Salzbrücke zwischen Glu-290 des einen und Arg-4 des anderen Protomers zur Dimerisierung beitragen.
Die beiden Protomere werden auch durch den sogenannten "N-Finger" verbunden. Er besteht aus den letzten 7 Aminosäureresten am N-Terminus und stellt einen Kontakt zur Domäne II des jeweils anderen Protomers her, wodurch eine Kontaktfläche von ~1394 Å2 entsteht. Die Bildung des Homodimers ist unerlässlich für die Protease-Aktivität, da der Ser-1 Rest am N-terminalen Ende mit dem Glu-166 Rest des anderen Protomers interagiert und so die Substratbindungsstelle in Form hält. Diese Substratbindungsstelle besteht aus der katalytischen Dyade Cys-145 und His-41. Direkt daneben befindet sich die Substratbindungstasche S1, die aus den aus den Aminosäureresten von Phe-140 und His-163, sowie den Hauptkettenatomen von Glu-166, Asn-142, Gly-143 und His-172, besteht. Die Substratbindungsstelle bindet spezifisch Glutamin im Motiv Leu-Gln↓(Ser,Ala,Gly) des zu schneidenden Protein-Substrats, da das Carbonyl-Sauerstoffatom des Glutamins durch die Aminosäuren Gly-143 und Cys-145 stabilisiert wird.
Durch die Entwicklung von Inhibitoren, die gezielt an die Substratbindungsstelle der Protease binden, könnte die Spaltung der Polyproteine gehemmt, und somit die Virus RNA-Replikation gestoppt werden, so dass keine neuen Viren entstehen. Der Vorteil hierbei ist, dass bis heute keine menschlichen Proteasen mit ähnlicher Spezifität bekannt sind, weshalb eine Toxizität der Inhibitoren unwahrscheinlich ist. Potenzielle Inhibitoren lassen sich aufgrund ihrer chemischen Struktur in zwei Klassen unterteilen. Die erste Klasse umfasst Peptide, die spezifisch an die katalytische Stelle des Enzyms binden und durch kovalente Bindung mit Cys-145 die Substratbindung verhindern. Die zweite Klasse besteht aus kleinen organischen Molekülen, welche an Gruppen in der Substratbindungsstelle des Enzyms binden und so kompetitiv den Substrateintritt in den Hohlraum des aktiven Zentrums verhindern. Ein solcher Stoff, der als Wirkstoff zur Therapie von COVID-19 eingesetzt werden könnte, ist der HIV1-Protease-Inhibitor Lopinavir. Sollte sich die Wirksamkeit von Lopinavir gegen SARS-CoV-2 bestätigen, hätte das den Vorteil, dass Lopinavir bereits als HIV-Medikament für den Menschen zugelassen ist - und somit schneller zum Einsatz kommen könnte.
