Neben mRNA-Impfstoffen wird auch eine andere Klasse von Impfstoffen gegen COVID-19 eingesetzt: Der Vektorimpfstoff von AstraZeneca enthält ein intaktes Virus. Doch wie wirkt er genau? Wo liegen Stärken und Schwächen? Und vor allem: Ist er sicher?
Die neuartigen mRNA-Impfstoffe gegen SARS-CoV-2 lösen einige Kontroversen aus, und die Bedenken sind vielfältig. Die Impfstoffe seien zu wenig getestet worden, man kenne die Langzeitfolgen nicht, die Impfstoffe könnten das Erbgut verändern.
Der Impfstoff von Oxford–AstraZeneca funktioniert anders als etwa der mRNA-Impfstoff von BioNTech. Statt nur das Erbgut des Stachelproteins in unsere Zellen zu bringen, befindet sich ein Teil des Genoms von SARS-CoV-2 in einem harmloseren, aber funktionsfähigen Virus. Über diesen Ansatz wirken die sogenannten "viralen Vektorimpfstoffe". Als Vektor (Überträger) der DNA des Virusproteins dient ein verändertes Erkältungsvirus.
Die Europäische Arzneimittelagentur EMA empfahl den Impfstoff Ende Januar zur Zulassung. Warum?
Viren sind Transporter für Gene
Viren gelten nicht als Lebewesen, denn sie verfügen nicht über einen eigenen Stoffwechsel. Stattdessen enthalten sie genetisches Material, das die Wirtzelle dazu bringt, neue Viren zu produzieren.
In der Evolution der Viren haben sich einige ihrer Fähigkeiten außergewöhnlich verfeinert. Viren können Wirtszellen spezifisch infizieren, der Immunabwehr des Wirtes entkommen und ihr Erbgut in jenes des Wirtes einbauen. Seit Molekularbiolog:innen diese Fähigkeiten erkannten und im Zeitalter von Genetic Engineering auch nutzen können, sind Viren zu Werkzeugen der Wissenschaft geworden.
Viren als Werkzeuge
Um Viren für Forschungszwecke zu verwenden, werden sie modifiziert. Ihre natürlichen Gene müssen verändert oder gelöscht werden. Was am Ende übrig bleibt, ist ein Transporter, mit dem man erwünschte Gene in einen Wirt einschleusen kann. Diese Methodik wird zum Beispiel zur Herstellung von transgenen (also erbgutveränderten) Pflanzen oder Zelllinien verwendet. Einige natürliche Fähigkeiten des Virus behält es dabei für gewöhnlich bei, wie z. B. das Eindringen in die Wirtszelle.
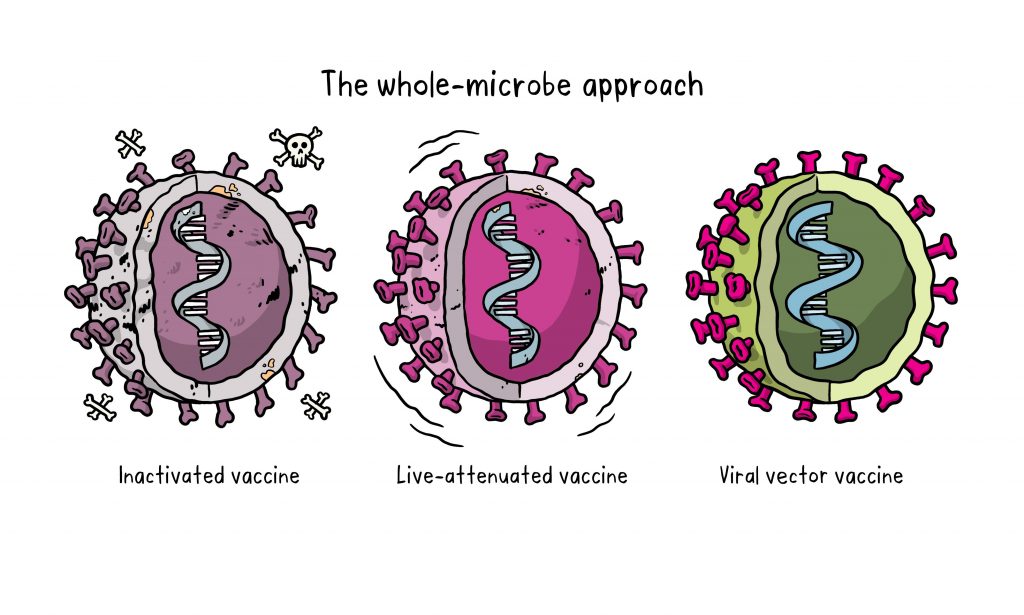
Viren als Impfstoffe
Wie genau sieht nun ein Virus aus, das zum Impfstoff umgewandelt wurde?
Um nicht doch eine Krankheit auszulösen, muss dem Virus die Fähigkeit genommen werden, sich im Körper zu vermehren. Viren ohne die Fähigkeit zur Reproduktion bezeichnet man als "nicht replizierend". Viren, die auf eine Weise modifiziert wurden, die ihre krankmachenden Eigenschaften unterdrückt, die Reproduktionsfähigkeit aber weitestgehend erhält, bezeichnet man als "attenuiert".
Virale Vektoren wurden erstmals 1972 beschrieben [1]. Anfang der 90er Jahre wurde erstmals Gentransfer vermittels eines attenuierten Adenovirus als Vektor zu therapeutischen Zwecken genutzt [2]. Seit den 2000ern ist es durch Gentechnik möglich, gezielt Gene eines Virus zu entfernen oder zu verändern, um die Reproduktion auszusetzen, wofür zuvor langwierige Zellkulturtechniken notwendig waren. Man kann so die Vermehrung des Virus kontrolliert stoppen und eine Rückkehr zur pathogenen (krankheitserregenden) Variante ausschließen [3]. Durch Genmodifikation können außerdem Strukturen wie das Gen für das Stachelprotein von SARS-CoV-2 in ein Virus, das für Menschen harmlos ist, eingeschleust werden.
Ein Beispiel für einen zugelassenen Vektorimpfstoff ist der Ebola-Impfstoff Ervebo [4]. Da es bisher keine Therapie gegen Ebola gibt, waren die Epidemien zwischen 2013 und 2020 verheerend für Zentralafrika. Die Sterblichkeitsraten lagen bei 25-90% [5]. Im August 2018 konnte mit Ervebo erstmals großflächig geimpft werden. Er erwies sich als hochwirksam [6] und wurde 2019 auch in der EU zugelassen [5]. Gegen Hepatitis B gibt es seit 1986 einen wirksamen genetisch veränderten Impfstoff [6].
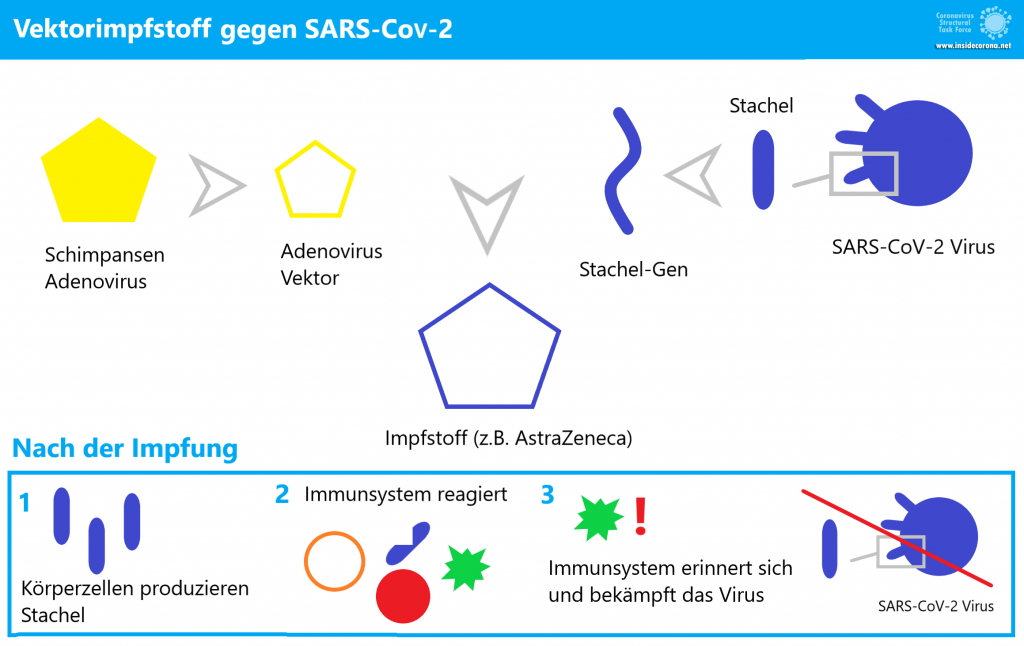
Wie funktioniert der AstraZeneca-Impfstoff?
Der Impfstoff heißt AZD1222 (auch bekannt als ChAdOx1 nCoV-19) [8]. Bei seinem Vektor handelt sich um ein nicht replizierendes Adenovirus ‒ einen Erkältungserreger ‒ das ursprünglich Schimpansen befällt. AZD1222 wurde so modifiziert, dass es das Gen des Coronavirus-Oberflächenproteins (Stachelprotein) [9] enthält. Man verwendet ein Schimpansenvirus, da die Immunsysteme der meisten Menschen an Adenoviren gewöhnt sind und diese daher ausgeschaltet werden, bevor sie Zellen befallen und dort die Bildung des Stachelproteins anregen können. Das Schimpansenvirus hingegen ist unbekannt für das menschliche Immunsystem und hat somit die Gelegenheit zu einer erfolgreichen Infektion [10]. Bei der Impfung kann es zu Nebenwirkungen kommen, die aber nichts mit dem Virus zu tun haben. Unwohlsein oder Krankheitssymptome werden durch unser Immunsystem ausgelöst, das mit allen Mitteln gegen einen Eindringling kämpft.
AZD1222 dringt genau wie ein herkömmliches Erkältungsvirus in einige menschliche Wirtszellen ein. Der Vektor transportiert dabei das Gen des Stachelproteins. Die DNA wird in unseren Zellen abgelesen und die enthaltenen Informationen zur Bildung des Stachelproteins herangezogen. Das Stachelprotein fungiert anschließend als Antigen, also eine Substanz, die eine Immunantwort auslöst. Das Immunsystem erkennt es als körperfremd und beginnt mit der Immunantwort: Es werden Antikörper produziert und die wenigen „infizierten“ Zellen mit dem AZD1222-Erbgut werden vernichtet. Antikörper binden wie Puzzleteile an das Antigen und fixieren es. Die Antigen-Antikörper-Klumpen werden anschließend abgebaut.
Was von dem ganzen Prozess zurückbleibt, sind Gedächtniszellen, die bei einem Angriff des echten SARS-CoV-2 das Stachelprotein wiedererkennen. Durch das Training mit dem Impfstoff verläuft die Immunreaktion dann schneller und effizienter. So wird eine Krankheit verhindert, bevor sie ausbrechen kann.
Wie wirksam und sicher ist der Impfstoff?
AZD1222 wurde in drei Phasen einer klinischen Studie schon an über 20.000 Probanden getestet [11]. Seit der Zulassung werden aber weiterhin Daten gesammelt und zu Real-World-Studien zusammengefasst. Durch diese kann man noch präzisere Aussagen über mögliche Nebenwirkungen machen, da in der „echten Welt“ Millionen Menschen geimpft und beobachtet werden.
So gab es bereits eine britische Studie zur Wirksamkeit von COVID-19-Impfstoffen – sowohl vektoriell als auch mRNA-basiert – bei Über-70-Jährigen. Das Ergebnis: Eine Einzeldosis schützt zu 60-75 % gegen einen symptomatischen Verlauf. Die Wahrscheinlichkeit für einen Krankenhausaufenthalt wird durch den AstraZeneca-Impfstoff außerdem um 80 % verringert, genau wie beim BioNTech-Impfstoff [12].
Die erste landesweite Studie fand in Schottland statt. Sie wurde von der Universität Edinburgh geleitet und sammelte Daten von 5,4 Millionen Geimpften. Aus den vorläufigen Daten geht hervor, dass der Impfstoff von AstraZeneca als Einzeldosis in 94 % der Fälle einen schweren Verlauf verhindert [13].
Durch die Real-World-Studien weiß man inzwischen, dass der Vektorimpfstoff umso wirksamer ist, je mehr Zeit zwischen erster und zweiter Dosis liegt. Deshalb empfehlen die offiziellen Stellen einen zeitlichen Abstand von 12 Wochen [14].
Was Nebenwirkungen angeht, hat man in Großbritannien durch den früheren Impfstart schon einige Daten über AZD1222 gesammelt [15].
Zu den häufigen Nebenwirkungen, die bei einem von zehn Geimpften auftreten, zählen:
- Empfindlichkeit, Schmerzen, Wärme, Juckreiz oder Blutergüsse an der Stelle, an der die Injektion verabreicht wurde,
- allgemeines Unwohlsein oder Müdigkeit,
- Schüttelfrost oder fiebriges Gefühl,
- Kopfschmerzen,
- Übelkeit, sowie
- Gelenkschmerzen oder Muskelkater.
Diese häufigen Nebenwirkungen sind mit denen des BioNTech-Impfstoffs vergleichbar. Weniger häufig treten Fieber oder grippeähnliche Symptome auf. Diese Art der Reaktion ist ein Zeichen, dass der Impfstoff das Immunsystem aktiviert und somit wirkt. In jedem Fall sind diese Symptome aber nicht mit den Risiken eines schweren COVID-19-Verlaufs vergleichbar. Schwere Nebenwirkungen wurden in den Studien nicht festgestellt, selbst mit geringer Wahrscheinlichkeit nicht.
Die EMA untersucht derzeit Fälle von Blutgerinnseln, die gleichzeitig mit einer Impfung von AstraZeneca aufgetreten sind. Bisher gibt es keine Hinweise darauf, dass der Impfstoff unsicher sei. Die Häufigkeit von Blutgerinnseln bei geimpften Personen ist nicht höher als allgemein in der Bevölkerung. Laut AstraZeneca [15.1] ist das Risiko einer Lungenembolie, einer tiefen Venenthrombose (TVT) oder einer Thrombozytopenie bei Geimpften nicht höher als in der Allgemeinbevölkerung. Unter den 17 Millionen geimpften Menschen in Europa ist es zu erwarten, dass bei einigen diese Krankheiten auftreten. Es ist sehr unwahrscheinlich, dass dies mit dem Impfstoff zusammenhängt.
Was ist im Impfstoff?
Neben dem modifizierten Adenovirus enthält der Impfstoff noch einige weitere Substanzen [16].
Es handelt sich um Zusatzstoffe, die den Impfstoff stabilisieren und das Verabreichen erleichtern. Dazu gehören: Aminosäuren, Stabilisatoren, Alkohol, Zucker, Salz, Bindemittel und Wasser. Außerdem ist der Impfstoff frei von Lebensmittelallergenen (wie Soja oder Lactose) und enthält keine menschlichen oder tierischen Bestandteile. Das klingt erstmal unglaublich bei einem Schimpansenvirus, der einen Wirt zur Vermehrung braucht. Wie kann das sein?
Alle Modifikationen und Vermehrungen des Virus fanden in einer menschlichen Zelllinie statt, die im Labor für solche Zwecke verwendet wird, in sogenannten HEK293-Zellen. Diese Zellen befinden sich jedoch nicht im Impfstoff.
Welche Vorteile bietet diese Art von Impfstoff?
Virale Vektorimpfstoffe können ‒ genau wie andere RNA/DNA-Impfstoffe ‒ sehr schnell entwickelt werden. Der Grund: Sobald die Gene des Erregers bekannt sind, können diese zur Impfstoff-Entwicklung verwendet werden. Somit verkürzt sich die Zeit bis zum Start klinischer Studien. Das macht Vektorimpfstoffe geeignet für plötzliche Ausbrüche von Epidemien [17].
Ein Vorteil des AstraZeneca-Impfstoffs ist seine Dosierung. Oxford-AstraZeneca haben für ihren Vektorimpfstoff zunächst verschiedene Dosierungsansätze getestet. Verglichen wurden Impfungen mit zwei Standard-Dosen in einem Abstand von vier bis zwölf Wochen und eine Impfung mit nur einer Dosis. Eine Standarddosis enthält hierbei 5x1010 Viruspartikel [18].
„Durch die Anwendung eines effektiveren Dosierungsschemas“, so Professor Pollard, der leitende Wissenschaftler in Oxford, „könnten mehr Menschen durch die gleiche Menge Impfstoff versorgt werden“ [19]. Als ideales Schema stellte sich die Verabreichung einer halben Dosis (2,2x1010 Viruspartikel) heraus, gefolgt von einer Standard-Dosis im Abstand von mindestens einem Monat. Die Wirksamkeit betrug hier 90 % [20]. DerBefund muss aber zunächst noch mit zusätzlichen Daten gesichert werden, sodass fürs Erste weiterhin mit zwei Standarddosen geimpft wird.
Fazit
Die Wissenschaft kann sich heutzutage die besonderen Fähigkeiten von Viren zunutze machen. Viren haben es in der Natur perfektioniert, ihr Erbgut in Wirte einzuschleusen. Daher werden sie eingesetzt, um genveränderte Pflanzen oder Zellen zu erzeugen, Erbkrankheiten zu behandeln oder Impfstoffe herzustellen.
Vektorimpfstoffe ‒ wie der von AstraZeneca ‒ enthalten harmlose und nicht replizierende Viren, die ein Merkmal eines krankmachenden Virus enthalten. Durch das künstliche „Infizieren“ mit dem Vektorimpfstoff wird das Immunsystem auf den Krankheitserreger trainiert und reagiert bei einer echten Infektion schneller und effektiver.
[1] Jackson D.A., Symons R.H., Berg P. Biochemical method for inserting new genetic information into DNA of Simian Virus 40: Circular SV40 DNA molecules containing lambda phage genes and the galactose operon of Escherichia coli. Proc. Natl. Acad. Sci. USA. 1972;69:2904–2909. doi: 10.1073/pnas.69.10.2904.
[2] Zabner, Joseph et al. 1993 Adenovirus-mediated gene transfer transiently corrects the chloride transport defect in nasal epithelia of patients with cystic fibrosis Cell, Volume 75, Issue 2, 207 – 216.
[3] Using directed attenuation to enhance vaccine immunity, Rustom Antia, Hasan Ahmed, James J Bull, bioRxiv 2020.03.22.002188; doi: https://doi.org/10.1101/2020.03.22.002188
[4] https://www.who.int/vaccine_safety/committee/topics/ebola/Jul_2019/en/
[5] https://www.ema.europa.eu/en/news/first-vaccine-protect-against-ebola
[6] https://www.thelancet.com/journals/lancet/article/PIIS0140-6736(16)32621-6/fulltext#seccestitle160
[7] https://www.nature.com/articles/d42859-020-00016-5
[8] https://www.ema.europa.eu/en/documents/product-information/covid-19-vaccine-astrazeneca-product-information-approved-chmp-29-january-2021-pending-endorsement_en.pdf
[9] https://jvi.asm.org/content/jvi/77/16/8801.full.pdf
[10] https://www.cdc.gov/vaccines/covid-19/hcp/viral-vector-vaccine-basics.html
[11] https://clinicaltrials.gov/ct2/show/NCT04516746
[12] https://www.medrxiv.org/content/10.1101/2021.03.01.21252652v1
[13] https://www.ed.ac.uk/files/atoms/files/scotland_firstvaccinedata_preprint.pdf
[14] https://www.rki.de/DE/Content/Kommissionen/STIKO/Empfehlungen/AstraZeneca-Impfstoff.html
[15] https://www.gov.uk/government/publications/regulatory-approval-of-pfizer-biontech-vaccine-for-covid-19/information-for-uk-recipients-on-pfizerbiontech-covid-19-vaccine#side-effects
[15.1] https://www.astrazeneca.com/media-centre/press-releases/2021/update-on-the-safety-of-covid-19-vaccine-astrazeneca.html
[16] https://www.astrazeneca.at/content/dam/az-at/pdf/2021/Vaccine%20guide%20for%20HCPs%20-%202021-02-02.pdf
[17] https://www.frontiersin.org/articles/10.3389/fimmu.2018.01963/full
[18] https://papers.ssrn.com/sol3/papers.cfm?abstract_id=3777268
[19] https://www.astrazeneca.com/media-centre/press-releases/2020/azd1222hlr.html
[20] https://www.thelancet.com/journals/lancet/article/PIIS0140-6736(20)32661-1/fulltext
