SARS-CoV-2: Nicht neu, aber anders
Das neuartige Coronavirus (SARS-CoV-2 oder 2019-nCoV) ist ein großes Einzelstrang-RNA Virus mit positiver Polarität. Es ist der Familie der Betacoronaviren zugeordnet und zeigt große genetische Ähnlichkeit zu SARS-CoV und MERS-CoV und ist besonders nah verwandt mit dem Bat-SARS-like-CoV (Fledermaus-Coronavirus), von dem es wahrscheinlich auch abstammt. Trotz vieler Gemeinsamkeiten erkennt man, wenn man den Infektionsmechanismus und die Struktur seiner Proteine genau analysiert, eindeutige Unterschiede zu SARS-CoV und anderen Coronaviren.
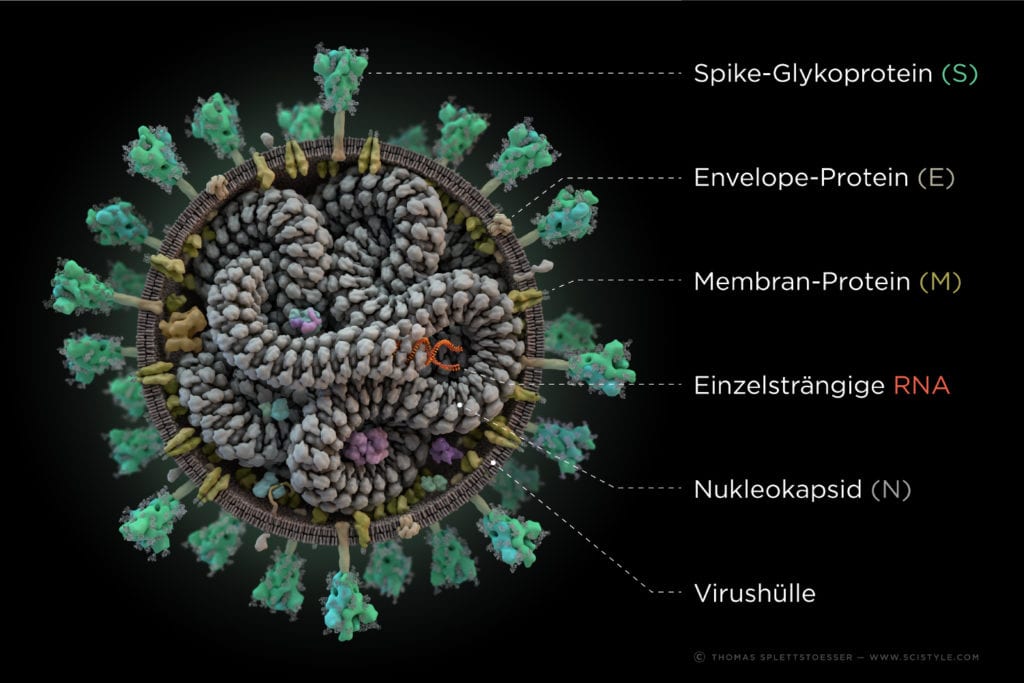
Ein wertvoller Inhalt
Wie fast alle RNA-Viren besitzt das Virus eine Lipid-Hülle, in die diverse Proteine integriert sind. Diese Hülle ist zuständig für die Interaktion mit der Wirtszelle und dient dem Schutz des Erbgutes, der viralen RNA. Diese RNA dient unter Anderem als direkte Vorlage für die Herstellung der beiden Polyproteine pp1a und pp1ab, die für 16 nicht-strukturelle-Proteine (nsp) kodieren. Diese 16 nsps, kodiert von etwa zwei Dritteln der gesamten Genom-Länge, werden von der Chymotrypsin-artigen-Protease (=Hauptprotease) und ein oder zwei Papain-artigen-Proteasen aus der langen Polypeptidkette herausgeschnitten und so in funktionelle Proteine umgewandelt. Infolgedessen kann sich der Replikations-Transkriptions-Komplex (RTC) bilden und eine Vielzahl sogenannter subgenomischer RNAs (sgRNAs) bilden, die wiederum als Grundlage für die Produktion subgenomischer mRNA und neuer viraler Proteine dienen. Andere Bereiche des Genoms kodieren für mindestens vier Strukturproteine, die Teil der Virushülle sind und als Nucleokapsid die RNA stabilisieren (bezeichnet als S-, M-, E- und N-Protein für Spike, Membran, Hülle und Nukleokapsid)
Der erste Kontakt
Das Virus befällt hauptsächlich Zellen, die den Oberflächenrezeptor ACE2 (Angiotensin-konvertierendes Enzym 2) tragen. Dieser Zelltyp findet sich häufig im Gewebe der Atemwege und bei Kontakt der Viren mit der Zelle und ihren Oberflächenproteinen kann die virale RNA eingeschleust werden. Aber wie geschieht dieses Einschleusen? Das Spike-Protein, welches die "Korona" um das Virus bildet, tritt mit einem dafür spezialisierten Teil des Proteins, der Rezeptor-bindenden-Domäne in Kontakt mit der Wirtszelle. Anschließend können die Viren über den komplexen Prozess der Endozytose mit der Zelle verschmelzen und ihre Erbinformation so einschleusen. Sobald eine Zelle infiziert ist, fungiert sie als kleine Fabrik zur Herstellung hunderter neuer Virus-Moleküle, was eine starke Reaktion des Immunsystems hervorruft. Die bekanntesten Symptome umfassen Husten, Fieber, Atemnot, Müdigkeit, Geschmacksverlust, Kopfschmerzen, Diarrhö, Lymphopenie, oder Lungenentzündung, die in schweren Fällen sogar den Tod des Patienten verursachen können.

PDB: 3SCL, Bild von Ferdinand Kirsten
Die Struktur und der Infektionsmechanismus des Virus bieten viele Möglichkeiten, um spezifische Medikamente oder Impfungen zu entwickeln. Eine Hemmung der Hauptprotease, die Störung der Virus-Zusammensetzung, des Spike-Proteins oder des RTCs sind nur einige von zahlreichen möglichen Angriffspunkten der Wissenschaft, um antivirale Methoden zu entwickeln und eine Ausbreitung zu verlangsamen oder zu stoppen. Bis zum jetzigen Zeitpunkt konnte jedoch noch kein Wirkstoff entwickelt werden, der in klinischen Studien zum gewünschten Ergebnis führt.
Zum Nachlesen (auf Englisch):
- https://onlinelibrary.wiley.com/doi/pdf/10.1002/jmv.25681
- https://www.sciencedirect.com/science/article/pii/S0896841120300469
- https://www.nature.com/articles/pr198040
- https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1829448/
- https://onlinelibrary.wiley.com/doi/full/10.1002/jmv.25685
- https://en.wikipedia.org/wiki/Angiotensin-converting_enzyme_2
