Am aussagekräftigsten ist ein Modell immer dann, wenn es der Wahrheit so nahe wie irgend möglich kommt. Genau das wollen wir mit dem 3D-Druck-Modell von SARS-CoV-2 erreichen. Allerdings gibt es in der Literatur zu dem Virus sehr viele, teils verwirrende und widersprüchliche Beschreibungen.
Diese Literatur durch zuarbeiten und auszuwerten, auf der Suche nach vollständigem Verständnis und einem klaren Bild, ist beinahe so etwas wie eine Lebensaufgabe. Und natürlich – jedes Mal, wenn neue wissenschaftliche Studien und Beobachtungen veröffentlicht werden, fängt man ein Stück weit wieder von vorne an, weil Anpassungen nötig werden oder feste Vorstellungen in Frage gestellt werden müssen.
Zu Beginn der Pandemie, als es noch keine oder nur wenige Daten des neuartigen Virus gab, hat man die Annahmen über das Aussehen von SARS-CoV-2 hauptsächlich aus dem Wissen über andere Coronaviren getroffen. Insbesondere SARS-CoV-1, das die vorherige SARS-Pandemie ausgelöst hatte, und eng verwandt mit SARS-CoV-2 ist, wurde als Grundlage für Annahmen herangezogen. [1]
Auf der Grundlage dieser ersten Annahmen und Bilder hat die Coronavirus Structural Task Force ein erstes 3D-Druck-Modell entworfen.
Doch seither sind viele neue Bilddaten und Aufnahmen von SARS-CoV-2 veröffentlicht worden und diese Bilder zeigen, dass viele Dinge eben doch anders sind, als zuvor angenommen.
Wir haben die Literatur durchgearbeitet und analysiert, dabei sehr viel gelernt und dieses Gelernte jetzt verwendet, um ein aktuelleres Modell zu entwerfen.
Das Erscheinungsbild des Virus
Viren sind winzig klein. So klein, dass es sogar mit modernen bildgebenden Verfahren sehr schwierig ist, sie direkt zu beobachten. (vgl hier )
Früher hat man, um Viren zu untersuchen, die Proben mit Metall beschichtet und dann ein elektronenmikroskopisches Bild (vgl. Abbildung) aufgenommen. Auf diesem Bild konnte man die Oberfläche deutlich sehen.
Aus diesem Bildgebungsverfahren stammt übrigens auch der Name „Coronavirus“. Auf den so aufgenommenen Bildern sahen die Viruspartikel ein bisschen aus, wie kleine Sonnen, die umgeben waren von einer Korona aus Lichtstrahlen.
Die erfolgversprechendste Methode, um etwas über das Aussehen von Viren herauszufinden, ist es übrigens nicht, sich nur ein einzelnes Virion anzusehen, sondern im Gegenteil. Man mittelt hunderte davon und erhält so eine Vorstellung davon, wie der Durchschnittswert aussieht.
In der Realität und ganz besonders bei Coronaviren gilt: jedes einzelne Viruspartikel sieht anders aus als die anderen. Zum Beispiel sind einige größer und andere kleiner und nur wenn keine äußeren Kräfte wirken, oder innere Strukturen stören, ist das Virus wirklich rund.
Es braucht keine große Krafteinwirkung, damit die „wobbelige“ und dünne Doppelmembranhülle von SARS-CoV-2 sich verformt.
Es wird beispielsweise vermutet, dass das Coronavirus-Partikel eigentlich in einer nahezu perfekten Kugelform entsteht, sich aber dann, wenn es einer leicht sauren Umgebung ausgesetzt wird, etwas deformiert und dass diese Veränderung wichtig für die Infektiosität sein könnte. Außerdem wird vermutet, dass die Konformation der M-Proteine die Membrankrümmung und somit die äußere Form des Virus beeinflusst.
Unser 3-D-Modell ist von daher nicht exakt rund, sondern eher geformt wie eine Kartoffel. Allerdings behaupten wir ausdrücklich nicht, dass das „die Form“ des Virus ist – es ist nur eine von vielen möglichen.
Beim Sichten und Vergleichen der neuen Daten zu SARS-CoV-2 ist uns eine weitere Neuerung sofort aufgefallen. Im Vergleich zu anderen Coronaviren, die vor der Pandemie untersucht wurden, scheint SARS-CoV-2 kleiner zu sein.
Um diese neue Erkenntnis widerzuspiegeln, haben wir den Durchmesser unseres Modells um 12% von 100 mm auf 88 mm reduziert. (Das Modell ist so skaliert, dass 1 mm 1 nm entspricht.)
Wie bei der Kartoffel-Form ist es so, dass einzelne Viren eine Vielzahl von unterschiedlichen Größen haben können und 88mm ist nur ein Durchschnittswert, der mit gemessenen Daten übereinstimmt.
Durch die Verkleinerung des Virions ergibt sich eine ca. 23% kleinere Oberfläche im Vergleich zum alten Modell. Messdaten legen nahe, dass die Verteilung der M-Proteine auf der Virionoberfläche sich in etwa konstant ist, weshalb wir die Anzahl der M-Proteine entsprechend um 23% verringert haben.
Für die E-Proteine zeigen die Daten allerdings, dass Anzahl in jedem Virion in etwa gleich ist, deshalb haben wir die Anzahl bei 20 belassen.
Im verwendetem Maßstab wäre die im Virion enthaltene RNA übrigens etwa 10m lang und 1mm dick.
Eines unserer Modelle haben wir mit einem Loch im Boden designt, damit man es flach aufstellen und zum Beispiel als Briefbeschwerer verwenden kann. Natürlich kann man aber auch 10m Schnur darin verstecken. Zu Demonstrationszwecken kann man diese dann herausziehen und veranschaulichen, wie erstaunlich lang das Molekül der RNA von SARS-CoV-2 ist.
Die Stacheln – nachgezählt
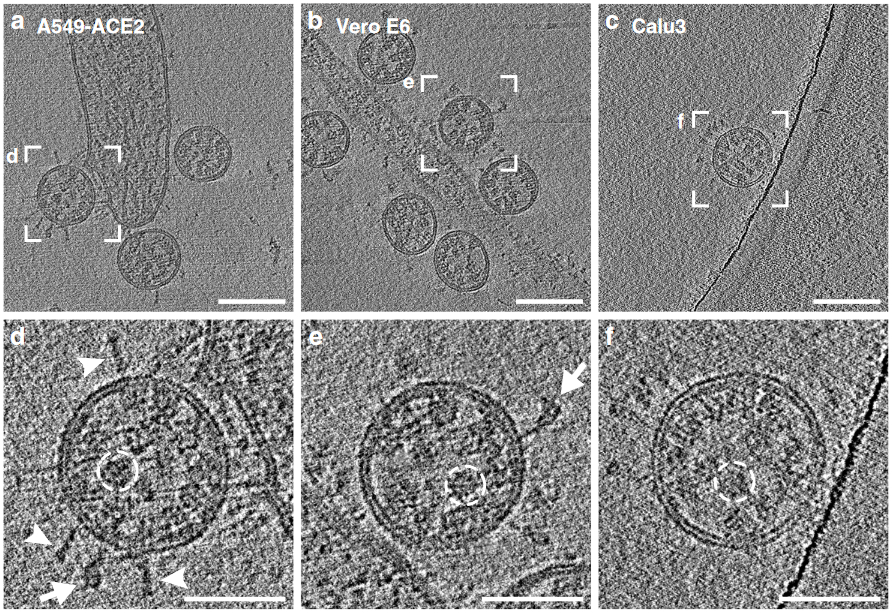
Die experimentellen Daten, die aus tatsächlichen Untersuchungen von SARS-CoV-2 stammen, zeigen, wie oben schon erwähnt, dass das Virion kleiner ist als angenommen und die Anzahl der Spikeproteine ist weitaus geringer als ältere Studien das angenommen hatten.
Die aktuelle Literatur legt nahe, dass die tatsächliche Anzahl der Spikeproteine auf der Virusoberfläche durchschnittlich zwischen 26 und 48 liegt.
Unser erstes Modell von SARS-CoV-2, dessen Daten vor allem auf den Bildern anderen Coronaviren basieren, hatte etwa 100 Spikes. Bei unserem neuen Modell haben wir diese Anzahl auf 26 reduziert.
Während es durchaus im Bereich des Möglichen ist, dass SARS-CoV-2 einfach weniger Stacheln hat als andere Coronaviren ist es genauso möglich, dass dieser Unterschied darauf zurückzuführen ist, dass die heutigen Mikroskope und bildgebenden Verfahren viel leistungsfähiger sind als sie es noch vor 10 Jahren waren.
Obwohl das Coronavirus unvorstellbar klein ist, war es in der Vergangenheit doch so, dass ein einzelnes Virion zu groß war, um alle Teile davon gleichzeitig in einem Bild „scharf“ abzulichten.
Nachdem man somit aber die Stacheln in einer begrenzten Region des Virions sehe konnte, wurden sehr ausgefeilte Methoden verwendet, um die Gesamtanzahl der Stacheln abzuschätzen.
Eine dieser Methoden ist das Tammes-Problem: Ein mathematisches Rätsel, bei dem es darum geht, die maximale Anzahl nicht überlappender, gleich großer Kreise auf der Oberfläche einer Kugel zu verteilen.
Sogar aus der 2D-Bildgebung kann man den minimalen Abstand zweier Spikeproteine auf der Virusoberfläche leicht abschätzen und wenn man davon ausgeht, dass die gesamte Oberfläche des Virions möglichst dich mit Spikes bedeckt ist, kommt man mit der Tammes-Methode auf etwa 50 Spikes pro Virion

Viele Bilder von SARS-CoV-2 zeigen aber Lücken zwischen den Stacheln und es ist sehr wahrscheinlich, dass sehr viel weniger sind als angenommen.
Mit neuen Techniken kann man mittlerweile ein scharfes Bild eines kompletten Virions erzeugen, so dass man die Stachelproteine einfach zählen kann. Diese neuartigen Bilder zeigen durchschnittlich 26 Stacheln pro Virion und somit ist das die vernünftigste verfügbare Zahl, bis vielleicht anderweitige Beweise aus Studien auftauchen. 26 ist nur ein Durchschnittswert – die exakte Anzahl von Spikeproteinen auf einem Virion ist variabel.
Die selbe Studie, aus de die oben genannten Daten stammen, spricht auch davon, dass die Stacheln sich auf der Virusoberfläche frei drehen können und nicht ganz aufrecht stehen, sondern im Durchschnitt um 40° geneigt sind. Dieser Neigungswinkel ist von Stachel zu Stachel unterschiedlich.
Alle diese neuen Erkenntnisse haben wir in der aktuellen Version unseres Modells berücksichtigt. Es hat 26 Spikeproteine, deren Stiele in Winkeln von 30°, 40° und 50° gebogen sind. Um die Flexibilität und die Robustheit des Modells zu verbessern, haben wir auch eine Version entworfen, bei der die Spitzen der Spikeproteine mit Spiralfedern mit dem Viruskörper verbunden sind. Allem Enthusiasmus zum Trotz kann man aber natürlich nicht alles mit einem 3D-Modell darstellen. So zum Beispiel, dass die Spikeproteine (ebenso wie die M- und E-Proteine) in der Bilipidmembran, die die äußere Hülle des Virions dient, „schwimmen“. Wir haben keine Möglichkeit gefunden, das darzustellen.

Also – wo ist denn jetzt das Modell?
Wir haben die aktualisierten Daten für das 3D-Druck-Modell des Virus bei thingiverse hochgeladen.
In unserm Blog finden Sie eine detaillierte Anleitung um das neue Modell zu drucken und zusammenzubauen.
Und hier ist noch eine 3D-Vorschau des neuen Modells auf Sketchfab:
Verweise
[1] "Coronavirus never before seen in humans is the cause of SARS". United Nations World Health Organization. 2003-04-16. Archived from the original on 2004-08-12. Retrieved 2022-02-17.
[2] Neuman, B., Kiss, G., Kunding, A. H. et al. A structural analysis of M protein in coronavirus assembly and morphology. J. Struct. Biol. 174, 1 (2011), 11–22. https://dx.doi.org/10.1016%2Fj.jsb.2010.11.021.
[3] Yao, H., Song, Y., Chen, Y. et al. Molecular Architecture of the SARS-CoV-2 Virus. Cell 183, 3 (2020), 730-738. https://doi.org/10.1016/j.cell.2020.09.018.
[4] Klein, S., Cortese, M., Winter, S.L. et al. SARS-CoV-2 structure and replication characterized by in situ cryo-electron tomography. Nat Commun 11, 5885 (2020). https://doi.org/10.1038/s41467-020-19619-7.
